转录是基因表达的关键步骤,也是真核生物生长发育和生命活动中必不可少的过程。该过程是以双链DNA中的一条链为模板,以A、U、C、G四种核糖核苷酸为原料,在RNA聚合酶的催化作用下转录形成mRNA。转录过程又可以被分为转录起始、延伸、终止3个阶段,首先RNA聚合酶与基因上的启动子[1]结合诱导转录的开始,在调控因子的协助下进一步完成延伸,直至遇到终止信号完成特定基因的转录。所以,转录是将遗传信息从DNA流向RNA的过程,这对生物的多样性及生命的存在均有重要的意义。
前期的研究展示,真核生物的细胞核内的转录主要由3种RNA聚合酶介导,分别是RNA聚合酶I,RNA聚合酶II,以及RNA聚合酶III。其中RNA聚合酶I主要定位在细胞核仁,负责催化合成核糖体RNA(rRNA)中的18S rRNA、28S rRNA和5.8S rRNA;RNA聚合酶III则主要定位于细胞核基质,负责指导转运RNA(tRNA)、5S rRNA以及一些非编码RNA的合成;RNA聚合酶II也定位在细胞核基质中,负责催化核糖核酸的转录,来合成信使RNA(mRNA)及大多数snRNA、hnRNA。近期对植物体的研究发现,植物细胞核内除了上述3种RNA聚合酶外,还包含两种特异性的RNA聚合酶,即RNA聚合酶 IV和RNA聚合酶 V。RNA聚合酶IV和RNA聚合酶V能够促进非编码RNA的产生,并且这些非编码RNA通过DNA甲基化(RdDM)途径[2]对基因沉默发挥重要的调控作用。
RNAP II在转录过程中发挥的作用最大,所以关于RNAP II转录调控的研究较多,这对于揭示真核生物生命活动的分子基础具有重要的生物学意义[3]。RNAP II在进行高水平的转录调控过程中,通常需要一组转录因子(general transcription factors,GTFs),包括TFIIA、TFIIB、TFIID、TFIIE、TFIIF和TFIIH来协调进行[4]。在过去的几十年里,人们针对这些转录因子的主要功能做了大量的研究,发现这些转录因子都是以一种动态结合和解离的方式来调控转录的发生[5]。那么RNAP II在转录过程中的动态变化又是怎样呢?随着单分子成像和标记技术的不断发展和应用,活细胞内单个RNAP II分子的动态研究得以实现并不断的推进。在活体胚胎干细胞内,Cho等[6]观察到RNAP II可以形成稳定存在的、或者短暂存在的clusters团簇结构。同时,孙育杰课题组[7]也在哺乳动物的活细胞核内,首次原位观察到了RNAP II clusters的动态组装和解聚过程。然而植物中关于RNAP II的活体动态研究比较少,因此本文主要对真核生物RNAP II结构和功能的研究进行综述,并重点总结动物活细胞内RNAP II的动态标记和成像技术的研究进展,以期为植物活细胞内观察RNAP II动态转录研究提供参考。
1 RNA聚合酶II的结构
Rpb1是RNAP II 的最大亚基,由于其具有催化活性,因此成为RNAP II 发挥转录功能的主要执行者,它的羧基末端有一段特殊的结构域CTD(carboxyl-terminal domain),在执行转录过程中发挥关键调控作用[10]。CTD具有高度保守的一致序列(Tyr-Ser-Pro-Thr-Ser-Pro-Ser),是一段基本上无结构的多肽链,并且在不同物种中的重复次数各不相同,如酵母中为26个重复,动物和人类中为52个重复,而植物中则有39个重复[11]。CTD的这种重复特性,能够增加与之结合的蛋白数目,这对其功能的发挥产生直接的影响[12]。例如,在mRNA加工修饰的过程中,CTD结构通过与加帽酶、剪接因子以及诸多聚尾因子的结合,从而直接参与调控这些复杂的加工反应[13]。另外,CTD也会发生一系列修饰作用,譬如当外界病原菌入侵时,拟南芥RNAP II的CTD上3个氨基酸残基发生快速而短暂的磷酸化,为植物激活初级免疫应答提供了一种快速途径[14]。
2 RNA聚合酶II的功能
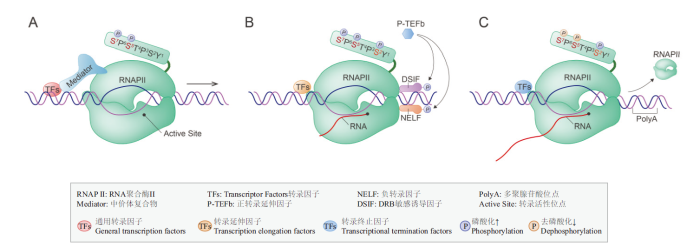
图1
图1
RNAP II介导的转录过程
A:转录起始;B:转录延伸;C:转录终止
Fig. 1
RNAP II mediated transcription
A:Transcription initiation. B:Transcription elongation. C:Transcription termination
2.1 RNA聚合酶II与转录调控起始
转录起始是一个动态协调的过程,也是转录发生过程中最关键的一个步骤[18]。在转录因子和中介体复合物(mediator complex)[19]的协助下,未经任何修饰的RNAP II 被招募到启动子附近,形成转录预起始复合物(pre-initiation-complex,PIC)[20]。在ATP的作用下,DNA双链被打开,其中一条DNA模板链进入RNAP II的活性位点,形成开放复合物来激活转录的起始。紧接着在RNAP II酶的催化作用下,核苷三磷酸(NTPs)作为底物开始合成mRNA的前体,标志着转录起始的正常进行[21]。通过单分子荧光成像技术观测RNAP II—PIC的组装过程发现,参与组装的转录因子是通过一种短暂的、动态的方式来参与转录的起始[22]。因此,转录起始是由RNAP II和多种转录因子共同参与的动态调控过程。
2.2 RNA聚合酶II与转录延伸
RNAP II离开启动子并开始合成RNA时,即进入了转录延伸阶段[26]。在早期延伸过程中,RNAP II在启动子的转录起始位点(transcription start site,TSS)下游20-100 bp处会发生转录暂停。转录暂停现象既是转录过程中的限速步骤,也是发生在早期延伸和生产性延伸之间的过渡阶段。当负转录延伸因子(negative elongation factor,NELF)和DRB敏感诱导因子(DRB-sensitivity-inducing factor,DSIF)与RNAP II结合时,会引起RNAP II 的转录暂停;而当正转录延伸因子B(positive transcription elongation factor b,P-TEFb)[27]磷酸化NELF和DSIF时,负转录延伸因子转化为正转录延伸因子,就会触发RNAP II 转录的再次启动[28]。
转录延伸过程中RNAP II的CTD氨基酸残基Ser2可以被P-TEFb磷酸化,来招募其他转录延伸因子和染色质修饰因子,并且CTD的另外两个氨基酸残基Ser5和Ser7在磷酸酶的作用下会逐渐脱磷酸化。总之,在RNAP II的CTD氨基酸残基Ser2的磷酸化状态和Ser5、Ser7的去磷酸化状态的共同作用下,转录的延伸过程进入生产性延伸阶段[29]。因此,RNAP II的CTD磷酸化状态明显影响转录延伸的进程,但反过来转录延伸的过程是否也会影响CTD的磷酸化修饰,以及CTD状态的转变为什么发生在转录的特定延伸阶段,这些问题仍需进一步的研究来解释。
2.3 RNA聚合酶II与转录终止
当转录延伸遇到终止子时,RNAP II在解旋酶的作用下从DNA模板上解离,发生转录终止[30]。RNAP II介导的转录终止是一个极其复杂的过程,大多数mRNA转录终止依赖于多聚腺苷酸(Poly A)位点,并且受到RNAP II的CTD磷酸化状态的调控[31]。在转录终止时,RNAP II的CTD氨基酸残基Ser2的磷酸化水平达到最高,而另一个氨基酸残基Ser5的磷酸化水平逐渐降低。因此,RNAP II的CTD磷酸化状态的转变,会促使Poly A位点上招募到新的转录因子,导致RNAP II转录复合物的构象发生变化[32],而从RNA-DNA杂合链上解离下来,至此一次完整的转录过程发生终止。另外在转录终止时,RNAP II在磷酸酶的作用下发生去磷酸化[33],产生非磷酸化修饰的RNAP II,从而可以参与到下一轮的转录循环反应中。
3 RNA聚合酶II标记及成像技术
RNAP II的结构和功能已经被广泛研究,然而关于活细胞内是如何调控转录过程,以及它的动态特征是怎样的,这些问题目前仍不清晰。近年来,随着活体荧光标记技术和超分辨率成像技术的发展,研究人员可以实时的追踪RNAP II的动态过程,能够实现对RNAP II的高时空动态成像与观察[34]。下面对这些标记和成像技术进行介绍。
3.1 RNA聚合酶II标记技术
荧光标记技术是指将不同的荧光探针特异性地连接到目标分子上的一种技术,常用的荧光探针包括:有机染料、荧光纳米晶体和荧光蛋白等[35]。其中荧光蛋白指的是带有荧光基团结构的蛋白质,它通过对目标分子进行特异性地标记[36],是目前荧光标记中最常用的手段。绿色荧光蛋白(green fluorescent protein,GFP)[37]及其他颜色的荧光蛋白[38]由于稳定性强和亮度高的优点,受到了研究人员的青睐并实现了广泛的应用。但新型超高分辨率技术的发展,对荧光蛋白的选择产生了更高的需求,亟需更亮、更稳定的荧光蛋白作为标签。光激活(photoactivatable fluorescent proteins,PAFPs)、光转化(photoconvertible fluorescent proteins,PCFPs)、光开关(photoswitchable fluorescent proteins,PSFPs)荧光蛋白等的出现,不仅提高了显微观察的精准性,也极大地推动了活细胞成像技术的发展[39]。
之前对转录调控的研究中,研究者通常选择荧光原位杂交技术(fluorescence in situ hybridization,FISH)[40]来进行,它能够同时识别特定的基因和转录位点并进行标记。但是这一技术需要将细胞固定,而固定后的“死细胞”获得的结果与活细胞中的结果有很大的不同,固定后的“死细胞”不能反映植物细胞感受外界信号后的真实转录情况,因而无法获取动态变化的准确信息。在这个过程中,基因编辑技术如CRISPR/Cas9(clustered regularly interspaced short palindromic repeats-CRISPR-associated protein 9)和自我标记蛋白(self-labeling proteins,SLPs)技术是目前非常有用的技术手段。
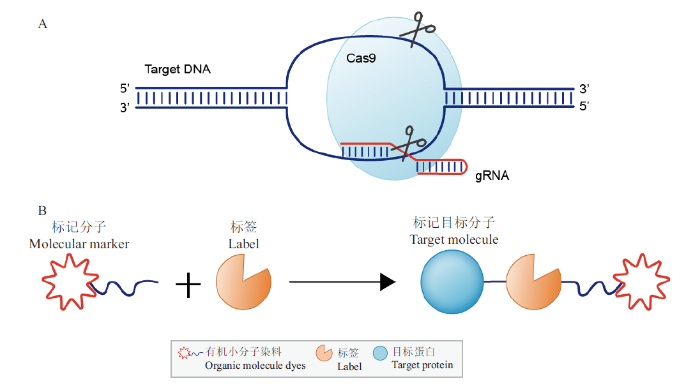
CRISPR/Cas9是一种基因靶向编辑技术,它可以将编码荧光蛋白的基因序列插入到基因组的特定位置,来实现基因转录的可视化[41]。CRISPR/Cas9技术由Cas9核酸酶和单链引导RNA(single guide RNA,sgRNA)两部分组成,Cas9核酸酶在sgRNA的指导下,可以靶向切割目的基因,再利用同源DNA修复技术完成对该目的基因的编辑和重组。研究者通过CRISPR/Cas9技术,利用光转换荧光蛋白(Dendra2)对小鼠胚胎细胞的内源性RNAP II进行特异性标记,观察到活细胞核内RNAP II团簇的存在,并且其寿命非常的短暂(约为8 s)[42],这一结果与之前标记外源性RNAP II的观察结果相一致。因此,CRISPR/Cas9是一种强大且精细的工具,通过基因工程技术将荧光蛋白敲进内源基因以使融合蛋白在生理相关水平上表达。同时由于该技术具有易于设计、载体构建简单、实施快捷和成本低的优点,在生物学领域里的多个物种中[43]都得到了广泛的应用,但CRISPR/Cas9方法在应用的过程中仍然存在脱靶[44]的问题,还需不断改进。
SLPs技术(如Halo-Tag和SNAP-Tag)[45]通过一种共价结合的方式让目标蛋白携带上荧光标签,共价键具有不可逆、稳定性强、特异性高等特点,是研究活细胞蛋白追踪的首选技术。常用的一些标签包括 HaloTag、SnapTag、CLIPtag和TMPTag,都可以通过遗传编码的方法与目标蛋白进行融合表达,并且这些标签可以催化有机染料标记的小分子底物共价连接到目标蛋白[46]。在小分子透膜染料中,janelia fluor(JF)系列的染料表现最佳[47],如JF549、JF646、PA-JF549及PA-JF646等,在近期的单分子成像领域被广泛应用。例如在一项最近的研究中,Cho[6]等分别用JF646-Halotag和Dendra2来标记转录因子Mediator和RNAP II,双色光片显微成像显示活体细胞核内有大量聚集的双色亮点,并且通过量化分析发现,Mediator和RNAP II团簇共定位比例达到90%。另外,在植物研究应用方面,利用一种新开发的Halo-Tag可编辑技术[48],来探索转录因子和蛋白之间的相互作用,这使得许多新的转录因子被识别和研究(图2)。
图2
图2
CRISPR/Cas9和SLPs技术标记目标分子的示意图
A:CRISPR/Cas9技术标记目标分子的示意图;B:SLPs技术标记目标分子的示意图
Fig. 2
The schematic of CRISPR/Cas9 and SLPs
A: The schematic of CRISPR/Cas9. B: The schematic of SLPs
3.2 RNA聚合酶II成像技术
表1 不同显微技术的主要优缺点
Table 1
| 参数Parameter | PALM | STORM | STED | LSFM | Bayesian nanoscopy |
|---|---|---|---|---|---|
| 分辨率Resolution/nm | <30 | <30 | 20-70 | 亚细胞分辨率 Subcellular resolution | 50 nm |
| 成像深度Imaging depth/μm | <10 | <10 | <100 | 通常<50 | 通常<50 |
| 成像速度Imaging speed | 慢Slow | 慢-低Slow to low | 中等Medium | 非常快Very fast | 非常快Very fast |
| 内源性Endogenous | 无No | 有Yes | 有Yes | 有Yes | 有Yes |
| 光毒性Phototoxicity | 中到高Medium to high | 中等Medium | 高High | 低Low | 低 Low |
| 蛇币价格Equipment price | 中等Medium | 中等Medium | 非常昂贵Very expensive | 非常昂贵Very expensive | 非常昂贵Very expensive |
下面对这几种技术在RNAP II动态研究中的应用进行介绍。
单分子定位的显微技术,如光激活定位(photo activated localization microscopy,PALM)和随机光学重构(stochastic optical reconstruction microscopy,STORM)显微技术,能够实现30 nm的空间分辨率和10 μm的成像深度,设备价格也中等,并且它们成像原理基本相同,都是利用荧光分子的特性和单分子定位的精准度,通过获取多张图片进而重构出一幅超分辨图像;区别在于STORM技术是利用荧光探针等技术来标记目标蛋白[52],而PALM技术则是利用融合表达的荧光蛋白来进行标记的,因此PALM技术更适合于活细胞内的蛋白成像。在活体胚胎细胞核内,研究者就利用PALM超分辨成像技术揭示了RNAP II的动态特征,表明内源性的RNAP II是以瞬时的团簇形式存在的[42]。同样通过PALM技术,Cisse等[34]观察到了RNAP II团簇的组装过程,但对于RNAP II团簇是否发生解聚过程仍不清楚。但无论是STORM还是PALM技术,都是利用“时间换空间”的方法来提高成像分辨率,这也就限制了它们的时间分辨率,不适合应用于分子的长期动态观察。
受激发射损耗技术(stimulated emission depletion,STED)及其它衍生方法,它作为共聚焦显微装置的附加模式,通常是比较容易操作的一项技术。STED的原理是利用两束光源,其中激发光照射目标区域发出荧光,而损耗光则会将目标样品以外的荧光淬灭,从而达到有效荧光发光面积的最小化[53],所以STED技术将光学显微镜的分辨率提高了近10倍。另外,Wildanger等[54]通过引入3D STED,又实现了3个方向上超分辨率的提升。在最近的一项研究中,Li等[55]利用3D STED技术来实时成像、追踪和定量分析活细胞内的RNAP II分子,观察其在转录周期中的动态变化,从而为揭示RNAP II的动力学与转录调控之间的联系奠定基础。但是,STED技术也有很多缺陷,如STED技术不适用于大的样品,并且会对样品产生光毒害等,最重要的是设备的价格非常昂贵等。
光片荧光显微镜(light sheet fluorescence microscopy,LSFM)是一种适用于大型样品成像的新型显微系统[56]。LSFM虽然局限于传统的分辨率,但通过与其他超高分辨率技术的结合,如LSFM与结构光照明(structured illumination microscopy,SIM)显微技术结合而发展出的贝塞尔光束照明技术(bessel beam illumination microscopy)和晶格光片显微术(lattice Light-Sheet Microscopy,LLS),成为一种广受欢迎的成像显微技术。该显微技术采用薄片光进行侧向照明,并在垂直方向上探测成像,不仅成像速度非常快、光毒性低、信噪比高,而且可以对活体样本进行长时间的三维成像[57]。Zhao等[58]利用LSFM技术观测了哺乳动物活细胞核中RNAP II分子的动态,结果显示大多数(>70%)的转录位点来源于单个RNAP II分子,并且未检测到RNAP II分子之间发生显著的聚类反应。而在Cho等[36]研究中,通过LSFM成像显示小鼠胚胎干细胞核内出现大量RNAP II聚集的现象,进一步分析显示这些RNAP II分子有两种存在形式,分别可以形成小的、瞬时的团簇和较大的、稳定的团簇。当然,LSM也有其自身的局限性,如设备价格非常昂贵、横向分辨率较低,后期的大数据处理也比较困难等[59]。如何与其它超高分辨率显微技术更好的融合,将是LSFM未来研究的主要方向。
贝叶斯纳米显微技术(bayesian nanoscopy)就是一个将LSFM与超分辨显微技术结合的典型例子,已经发展成为一种新型快速成像方法[60]。贝叶斯算法是利用大量荧光分子的闪烁和漂白信息来还原样品的结构,通过将高斯光片照明引入到贝叶斯算法中[61],发展出一种新型的贝叶斯纳米显微技术。该技术有效地提高了显微成像的时空分辨率(50 nm的空间分辨率和4 s的时间分辨率),并大大减少了对样品的光毒性。Chen等[7]利用贝叶斯纳米显微技术的优势,观察到活细胞核中RNAP II分子的空间结构为不规则的球状几何形式,并揭示了RNAP II分子的动态组装和解聚过程。虽然贝叶斯纳米显微技术也存在局限性,比如不适合长期观察活细胞内的分子动态[57],但此方法是目前为止研究活体细胞内RNAP II分子动力学的最佳选择。
4 展望
综上所述,RNAP II的动力学、聚类反应及与转录因子之间的相互作用,对于揭示真核生物的转录调控具有重要的意义。近年来,多个实验室在活细胞荧光成像和分子标记技术的研究中取得了长足的进展,为实现在单分子水平上直接观察RNAP II的动态转录奠定了基础。例如,研究者可以选择CRISPR/Cas9或SLPs标记技术使目标分子携带荧光标签,再通过LSFM与超分辨显微技术结合的方法来实时成像和追踪目标分子,这样就使得活体细胞内的分子动态行为变得可视化。与动物细胞相比,植物的细胞存在细胞壁、叶绿体等物质极大的限制了植物细胞的活体成像,并且植物细胞也无法像动物细胞一样进行贴壁生长,这无疑对植物细胞的活体成像观察提出了更大的挑战,所以目前关于植物RNAP II的活体动态研究甚少,其响应不同生理条件下的动态调控机制也尚不清晰。因此,植物活体细胞内RNAP II动态调控转录是今后重点关注的方向,这也将会为揭示真核生物的转录机制带来更多的启示。
参考文献
真核生物RNA聚合酶Ⅱ核心启动子
[J].
Eukaryotic RNA polymerase Ⅱ core promoter
[J].
RNA Pol IV and V in gene silencing:rebel polymerases evolving away from Pol II’s rules
[J].DOI:10.1016/j.pbi.2015.07.005 URL [本文引用: 1]
Size-Dependent increase in RNA Polymerase II initiation rates mediates gene expression scaling with cell size
[J].DOI:10.1016/j.cub.2020.01.053 URL [本文引用: 1]
Corrigendum:structure of promoter-bound TFIID and model of human pre-initiation complex assembly
[J].
Measuring dynamics of eukaryotic transcription initiation:challenges, insights and opportunities
[J].DOI:10.1080/21541264.2017.1363017 URL [本文引用: 1]
Mediator and RNA polymerase II clusters associate in transcription-dependent condensates
[J].DOI:10.1126/science.aar4199 URL [本文引用: 2]
Study of RNA polymerase II clustering inside live-cell nuclei using bayesian nanoscopy
[J].DOI:10.1021/acsnano.5b07257 URL [本文引用: 2]
真核生物RNA聚合酶Ⅱ的研究进展
[J].
Research progress of eukaryotic RNA polymerase Ⅱ
[J].
拟南芥RNA聚合酶Ⅱ第三大亚基NRPB3调控气孔图式发育和分化
[D].
NRPB3, the third largest subunit of RNA polymerase II, is essential for stomatal patterning and differentiation in Arabidopsis
[D].
HECT家族泛素连接酶Wwp2泛素化修饰RNA聚合酶II大亚基Rpb1
[D].
HECT domain ubiquitin-protein ligase Wwp2 ubiquitinates the large subunit of RNA polymerase II
[D].
Yeast Spt6 reads multiple phosphorylation patterns of RNA polymerase II C-terminal domain in vitro
[J].DOI:10.1016/j.jmb.2020.05.007 URL [本文引用: 1]
The Nrd1-Nab3-Sen1 termination complex interacts with the Ser5-phosphorylated RNA polymerase II C-terminal domain
[J].DOI:10.1038/nsmb.1468 URL [本文引用: 1]
RNA聚合酶II中的CTD结构在mRNA转录和加工偶联过程中的重要作用
[J].
The Role of the carboxy-terminal domain of the RNA polymerase II on coupling mRNA transcription with processing
[J].
Transcriptional regulation of pattern-triggered immunity in plants
[J].
RNA聚合酶Ⅱ最大亚基RPB1的全长CTD通过控制细胞周期影响根尖干细胞池的命运
[D].
Intact CTD of RNA polymerse II maintains the root stem cell niches mediated by cell cycling control in arabidopsis
[D].
Enhancer activation requires trans-recruitment of a mega transcription factor complex
[J].DOI:10.1016/j.cell.2014.08.027 URL [本文引用: 1]
真核生物转录因子结合位点聚集区间的演化规律研究
[D].
The evolution of transcription factors binding sites clustered regions in eukaryote
[D].
Mediator undergoes a compositional change during transcriptional activation
[J].DOI:10.1016/j.molcel.2016.09.015 URL [本文引用: 1]
中介体亚基MED25调控植物激素信号转导的研究进展
[J].
Regulatory mechanism of mediator subunit MED25 on multi-phytohormone signaling pathways
[J].
Taf14 is required for the stabilization of transcription pre-initiation complex in saccharomyces cerevisiae
[J].
CREPT和p15RS结合RNA聚合酶II的结构生物学研究
[D].
Structural study of the interaction between CREPT/p15RS and RNA polymerase II
[D].
Single-molecule analysis of steroid receptor and cofactor action in living cells
[J].DOI:10.1038/ncomms15896 URL [本文引用: 1]
The code and beyond:transcription regulation by the RNA polymerase II carboxy-terminal domain
[J].DOI:10.1038/nrm.2017.10 URL [本文引用: 1]
CDKF;1 and CDKD protein kinases regulate phosphorylation of serine residues in the C-terminal domain of Arabidopsis RNA polymerase II
[J].
DOI:10.1105/tpc.112.096834
PMID:22547781
[本文引用: 1]

Phosphorylation of conserved Y₁S₂P₃T₄S₅P₆S₇ repeats in the C-terminal domain of largest subunit of RNA polymerase II (RNAPII CTD) plays a central role in the regulation of transcription and cotranscriptional RNA processing. Here, we show that Ser phosphorylation of Arabidopsis thaliana RNAPII CTD is governed by CYCLIN-DEPENDENT KINASE F;1 (CDKF;1), a unique plant-specific CTD S₇-kinase. CDKF;1 is required for in vivo activation of functionally redundant CYCLIN-DEPENDENT KINASE Ds (CDKDs), which are major CTD S₅-kinases that also phosphorylate in vitro the S₂ and S₇ CTD residues. Inactivation of CDKF;1 causes extreme dwarfism and sterility. Inhibition of CTD S₇-phosphorylation in germinating cdkf;1 seedlings is accompanied by 3'-polyadenylation defects of pre-microRNAs and transcripts encoding key regulators of small RNA biogenesis pathways. The cdkf;1 mutation also decreases the levels of both precursor and mature small RNAs without causing global downregulation of the protein-coding transcriptome and enhances the removal of introns that carry pre-microRNA stem-loops. A triple cdkd knockout mutant is not viable, but a combination of null and weak cdkd;3 alleles in a triple cdkd123* mutant permits semidwarf growth. Germinating cdkd123* seedlings show reduced CTD S₅-phosphorylation, accumulation of uncapped precursor microRNAs, and a parallel decrease in mature microRNA. During later development of cdkd123* seedlings, however, S₇-phosphorylation and unprocessed small RNA levels decline similarly as in the cdkf;1 mutant. Taken together, cotranscriptional processing and stability of a set of small RNAs and transcripts involved in their biogenesis are sensitive to changes in the phosphorylation of RNAPII CTD by CDKF;1 and CDKDs.
Release of human TFIIB from actively transcribing complexes is triggered upon synjournal of 7- and 9-nt RNAs
[J].DOI:10.1016/j.jmb.2020.05.005 URL [本文引用: 1]
Gene-specific control of tRNA expression by RNA polymerase II
[J].DOI:10.1016/j.molcel.2020.03.023 URL [本文引用: 1]
Controlling the elongation phase of transcription with P-TEFb
[J].DOI:10.1016/j.molcel.2006.06.014 URL [本文引用: 1]
The plant RNA polymerase II elongation complex:A hub coordinating transcript elongation and mRNA processing
[J].DOI:10.1080/21541264.2017.1356902 URL [本文引用: 1]
转录因子ⅡA及其识别元件在RNA聚合酶II指导的基因转录中的作用和调节机制的研究
[D].
The roles of transcription factor IIA and its recognition element in gene transcription directed by RNA polymerase II and their regulatory mechanisms
[D].
Termination of transcription by RNA polymerase II:BOOM!
[J].DOI:10.1016/j.tig.2020.05.008 URL [本文引用: 1]
转录终止及其调控
[J].
Transcriptional termination and its regulation
[J].
Genetic interactions and transcriptomics implicate fission yeast CTD prolyl isomerase pin1 as an agent of RNA 3' processing and transcription termination that functions via its effects on CTD phosphatase Ssu72
[J].
DOI:10.1093/nar/gkaa212
PMID:32282918
[本文引用: 1]

The phosphorylation pattern of Pol2 CTD Y1S2P3T4S5P6S7 repeats comprises an informational code coordinating transcription and RNA processing. cis-trans isomerization of CTD prolines expands the scope of the code in ways that are not well understood. Here we address this issue via analysis of fission yeast peptidyl-prolyl isomerase Pin1. A pin1Δ allele that does not affect growth per se is lethal in the absence of cleavage-polyadenylation factor (CPF) subunits Ppn1 and Swd22 and elicits growth defects absent CPF subunits Ctf1 and Dis2 and termination factor Rhn1. Whereas CTD S2A, T4A, and S7A mutants thrive in combination with pin1Δ, a Y1F mutant does not, nor do CTD mutants in which half the Pro3 or Pro6 residues are replaced by alanine. Phosphate-acquisition genes pho1, pho84 and tgp1 are repressed by upstream lncRNAs and are sensitive to changes in lncRNA 3' processing/termination. pin1Δ hyper-represses PHO gene expression and erases the de-repressive effect of CTD-S7A. Transcriptional profiling delineated sets of 56 and 22 protein-coding genes that are down-regulated and up-regulated in pin1Δ cells, respectively, 77% and 100% of which are downregulated/upregulated when the cis-proline-dependent Ssu72 CTD phosphatase is inactivated. Our results implicate Pin1 as a positive effector of 3' processing/termination that acts via Ssu72.© The Author(s) 2020. Published by Oxford University Press on behalf of Nucleic Acids Research.
细胞核肌动蛋白参与基因转录调控的分子机制研究
[D].
Studies on the molecular mechanism of nuclear actin in gene transcription regulation
[D].
Real-time dynamics of RNA polymerase II clustering in live human cells
[J].DOI:10.1126/science.1239053 URL [本文引用: 2]
可逆光开关荧光蛋白的开发及其在超高分辨率显微成像中的应用
[D].
Development of reversibly switchable fluorescent proteins for super-resolution imaging
[D].
活细胞单分子荧光标记——点亮生命微观世界的繁星
[J].
Single molecule fluorescent labeling of living cells-stars that light up the microscopic world of life
[J].
Green fluorescent protein(GFP)transgenic pig produced by somatic cell nuclear transfer
[J].
荧光标记技术的研究进展分析
[J].
Research progress of fluorescent labeling technology
[J].
红色荧光蛋白的研究进展
[J].
Research progress on red fluorescent protein
[J].
Using amino-labeled nucleotide probes for simultaneous single molecule RNA-DNA fISH
[J].DOI:10.1371/journal.pone.0107425 URL [本文引用: 1]
CRISPR/Cas9技术研究进展
[J].
Progress of CRISPR/Cas9 system
[J].
Super-resolution imaging of fluorescently labeled, endogenous RNA polymerase II in living cells with CRISPR/Cas9-mediated gene editing
[J].DOI:10.1038/srep35949 URL [本文引用: 2]
A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants
[J].DOI:10.1016/j.molp.2015.04.007 URL [本文引用: 1]
CRISPR/Cas9系统中的脱靶效应及检测技术研究进展
[J].
Research progress on off-target effects and detection techniques in CRISPR/Cas9 systems
[J].
The cation-π interaction enables a Halo-Tag fluorogenic probe for fast no-wash live cell imaging and gel-free protein quantification
[J].
DOI:10.1021/acs.biochem.7b00056
PMID:28221782
[本文引用: 1]

The design of fluorogenic probes for a Halo tag is highly desirable but challenging. Previous work achieved this goal by controlling the chemical switch of spirolactones upon the covalent conjugation between the Halo tag and probes or by incorporating a "channel dye" into the substrate binding tunnel of the Halo tag. In this work, we have developed a novel class of Halo-tag fluorogenic probes that are derived from solvatochromic fluorophores. The optimal probe, harboring a benzothiadiazole scaffold, exhibits a 1000-fold fluorescence enhancement upon reaction with the Halo tag. Structural, computational, and biochemical studies reveal that the benzene ring of a tryptophan residue engages in a cation-π interaction with the dimethylamino electron-donating group of the benzothiadiazole fluorophore in its excited state. We further demonstrate using noncanonical fluorinated tryptophan that the cation-π interaction directly contributes to the fluorogenicity of the benzothiadiazole fluorophore. Mechanistically, this interaction could contribute to the fluorogenicity by promoting the excited-state charge separation and inhibiting the twisting motion of the dimethylamino group, both leading to an enhanced fluorogenicity. Finally, we demonstrate the utility of the probe in no-wash direct imaging of Halo-tagged proteins in live cells. In addition, the fluorogenic nature of the probe enables a gel-free quantification of fusion proteins expressed in mammalian cells, an application that was not possible with previously nonfluorogenic Halo-tag probes. The unique mechanism revealed by this work suggests that incorporation of an excited-state cation-π interaction could be a feasible strategy for enhancing the optical performance of fluorophores and fluorogenic sensors.
Labeling strategies matter for super-resolution microscopy:a comparison between Halo-Tags and SNAP-tags
[J].DOI:10.1016/j.chembiol.2019.01.003 URL [本文引用: 1]
The HaloTag:a novel technology for cell imaging and protein analysis
[J].
Mapping transcription factor interactome networks using HaloTag protein arrays
[J].
超高分辨率显微技术研究进展
[J].
Progress of super-resolution microscopy
[J].
超分辨显微技术在活细胞中的应用与发展
[J].
Super-resolution microscopy applications and development in living cell
[J].
Super-resolution microscopy demystified
[J].DOI:10.1038/s41556-018-0251-8 URL [本文引用: 1]
随机光学重构显微镜在外泌体观察中的应用
[J].
Research progress on red fluorescent protein
[J].
超高分辨率荧光显微成像:2014年诺贝尔化学奖解析
[J].
Super-resolution fluorescence microscopy:analysis of the 2014 Nobel Prize in Chemistry
[J].
A compact STED microscope providing 3D nanoscale resolution
[J].DOI:10.1111/jmi.2009.236.issue-1 URL [本文引用: 1]
Single-molecule nanoscopy elucidates RNA polymerase II transcription at single genes in live cells
[J].DOI:10.1016/j.cell.2019.05.029 URL [本文引用: 1]
Light sheet fluorescence microscopy as a new method for unbiased three-dimensional analysis of vascular injury
[J].DOI:10.1093/cvr/cvaa037 URL [本文引用: 1]
结合光片照明与超分辨的三维荧光显微成像
[J].
Combination light-sheet illumination with super-resolution three-dimensional fluorescence microimaging
[J].
Spatial organization of RNA polymerase II inside a mammalian cell nucleus revealed by reflected light-sheet superresolution microscopy
[J].
光片荧光显微成像技术及应用进展
[J].
Progress in light-sheet fluorescence microscopy and applications
[J].
Bayesian localization microscopy reveals nanoscale podosome dynamics
[J].DOI:10.1038/nmeth.1812 URL [本文引用: 1]
Light-sheet bayesian microscopy enables deep-cell super-resolution imaging of heterochromatin in live human embryonic stem cells
[J].DOI:10.1186/2192-2853-2-1 URL [本文引用: 1]