蛋白质翻译后修饰之间的互作关系及其协同调控机理
1
2024
... 蛋白质活性的调节对生物体发挥正常功能至关重要,蛋白质的翻译后修饰(post-translational modification,PTM)可通过将小分子基团添加到目标蛋白质上改变蛋白的活性、稳定性或者亚细胞定位,使生物体能够迅速发生生理反应,从而响应快速变化的环境[1].植物蛋白质PTM类型主要包括:甲基化(methylation)、磷酸化(phosphorylation)、乙酰化(acetylation)、糖基化(glycosylation)、亚硝基化(nitrosylation)、泛素化(ubiquitination)等,它们协同调控植物体内的多种生理生化过程,在生长发育、代谢调控以及胁迫响应等生物学过程中发挥关键作用[2]. ...
Crosstalk between different post-translational modifications and its regulatory mechanisms in plant growth and development
1
2024
... 蛋白质活性的调节对生物体发挥正常功能至关重要,蛋白质的翻译后修饰(post-translational modification,PTM)可通过将小分子基团添加到目标蛋白质上改变蛋白的活性、稳定性或者亚细胞定位,使生物体能够迅速发生生理反应,从而响应快速变化的环境[1].植物蛋白质PTM类型主要包括:甲基化(methylation)、磷酸化(phosphorylation)、乙酰化(acetylation)、糖基化(glycosylation)、亚硝基化(nitrosylation)、泛素化(ubiquitination)等,它们协同调控植物体内的多种生理生化过程,在生长发育、代谢调控以及胁迫响应等生物学过程中发挥关键作用[2]. ...
Protein post-translational modification in plants: Regulation and beyond
1
2025
... 蛋白质活性的调节对生物体发挥正常功能至关重要,蛋白质的翻译后修饰(post-translational modification,PTM)可通过将小分子基团添加到目标蛋白质上改变蛋白的活性、稳定性或者亚细胞定位,使生物体能够迅速发生生理反应,从而响应快速变化的环境[1].植物蛋白质PTM类型主要包括:甲基化(methylation)、磷酸化(phosphorylation)、乙酰化(acetylation)、糖基化(glycosylation)、亚硝基化(nitrosylation)、泛素化(ubiquitination)等,它们协同调控植物体内的多种生理生化过程,在生长发育、代谢调控以及胁迫响应等生物学过程中发挥关键作用[2]. ...
Sumoylation in plants: mechanistic insights and its role in drought stress
3
2018
... 小泛素样修饰因子(small ubiquitin-like modifier, SUMO)是一类由约100个氨基酸残基组成小分子蛋白,在物种进化过程中高度保守,因其与泛素具有相似的作用过程和空间结构而得名,尽管SUMO在氨基酸水平上与泛素的序列一致性非常低(不到20%),但二者均具有典型的β折叠核心结构,包括5条反平行β链(β1-β5)和β4-β5间的α螺旋,呈现高度相似的三维构象[3].SUMO化是一种动态可逆的PTM,通过调节蛋白质的活性、稳定性、位置和相互作用,精细调控植物的生长发育和对胁迫的应答[3-4].SUMO化修饰遵循与泛素化修饰相似的ATP依赖型酶促级联反应机制,该过程由特异的E1活化酶(SUMO-activating enzyme)、E2结合酶(SUMO-conjugating enzyme)和E3连接酶(SUMO ligase)协同催化完成:SUMO化修饰始于前体蛋白的成熟加工,特异性水解酶对SUMO前体C端的若干个氨基酸进行水解,暴露其C端双甘氨酸残基从而形成活性SUMO,随后通过ATP依赖反应将SUMO羧基端腺苷酸化形成SUMO-AMP中间体,E1活化酶上半胱氨酸的活性巯基可以与SUMO-腺苷酸复合体形成过渡性的高能硫酯键,被E1活化的SUMO分子转移到E2结合酶的活性半胱氨酸上,形成SUMO-E2硫酯中间体,随后SUMO在E3连接酶的作用下通过异肽键共价连接至靶蛋白的赖氨酸ε-氨基上[3,5].被SUMO修饰的靶蛋白可受到两种动态机制的进一步调控:一是SUMO特异性蛋白酶水解异肽键,将SUMO从靶蛋白上切除,重新进入SUMO循环;二是靶标蛋白在SUMO E4连接酶的作用下形成多聚SUMO链,最终通过泛素-蛋白酶体系统被降解[5]. ...
... [3-4].SUMO化修饰遵循与泛素化修饰相似的ATP依赖型酶促级联反应机制,该过程由特异的E1活化酶(SUMO-activating enzyme)、E2结合酶(SUMO-conjugating enzyme)和E3连接酶(SUMO ligase)协同催化完成:SUMO化修饰始于前体蛋白的成熟加工,特异性水解酶对SUMO前体C端的若干个氨基酸进行水解,暴露其C端双甘氨酸残基从而形成活性SUMO,随后通过ATP依赖反应将SUMO羧基端腺苷酸化形成SUMO-AMP中间体,E1活化酶上半胱氨酸的活性巯基可以与SUMO-腺苷酸复合体形成过渡性的高能硫酯键,被E1活化的SUMO分子转移到E2结合酶的活性半胱氨酸上,形成SUMO-E2硫酯中间体,随后SUMO在E3连接酶的作用下通过异肽键共价连接至靶蛋白的赖氨酸ε-氨基上[3,5].被SUMO修饰的靶蛋白可受到两种动态机制的进一步调控:一是SUMO特异性蛋白酶水解异肽键,将SUMO从靶蛋白上切除,重新进入SUMO循环;二是靶标蛋白在SUMO E4连接酶的作用下形成多聚SUMO链,最终通过泛素-蛋白酶体系统被降解[5]. ...
... [3,5].被SUMO修饰的靶蛋白可受到两种动态机制的进一步调控:一是SUMO特异性蛋白酶水解异肽键,将SUMO从靶蛋白上切除,重新进入SUMO循环;二是靶标蛋白在SUMO E4连接酶的作用下形成多聚SUMO链,最终通过泛素-蛋白酶体系统被降解[5]. ...
Protein post-translational modifications (PTMs) unlocking resilience to abiotic stress in horticultural crops: a review
1
2025
... 小泛素样修饰因子(small ubiquitin-like modifier, SUMO)是一类由约100个氨基酸残基组成小分子蛋白,在物种进化过程中高度保守,因其与泛素具有相似的作用过程和空间结构而得名,尽管SUMO在氨基酸水平上与泛素的序列一致性非常低(不到20%),但二者均具有典型的β折叠核心结构,包括5条反平行β链(β1-β5)和β4-β5间的α螺旋,呈现高度相似的三维构象[3].SUMO化是一种动态可逆的PTM,通过调节蛋白质的活性、稳定性、位置和相互作用,精细调控植物的生长发育和对胁迫的应答[3-4].SUMO化修饰遵循与泛素化修饰相似的ATP依赖型酶促级联反应机制,该过程由特异的E1活化酶(SUMO-activating enzyme)、E2结合酶(SUMO-conjugating enzyme)和E3连接酶(SUMO ligase)协同催化完成:SUMO化修饰始于前体蛋白的成熟加工,特异性水解酶对SUMO前体C端的若干个氨基酸进行水解,暴露其C端双甘氨酸残基从而形成活性SUMO,随后通过ATP依赖反应将SUMO羧基端腺苷酸化形成SUMO-AMP中间体,E1活化酶上半胱氨酸的活性巯基可以与SUMO-腺苷酸复合体形成过渡性的高能硫酯键,被E1活化的SUMO分子转移到E2结合酶的活性半胱氨酸上,形成SUMO-E2硫酯中间体,随后SUMO在E3连接酶的作用下通过异肽键共价连接至靶蛋白的赖氨酸ε-氨基上[3,5].被SUMO修饰的靶蛋白可受到两种动态机制的进一步调控:一是SUMO特异性蛋白酶水解异肽键,将SUMO从靶蛋白上切除,重新进入SUMO循环;二是靶标蛋白在SUMO E4连接酶的作用下形成多聚SUMO链,最终通过泛素-蛋白酶体系统被降解[5]. ...
Analysis of small ubiquitin-like modifier (SUMO) targets reflects the essential nature of protein SUMOylation and provides insight to elucidate the role of SUMO in plant development
2
2015
... 小泛素样修饰因子(small ubiquitin-like modifier, SUMO)是一类由约100个氨基酸残基组成小分子蛋白,在物种进化过程中高度保守,因其与泛素具有相似的作用过程和空间结构而得名,尽管SUMO在氨基酸水平上与泛素的序列一致性非常低(不到20%),但二者均具有典型的β折叠核心结构,包括5条反平行β链(β1-β5)和β4-β5间的α螺旋,呈现高度相似的三维构象[3].SUMO化是一种动态可逆的PTM,通过调节蛋白质的活性、稳定性、位置和相互作用,精细调控植物的生长发育和对胁迫的应答[3-4].SUMO化修饰遵循与泛素化修饰相似的ATP依赖型酶促级联反应机制,该过程由特异的E1活化酶(SUMO-activating enzyme)、E2结合酶(SUMO-conjugating enzyme)和E3连接酶(SUMO ligase)协同催化完成:SUMO化修饰始于前体蛋白的成熟加工,特异性水解酶对SUMO前体C端的若干个氨基酸进行水解,暴露其C端双甘氨酸残基从而形成活性SUMO,随后通过ATP依赖反应将SUMO羧基端腺苷酸化形成SUMO-AMP中间体,E1活化酶上半胱氨酸的活性巯基可以与SUMO-腺苷酸复合体形成过渡性的高能硫酯键,被E1活化的SUMO分子转移到E2结合酶的活性半胱氨酸上,形成SUMO-E2硫酯中间体,随后SUMO在E3连接酶的作用下通过异肽键共价连接至靶蛋白的赖氨酸ε-氨基上[3,5].被SUMO修饰的靶蛋白可受到两种动态机制的进一步调控:一是SUMO特异性蛋白酶水解异肽键,将SUMO从靶蛋白上切除,重新进入SUMO循环;二是靶标蛋白在SUMO E4连接酶的作用下形成多聚SUMO链,最终通过泛素-蛋白酶体系统被降解[5]. ...
... [5]. ...
SUMO interacting motifs: structure and function
1
2021
... SUMO化修饰通常优先发生在具有保守序列ψKXE的赖氨酸(K)位点上,该序列被称为SUMO相互作用基序(SUMO-interacting motif, SIM),其中ψ表示疏水性氨基酸[6].与泛素化不同,SUMO化通常与蛋白质降解没有直接关系,而是通过调控靶蛋白的构象来影响其活性[7].由于SUMO化和泛素化修饰的靶点均为赖氨酸,早期研究认为这两种修饰过程存在拮抗作用;然而,后续研究表明,在人类(Homo sapiens)和酵母(Saccharomyces cerevisiae)中,SUMO链可通过SUMO靶向泛素连接酶(STUbL)招募泛素,暗示两者之间可能存在协同作用[8].值得注意的是,在SUMO化修饰的酶促级联反应中,E3连接酶作为决定底物选择特异性和修饰效率的核心调控成分[9],其功能多样性直接影响SUMO信号网络的时空特异性调控能力.近年来的研究发现,植物SUMO E3连接酶通过精密调控底物SUMO化水平,在光周期响应、开花调控、逆境信号转导等关键生物学过程中发挥枢纽作用.本文重点综述植物SUMO E3连接酶在生长发育调控和胁迫应答中的作用机制,以期为解析SUMO介导的植物环境适应性复杂调控网络提供新的视角. ...
Plant SUMO E3 ligases: function, structural organization, and connection with DNA
1
2021
... SUMO化修饰通常优先发生在具有保守序列ψKXE的赖氨酸(K)位点上,该序列被称为SUMO相互作用基序(SUMO-interacting motif, SIM),其中ψ表示疏水性氨基酸[6].与泛素化不同,SUMO化通常与蛋白质降解没有直接关系,而是通过调控靶蛋白的构象来影响其活性[7].由于SUMO化和泛素化修饰的靶点均为赖氨酸,早期研究认为这两种修饰过程存在拮抗作用;然而,后续研究表明,在人类(Homo sapiens)和酵母(Saccharomyces cerevisiae)中,SUMO链可通过SUMO靶向泛素连接酶(STUbL)招募泛素,暗示两者之间可能存在协同作用[8].值得注意的是,在SUMO化修饰的酶促级联反应中,E3连接酶作为决定底物选择特异性和修饰效率的核心调控成分[9],其功能多样性直接影响SUMO信号网络的时空特异性调控能力.近年来的研究发现,植物SUMO E3连接酶通过精密调控底物SUMO化水平,在光周期响应、开花调控、逆境信号转导等关键生物学过程中发挥枢纽作用.本文重点综述植物SUMO E3连接酶在生长发育调控和胁迫应答中的作用机制,以期为解析SUMO介导的植物环境适应性复杂调控网络提供新的视角. ...
An additional role for SUMO in ubiquitin-mediated proteolysis
1
2009
... SUMO化修饰通常优先发生在具有保守序列ψKXE的赖氨酸(K)位点上,该序列被称为SUMO相互作用基序(SUMO-interacting motif, SIM),其中ψ表示疏水性氨基酸[6].与泛素化不同,SUMO化通常与蛋白质降解没有直接关系,而是通过调控靶蛋白的构象来影响其活性[7].由于SUMO化和泛素化修饰的靶点均为赖氨酸,早期研究认为这两种修饰过程存在拮抗作用;然而,后续研究表明,在人类(Homo sapiens)和酵母(Saccharomyces cerevisiae)中,SUMO链可通过SUMO靶向泛素连接酶(STUbL)招募泛素,暗示两者之间可能存在协同作用[8].值得注意的是,在SUMO化修饰的酶促级联反应中,E3连接酶作为决定底物选择特异性和修饰效率的核心调控成分[9],其功能多样性直接影响SUMO信号网络的时空特异性调控能力.近年来的研究发现,植物SUMO E3连接酶通过精密调控底物SUMO化水平,在光周期响应、开花调控、逆境信号转导等关键生物学过程中发挥枢纽作用.本文重点综述植物SUMO E3连接酶在生长发育调控和胁迫应答中的作用机制,以期为解析SUMO介导的植物环境适应性复杂调控网络提供新的视角. ...
The expanding universe of ubiquitin and ubiquitin-like modifiers
1
2012
... SUMO化修饰通常优先发生在具有保守序列ψKXE的赖氨酸(K)位点上,该序列被称为SUMO相互作用基序(SUMO-interacting motif, SIM),其中ψ表示疏水性氨基酸[6].与泛素化不同,SUMO化通常与蛋白质降解没有直接关系,而是通过调控靶蛋白的构象来影响其活性[7].由于SUMO化和泛素化修饰的靶点均为赖氨酸,早期研究认为这两种修饰过程存在拮抗作用;然而,后续研究表明,在人类(Homo sapiens)和酵母(Saccharomyces cerevisiae)中,SUMO链可通过SUMO靶向泛素连接酶(STUbL)招募泛素,暗示两者之间可能存在协同作用[8].值得注意的是,在SUMO化修饰的酶促级联反应中,E3连接酶作为决定底物选择特异性和修饰效率的核心调控成分[9],其功能多样性直接影响SUMO信号网络的时空特异性调控能力.近年来的研究发现,植物SUMO E3连接酶通过精密调控底物SUMO化水平,在光周期响应、开花调控、逆境信号转导等关键生物学过程中发挥枢纽作用.本文重点综述植物SUMO E3连接酶在生长发育调控和胁迫应答中的作用机制,以期为解析SUMO介导的植物环境适应性复杂调控网络提供新的视角. ...
Structural basis for E2-mediated SUMO conjugation revealed by a complex between ubiquitin-conjugating enzyme Ubc9 and RanGAP1
1
2002
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
An E3-like factor that promotes SUMO conjugation to the yeast septins
1
2001
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
Structure of the siz/PIAS SUMO E3 ligase Siz1 and determinants required for SUMO modification of PCNA
1
2009
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
The nucleoporin RanBP2 has SUMO1 E3 ligase activity
1
2002
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
The polycomb protein Pc2 is a SUMO E3
1
2003
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
The Arabidopsis SUMO E3 ligase SIZ1 controls phosphate deficiency responses
3
2005
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
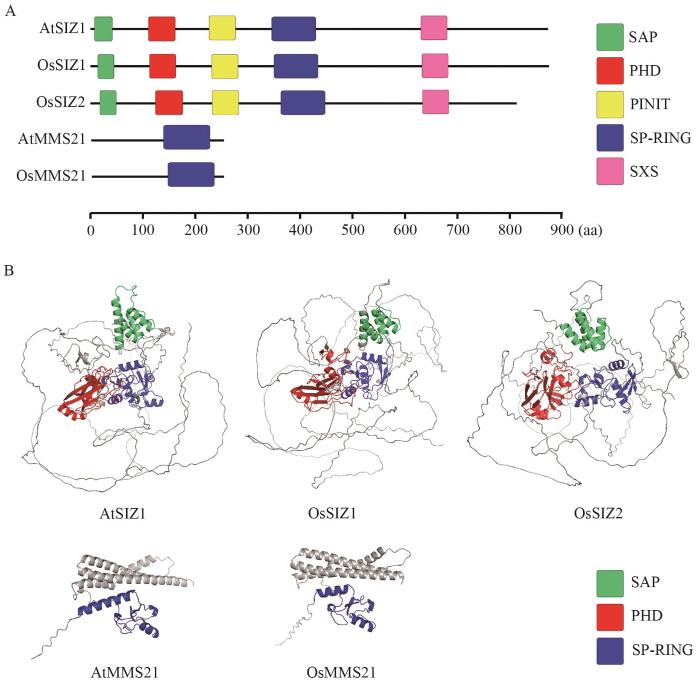
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
The Arabidopsis SUMO E3 ligase AtMMS21, a homologue of NSE2/MMS21, regulates cell proliferation in the root
3
2009
... 在体外实验中,某些底物蛋白即使在没有SUMO E3连接酶的情况下,也能被高效地SUMO化修饰[10],但体内研究尚未发现SUMO E3非依赖性修饰的证据,表明SUMO E3连接酶通过增强底物特异性或修饰效率发挥作用.迄今为止,研究者们在动物和真菌中共发现了3种SUMO E3连接酶,第1种是SIZ1(SAP and Miz 1)/PIAS(Protein inhibitor of activated STAT)类型.Johnson等[11]首次在酵母中鉴定了SUMO E3连接酶Siz1,并揭示其能够通过促进SUMO分子与特定底物的结合来调节蛋白质活性,这类E3中包含类似泛素E3的保守RING结构域,其作用是结合SCE-SUMO复合体[12];第2种是在人类中发现的RanBP2(RAN binding protein 2),然而其底物RanGAP1在真菌和植物中缺乏保守性,无法被RanBP2介导完成SUMO化修饰,因此,目前认为RanBP2类SUMO E3连接酶仅存在于动物中[13];第3种是2003年在人类中发现的多梳蛋白Pc2(Polycomb2)[14],在拟南芥(Arabidopsisthaliana)中是否存在与Pc2同源的SUMO连接酶尚未可知.目前植物中发现的2种SUMO E3连接酶分别是SIZ1和MMS21/HPY2,均与动物中的SIZ1/PIAS类SUMO E3连接酶同源[15-16],其中SIZ1的相关报道较多. ...
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
Structural and functional studies of SIZ1, a PIAS-type SUMO E3 ligase from Arabidopsis
2
2010
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
Functional characterization of the SIZ/PIAS-type SUMO E3 ligases, OsSIZ1 and OsSIZ2 in rice
1
2010
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
MMS21/HPY2 and SIZ1, two Arabidopsis SUMO E3 ligases, have distinct functions in development
3
2012
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... ,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... [19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
The Arabidopsis E3 SUMO ligase SIZ1 regulates plant growth and drought responses
1
2007
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
SIZ1 small ubiquitin-like modifier E3 ligase facilitates basal thermotolerance in Arabidopsis independent of salicylic acid
2
2006
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
The small ubiquitin-like modifier E3 ligase MdSIZ1 promotes anthocyanin accumulation by sumoylating MdMYB1 under low-temperature conditions in apple
2
2017
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... SIZ1在植物低温胁迫响应中具有关键的调控作用.拟南芥AtSIZ1通过介导ICE1转录因子的SUMO化修饰显著增强其蛋白稳定性及转录活性,进而激活CBF3/DREB1A信号通路,最终促进冷响应基因表达并提高植物抗冻性;SUMO化的ICE1可形成双重调控机制:一方面通过抑制MYB15转录因子的活性解除其对CBF通路的负调控,另一方面阻断泛素E3泛素连接酶HOS1介导的ICE1泛素化降解,从而维持低温信号通路的持续激活[42].进一步研究发现,SIZ1通过调控SA代谢途径影响拟南芥耐寒性,siz1突变体中SA异常积累,且CBF3/DREB1A及其下游基因表达下降,对低温和冷冻条件的敏感性提高,而降低SA水平可部分恢复其低温耐受性,证实SA信号与SIZ1途径在拟南芥对冷胁迫的响应中存在拮抗作用[43].前人在拟南芥中研究发现,泛素E3连接酶COP1与AtSIZ1相互作用,并通过多泛素化修饰促进其降解,当COP1活性被抑制或缺失时,AtSIZ1蛋白水平显著增加,从而导致植株中蛋白SUMO化水平升高,这种调控机制使得植物对低温、干旱和高盐等逆境条件表现出更强的耐受性;此外,AtSIZ1还可能通过SUMO化修饰反向调控COP1的活性[44],这一发现揭示了泛素化和SUMO化系统之间的交叉调控机制.苹果MdSIZ1通过SUMO化修饰稳定MdMYB1转录因子,促进低温诱导的花青素生物合成[22],MdMYB2作为上游调控因子,可直接结合MdSIZ1启动子并增强其转录活性,从而形成MdMYB2-MdSIZ1-MdMYB1级联调控网络[45].拟南芥HPY2通过对细胞分裂素响应因子ARR1的K236位点进行SUMO化修饰激活其转录活性,在低温或细胞分裂素处理下ARR1 D89位点的磷酸化介导其与HPY2结合,SUMO化的ARR1进一步促进组蛋白H3乙酰化,增强靶基因表达,参与细胞分裂素与低温信号的整合调控[46]. ...
Salicylic acid-mediated innate immunity in Arabidopsis is regulated by SIZ1 SUMO E3 ligase
2
2007
... 如图1所示,植物SIZ1蛋白一般包含5个保守结构域:SAP(scaffold attachment factor A/B/acinus/PIAS)结构域,参与DNA结合;PINIT(Pro-Ile-Asn-Ile-Thr)基序,介导其核定位;SP-RING(Siz/PIAS RING)催化域,赋予其E3连接酶活性;SXS(Ser-X-Ser,X代表任意氨基酸)基序,有助于增强SIZ1对SUMO底物的识别能力,促进SUMO从E2酶向靶蛋白的转移;以及植物特有的PHD锌指结构域,参与催化底物连接的过程,其中PINIT和SP-RING结构域是SIZ1发挥SUMO E3连接酶活性必不可少的[17-18].与SIZ1不同,MMS21仅保留SP-RING结构域,而缺失了SAP、PINIT和PHD等结构域[19].Cheong等 [17]揭示了SIZ1蛋白各结构域在植物中的生物学功能:PHD和PINIT结构域参与糖和光协同调控的下胚轴伸长;SP-RING结构域调控植物中水杨酸(salicylic acid, SA)积累、基础耐热性以及对脱落酸(abscisic acid, ABA)的敏感性;SXS结构域参与调控ABA介导的子叶变绿;SAP结构域则主要参与DNA结合和染色质调控,而不直接参与SUMO化或SA积累.拟南芥SIZ1广泛表达于植物叶片、根尖及生殖器官等[20],通过调控SA信号参与非生物胁迫响应(如低磷、盐/冷/热胁迫)及免疫调节[15,21-22],siz1突变体表现为植株矮化,且通过积累SA增强植株抗病性;而MMS21则在分生组织细胞中特异表达,主要调控细胞周期进程和根发育,其突变体hpy2-2地上部表型正常,但根系出现严重缺陷[16,19,23].遗传互补实验表明,拟南芥SIZ1与MMS21功能独立且不可相互替代:siz1/mms21双突变体胚胎致死,且在hpy2-1背景中异源表达SIZ1无法回补hpy2-1表型,证实两者在SUMO化调控中具有协同性与特异性[19].植物中的SIZ1与动物Siz/PIAS家族成员具有同源性,但其特有的PHD结构域可能反映了植物在适应逆境过程中演化出的独特调控机制. ...
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
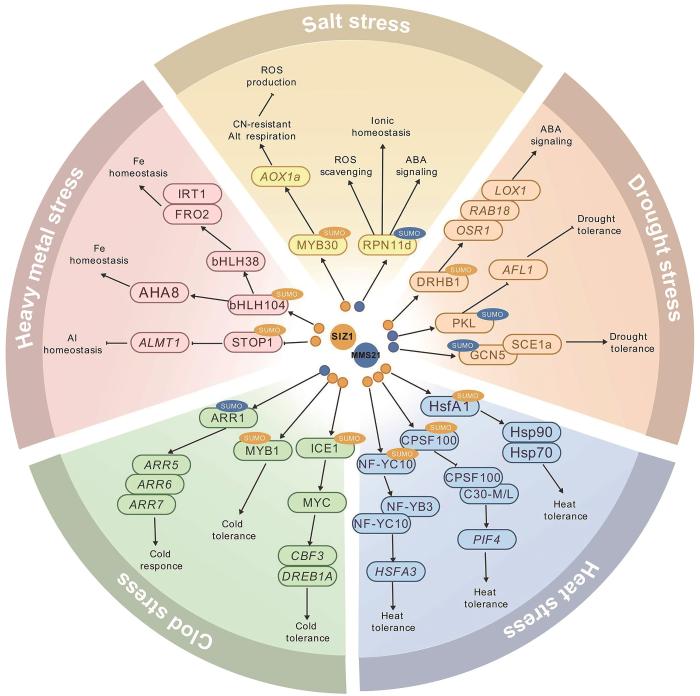
Increased tolerance to salt stress in the phosphate-accumulating Arabidopsis mutants siz1 and pho2
1
2011
... 在拟南芥中,siz1突变体在盐胁迫下通过抑制Na+积累表现出一定程度的耐盐性,其机制独立于SOS3信号通路[24],而异源过表达水稻(Oryzasativa)OsSIZ1显著提高了转基因拟南芥的耐盐性,植株中K+含量增加、Na+/K+比例降低,渗透调节基因P5CS表达上调[25].Han等[26]进一步研究证实了AtSIZ1通过调控Na+/K+稳态和渗透平衡来应对盐胁迫,AtSIZ1过表达植株在盐胁迫下通过激活SOS1、AtP5CS1等关键基因维持离子稳态和积累渗透调节物质.AtSIZ1通过介导MYB30的SUMO化增强其对AOX1a启动子的结合能力,从而上调AOX1a表达,促进交替呼吸,降低盐胁迫下拟南芥植株中活性氧(reactive oxygen species,ROS)的积累,siz1-2突变体表现出盐敏感表型,而过表达AOX1a可恢复其耐盐能力,表明SIZ1通过调控MYB30-AOX1a途径正调控拟南芥耐盐性[27].辣椒(Capsicumannuum)CaSIZ1通过与CaABI5互作介导ABA诱导的SUMO化修饰,正调控ABA响应基因表达,敲低CaSIZ1导致辣椒耐盐性显著下降[28].最新研究发现,大豆(Glycine max)GmMMS21通过SUMO化26S蛋白酶调节颗粒亚基GmRPN11d,在盐胁迫下显著增强其蛋白稳定性;过表达GmRPN11d可提高拟南芥和大豆毛状根对盐胁迫的耐受性,转基因植株中ROS清除能力增强,Na+/K+离子稳态改善,ABA信号通路被激活[29]. ...
Overexpression of the rice gene OsSIZ1 in Arabidopsis improves drought-, heat-, and salt-tolerance simultaneously
4
2018
... 在拟南芥中,siz1突变体在盐胁迫下通过抑制Na+积累表现出一定程度的耐盐性,其机制独立于SOS3信号通路[24],而异源过表达水稻(Oryzasativa)OsSIZ1显著提高了转基因拟南芥的耐盐性,植株中K+含量增加、Na+/K+比例降低,渗透调节基因P5CS表达上调[25].Han等[26]进一步研究证实了AtSIZ1通过调控Na+/K+稳态和渗透平衡来应对盐胁迫,AtSIZ1过表达植株在盐胁迫下通过激活SOS1、AtP5CS1等关键基因维持离子稳态和积累渗透调节物质.AtSIZ1通过介导MYB30的SUMO化增强其对AOX1a启动子的结合能力,从而上调AOX1a表达,促进交替呼吸,降低盐胁迫下拟南芥植株中活性氧(reactive oxygen species,ROS)的积累,siz1-2突变体表现出盐敏感表型,而过表达AOX1a可恢复其耐盐能力,表明SIZ1通过调控MYB30-AOX1a途径正调控拟南芥耐盐性[27].辣椒(Capsicumannuum)CaSIZ1通过与CaABI5互作介导ABA诱导的SUMO化修饰,正调控ABA响应基因表达,敲低CaSIZ1导致辣椒耐盐性显著下降[28].最新研究发现,大豆(Glycine max)GmMMS21通过SUMO化26S蛋白酶调节颗粒亚基GmRPN11d,在盐胁迫下显著增强其蛋白稳定性;过表达GmRPN11d可提高拟南芥和大豆毛状根对盐胁迫的耐受性,转基因植株中ROS清除能力增强,Na+/K+离子稳态改善,ABA信号通路被激活[29]. ...
... 此外,SIZ1还能通过影响渗透调节参与植物干旱胁迫应答,在拟南芥中异源表达OsSIZ1通过上调脯氨酸合成基因P5CS的表达增强渗透调节能力,从而提高了转基因植株对干旱、盐、高温等多种非生物胁迫的耐受能力[25],SUMO系统的动态平衡对植物响应干旱至关重要.在拟南芥中,去SUMO化酶ULP1c/d与SIZ1形成调控回路,ulp1culp1d双突变体因气孔开度增加而表现出对渗透敏感性增强,而siz1突变体则表现出气孔开度减小、耐旱性增强,siz1ulp1culp1d三重突变体的耐旱性表型与突变体siz1类似,证实了ULP1c和ULP1d蛋白酶作为生长的调控因子发挥冗余作用,作用于SIZ1下游来控制植物对干旱的应答[32]. ...
... SUMO E3连接酶介导的PTM在植物逆境响应和生长发育的多个过程中发挥重要作用,因此被视为改良作物性状的新途径.SUMO E3连接酶的功能鉴定及相关机制的研究突破为利用SUMO通路对作物进行遗传改良提供了新的切入点.育种家们可以将SUMO通路相关基因作为育种或基因编辑的目标,例如,通过基因工程适度上调关键的SUMO E3连接酶(如提高水稻OsSIZ1或OsMMS21的表达)[25, 72],有望增强作物对干旱、盐碱等胁迫的耐受性,促进作物在逆境条件下的稳产.SUMO化修饰在植物根系发育调控中的重要作用也为作物遗传改良提供了新的思路.通过有针对性对调控根系发育的SUMO相关基因进行改造,有望培育出更能适应贫瘠土壤、具备较高产量潜力的作物新种质. ...
... 尽管SUMO通路在作物性状改良中具有应用潜力,但仍面临技术瓶颈.其关键挑战在于实现特异性调控:SUMO E3连接酶具有多种底物,过度表达或敲除关键基因可能产生多重效应,既可能增强抗逆性,也可能扰乱激素平衡、影响生长发育.因此,需采用组织特异或诱导型策略,在特定时空精确调控SUMO化水平.此外,需要建立简单易行的检测体系:相较于传统育种性状,植物中蛋白的SUMO化水平难以直接检测.目前植物种蛋白SUMO化水平的检测主要依赖实验室条件下的分子分析,未来或可借助标记报告系统,在植物体内指示SUMO通路的活性状态,加速育种筛选进程.三是SUMO化通路的成员组成和作用机制在不同植物物种中可能存在差异,还需深入研究阐明.有趣的是,一些跨物种研究证实SUMO途径在植物中具有较强的保守性,说明不同作物中的SUMO调控网络在整体框架上可能存在共性[106-107].植物SIZ1和MMS21/HPY2型SUMO E3连接酶均包含进化上高度保守的SP-RING核心催化结构域,保障其在调控植物发育、胁迫应答和维持基因组稳定性中的基本功能;然而,非结构域区域的序列特征和某些植物中的基因家族扩张也赋予了SUMO E3连接酶在底物识别与调控途径方面的特异性调控能力和功能分化潜力.例如,拟南芥中的AtSIZ1在免疫抑制方面具有独特作用,如抑制TPR1-HDA19复合物的活性以阻止过度免疫反应[98],而水稻中的OsSIZ1对干旱、高温和盐胁迫表现出更强的适应性[25],可能涉及不同SUMO化底物的选择.这种保守性与多样性的结合使得SUMO E3连接酶能够灵活参与并调节复杂的信号网络,以适应环境变化与植物自身的生长需求.因此,结合比较基因组学与模式植物研究,系统鉴定各主要农作物中特有的SUMO E3及其功能靶标,将有助于在保守机制基础上制定更具作物特异性的分子育种策略.未来,随着研究的不断深入,SUMO E3连接酶有望被应用于作物抗逆性和发育相关性状的遗传改良,为培育适应复杂环境条件的作物新品种提供新的途径. ...
AtSIZ1 improves salt tolerance by maintaining ionic homeostasis and osmotic balance in Arabidopsis
1
2019
... 在拟南芥中,siz1突变体在盐胁迫下通过抑制Na+积累表现出一定程度的耐盐性,其机制独立于SOS3信号通路[24],而异源过表达水稻(Oryzasativa)OsSIZ1显著提高了转基因拟南芥的耐盐性,植株中K+含量增加、Na+/K+比例降低,渗透调节基因P5CS表达上调[25].Han等[26]进一步研究证实了AtSIZ1通过调控Na+/K+稳态和渗透平衡来应对盐胁迫,AtSIZ1过表达植株在盐胁迫下通过激活SOS1、AtP5CS1等关键基因维持离子稳态和积累渗透调节物质.AtSIZ1通过介导MYB30的SUMO化增强其对AOX1a启动子的结合能力,从而上调AOX1a表达,促进交替呼吸,降低盐胁迫下拟南芥植株中活性氧(reactive oxygen species,ROS)的积累,siz1-2突变体表现出盐敏感表型,而过表达AOX1a可恢复其耐盐能力,表明SIZ1通过调控MYB30-AOX1a途径正调控拟南芥耐盐性[27].辣椒(Capsicumannuum)CaSIZ1通过与CaABI5互作介导ABA诱导的SUMO化修饰,正调控ABA响应基因表达,敲低CaSIZ1导致辣椒耐盐性显著下降[28].最新研究发现,大豆(Glycine max)GmMMS21通过SUMO化26S蛋白酶调节颗粒亚基GmRPN11d,在盐胁迫下显著增强其蛋白稳定性;过表达GmRPN11d可提高拟南芥和大豆毛状根对盐胁迫的耐受性,转基因植株中ROS清除能力增强,Na+/K+离子稳态改善,ABA信号通路被激活[29]. ...
SUMOylation of MYB30 enhances salt tolerance by elevating alternative respiration via transcriptionally upregulating AOX1a in Arabidopsis
1
2020
... 在拟南芥中,siz1突变体在盐胁迫下通过抑制Na+积累表现出一定程度的耐盐性,其机制独立于SOS3信号通路[24],而异源过表达水稻(Oryzasativa)OsSIZ1显著提高了转基因拟南芥的耐盐性,植株中K+含量增加、Na+/K+比例降低,渗透调节基因P5CS表达上调[25].Han等[26]进一步研究证实了AtSIZ1通过调控Na+/K+稳态和渗透平衡来应对盐胁迫,AtSIZ1过表达植株在盐胁迫下通过激活SOS1、AtP5CS1等关键基因维持离子稳态和积累渗透调节物质.AtSIZ1通过介导MYB30的SUMO化增强其对AOX1a启动子的结合能力,从而上调AOX1a表达,促进交替呼吸,降低盐胁迫下拟南芥植株中活性氧(reactive oxygen species,ROS)的积累,siz1-2突变体表现出盐敏感表型,而过表达AOX1a可恢复其耐盐能力,表明SIZ1通过调控MYB30-AOX1a途径正调控拟南芥耐盐性[27].辣椒(Capsicumannuum)CaSIZ1通过与CaABI5互作介导ABA诱导的SUMO化修饰,正调控ABA响应基因表达,敲低CaSIZ1导致辣椒耐盐性显著下降[28].最新研究发现,大豆(Glycine max)GmMMS21通过SUMO化26S蛋白酶调节颗粒亚基GmRPN11d,在盐胁迫下显著增强其蛋白稳定性;过表达GmRPN11d可提高拟南芥和大豆毛状根对盐胁迫的耐受性,转基因植株中ROS清除能力增强,Na+/K+离子稳态改善,ABA信号通路被激活[29]. ...
Capsicum SIZ1 contributes to ABA-induced SUMOylation in pepper
1
2022
... 在拟南芥中,siz1突变体在盐胁迫下通过抑制Na+积累表现出一定程度的耐盐性,其机制独立于SOS3信号通路[24],而异源过表达水稻(Oryzasativa)OsSIZ1显著提高了转基因拟南芥的耐盐性,植株中K+含量增加、Na+/K+比例降低,渗透调节基因P5CS表达上调[25].Han等[26]进一步研究证实了AtSIZ1通过调控Na+/K+稳态和渗透平衡来应对盐胁迫,AtSIZ1过表达植株在盐胁迫下通过激活SOS1、AtP5CS1等关键基因维持离子稳态和积累渗透调节物质.AtSIZ1通过介导MYB30的SUMO化增强其对AOX1a启动子的结合能力,从而上调AOX1a表达,促进交替呼吸,降低盐胁迫下拟南芥植株中活性氧(reactive oxygen species,ROS)的积累,siz1-2突变体表现出盐敏感表型,而过表达AOX1a可恢复其耐盐能力,表明SIZ1通过调控MYB30-AOX1a途径正调控拟南芥耐盐性[27].辣椒(Capsicumannuum)CaSIZ1通过与CaABI5互作介导ABA诱导的SUMO化修饰,正调控ABA响应基因表达,敲低CaSIZ1导致辣椒耐盐性显著下降[28].最新研究发现,大豆(Glycine max)GmMMS21通过SUMO化26S蛋白酶调节颗粒亚基GmRPN11d,在盐胁迫下显著增强其蛋白稳定性;过表达GmRPN11d可提高拟南芥和大豆毛状根对盐胁迫的耐受性,转基因植株中ROS清除能力增强,Na+/K+离子稳态改善,ABA信号通路被激活[29]. ...
GmRPN11d positively regulates plant salinity tolerance by improving protein stability through SUMOylation
1
2025
... 在拟南芥中,siz1突变体在盐胁迫下通过抑制Na+积累表现出一定程度的耐盐性,其机制独立于SOS3信号通路[24],而异源过表达水稻(Oryzasativa)OsSIZ1显著提高了转基因拟南芥的耐盐性,植株中K+含量增加、Na+/K+比例降低,渗透调节基因P5CS表达上调[25].Han等[26]进一步研究证实了AtSIZ1通过调控Na+/K+稳态和渗透平衡来应对盐胁迫,AtSIZ1过表达植株在盐胁迫下通过激活SOS1、AtP5CS1等关键基因维持离子稳态和积累渗透调节物质.AtSIZ1通过介导MYB30的SUMO化增强其对AOX1a启动子的结合能力,从而上调AOX1a表达,促进交替呼吸,降低盐胁迫下拟南芥植株中活性氧(reactive oxygen species,ROS)的积累,siz1-2突变体表现出盐敏感表型,而过表达AOX1a可恢复其耐盐能力,表明SIZ1通过调控MYB30-AOX1a途径正调控拟南芥耐盐性[27].辣椒(Capsicumannuum)CaSIZ1通过与CaABI5互作介导ABA诱导的SUMO化修饰,正调控ABA响应基因表达,敲低CaSIZ1导致辣椒耐盐性显著下降[28].最新研究发现,大豆(Glycine max)GmMMS21通过SUMO化26S蛋白酶调节颗粒亚基GmRPN11d,在盐胁迫下显著增强其蛋白稳定性;过表达GmRPN11d可提高拟南芥和大豆毛状根对盐胁迫的耐受性,转基因植株中ROS清除能力增强,Na+/K+离子稳态改善,ABA信号通路被激活[29]. ...
Pepper SUMO E3 ligase CaDSIZ1 enhances drought tolerance by stabilizing the transcription factor CaDRHB1
1
2022
... SUMO E3连接酶通过多维机制调控植物干旱胁迫响应.前人研究发现,SIZ1能够通过ABA信号通路参与植物干旱应答,辣椒CaDSIZ1通过SUMO化修饰稳定转录因子CaDRHB1,并增强其对CaOSR1、CaRAB18和CaLOX1等启动子区包含ABA响应元件的下游基因的转录激活活性,从而在干旱胁迫下增强植物的耐旱能力[30];而AtMMS21则可能通过调控ABA依赖途径基因负调控干旱胁迫应答,AtMMS21的组成性表达抑制ABA和干旱胁迫响应基因RAB18、RD29A和RD29B的表达,使植株耐旱性降低,而mms21突变体中胁迫响应基因COR15、RD22和P5CS1的表达显著上调,使其表现出更强的耐旱性[31]. ...
SUMO E3 ligase AtMMS21 regulates drought tolerance in Arabidopsis thaliana
1
2013
... SUMO E3连接酶通过多维机制调控植物干旱胁迫响应.前人研究发现,SIZ1能够通过ABA信号通路参与植物干旱应答,辣椒CaDSIZ1通过SUMO化修饰稳定转录因子CaDRHB1,并增强其对CaOSR1、CaRAB18和CaLOX1等启动子区包含ABA响应元件的下游基因的转录激活活性,从而在干旱胁迫下增强植物的耐旱能力[30];而AtMMS21则可能通过调控ABA依赖途径基因负调控干旱胁迫应答,AtMMS21的组成性表达抑制ABA和干旱胁迫响应基因RAB18、RD29A和RD29B的表达,使植株耐旱性降低,而mms21突变体中胁迫响应基因COR15、RD22和P5CS1的表达显著上调,使其表现出更强的耐旱性[31]. ...
SUMO proteases ULP1c and ULP1d are required for development and osmotic stress responses in Arabidopsis thaliana
1
2016
... 此外,SIZ1还能通过影响渗透调节参与植物干旱胁迫应答,在拟南芥中异源表达OsSIZ1通过上调脯氨酸合成基因P5CS的表达增强渗透调节能力,从而提高了转基因植株对干旱、盐、高温等多种非生物胁迫的耐受能力[25],SUMO系统的动态平衡对植物响应干旱至关重要.在拟南芥中,去SUMO化酶ULP1c/d与SIZ1形成调控回路,ulp1culp1d双突变体因气孔开度增加而表现出对渗透敏感性增强,而siz1突变体则表现出气孔开度减小、耐旱性增强,siz1ulp1culp1d三重突变体的耐旱性表型与突变体siz1类似,证实了ULP1c和ULP1d蛋白酶作为生长的调控因子发挥冗余作用,作用于SIZ1下游来控制植物对干旱的应答[32]. ...
PKL is stabilized by MMS21 to negatively regulate Arabidopsis drought tolerance through directly repressing AFL1 transcription
1
2023
... 研究发现,拟南芥MMS21通过与染色质重塑因子PICKLE(PKL)直接互作促进其SUMO化并增强其蛋白稳定性,PKL作为转录抑制因子结合干旱响应基因AFL1的启动子,促进其染色质所在区域组蛋白H3K27me3的沉积,从而抑制其表达;pkl和mms21-1突变体耐旱性均增强,mms21-1植株中PKL蛋白水平下降,证实了MMS21通过影响PKL稳定性参与调控植物耐旱性[33].玉米ZmMMS21与E2结合酶ZmSCE1a协同作用介导组蛋白乙酰转移酶ZmGCN5的SUMO化修饰,从而增强其稳定性,过表达ZmGCN5能有效降低O2-积累、丙二醛含量和电解质渗漏,从而提高玉米抗旱性,ZmSCE1a增强了ZmGCN5的稳定性,从而减轻了干旱诱导的氧化损伤,增强了玉米对干旱胁迫的响应[34]. ...
ZmSCE1a positively regulates drought tolerance by enhancing the stability of ZmGCN5
1
2024
... 研究发现,拟南芥MMS21通过与染色质重塑因子PICKLE(PKL)直接互作促进其SUMO化并增强其蛋白稳定性,PKL作为转录抑制因子结合干旱响应基因AFL1的启动子,促进其染色质所在区域组蛋白H3K27me3的沉积,从而抑制其表达;pkl和mms21-1突变体耐旱性均增强,mms21-1植株中PKL蛋白水平下降,证实了MMS21通过影响PKL稳定性参与调控植物耐旱性[33].玉米ZmMMS21与E2结合酶ZmSCE1a协同作用介导组蛋白乙酰转移酶ZmGCN5的SUMO化修饰,从而增强其稳定性,过表达ZmGCN5能有效降低O2-积累、丙二醛含量和电解质渗漏,从而提高玉米抗旱性,ZmSCE1a增强了ZmGCN5的稳定性,从而减轻了干旱诱导的氧化损伤,增强了玉米对干旱胁迫的响应[34]. ...
The Arabidopsis SUMO E3 ligase SIZ1 mediates the temperature dependent trade-off between plant immunity and growth
2
2018
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
SUMOylome profiling reveals a diverse array of nuclear targets modified by the SUMO ligase SIZ1 during heat stress
1
2018
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
SUMO E3 ligase SlSIZ1 facilitates heat tolerance in tomato
1
2018
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
SIZ1-mediated SUMOylation of CPSF100 promotes plant thermomorphogenesis by controlling alternative polyadenylation
1
2024
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
Importation of chloroplast proteins under heat stress is facilitated by their SUMO conjugations
1
2022
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
SUMOylation facilitates the assembly of a Nuclear Factor-Y complex to enhance thermotolerance in Arabidopsis
1
2023
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
E3 SUMO ligase SIZ1 splicing variants localize and function according to external conditions
1
2024
... SIZ1通过SUMO化修饰动态调控植物的基础耐热性(basal thermotolerance),即植物在未经过高温驯化的情况下,天然具备的抵抗高温胁迫的能力.拟南芥siz1突变体表现出热敏感表型,植物体内SUMO1/2结合蛋白的积累水平显著下降,并伴随SA含量的异常升高;为明确SIZ1是否通过SA途径发挥功能,研究人员在siz1背景中导入水杨酸羟化酶NahG以降解内源SA,反而导致植株热敏感性增强,表明SIZ1通过SA非依赖途径调控耐热性;siz1中HSP101、HSP70等热激蛋白的表达未受影响,表明SIZ1可能通过SUMO化修饰热休克转录因子HSF以外的底物(如核定位蛋白)调控耐热性[21].拟南芥SIZ1通过SUMO化COP1泛素连接酶促进其对HY5的降解,从而激活PIF4/BZR1驱动热形态建成,siz1突变体中该信号轴功能受损,表现为热诱导基因表达延迟及生长恢复受阻[35].进一步研究发现,SIZ1在参与植物高温胁迫应答中介导的SUMO化修饰具有多重调控层次:1)底物选择性:主要靶向核定位蛋白,包括热响应转录因子(HSFB2b、DREB2A)、共激活因子(TPL、DRIP2)及染色质重塑复合物成员(SWI3C、SWI3D、CHR11、CHR17和PICKLE),SIZ1蛋白本身也是SUMO化的靶标[36].番茄SlSIZ1通过SUMO化修饰SlHsfA1,协同促进Hsp70等热激蛋白表达,从而增强番茄的耐热性[37].SIZ1通过介导CPSF复合物调控多聚腺苷酸化的关键亚基CPSF100的SUMO化修饰,促进其在细胞核中积累,干扰其与CPSF30-M/L的互作,进而调控可变加尾事件,影响PIF4、YUC8、ELF3等温度响应基因的表达[38].2)亚细胞调控:AtSIZ1通过SUMO化维持PSBR等核编码叶绿体蛋白的正确定位,保障高温下光合系统功能的发挥[39].3)信号整合:拟南芥SIZ1在高温胁迫下促进NF-YC10与NF-YB3复合体组装及核易位,激活HSFA3等热响应基因,从而增强植株的耐热性[40].4)剪接变体调控:拟南芥SIZ1的4个剪接变异体(SIZ1 splicing variant, SSV)编码3种不同的蛋白质亚型,SIZ1和所有SSV表现出相似的SUMO E3连接酶活性,热处理使SSV2的转录水平显著提高,SSV2通过SUMO化修饰质膜定位的CNGC6离子通道增强其稳定性,从而增强植株对高温信号的感知[41]. ...
SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in Arabidopsis
1
2007
... SIZ1在植物低温胁迫响应中具有关键的调控作用.拟南芥AtSIZ1通过介导ICE1转录因子的SUMO化修饰显著增强其蛋白稳定性及转录活性,进而激活CBF3/DREB1A信号通路,最终促进冷响应基因表达并提高植物抗冻性;SUMO化的ICE1可形成双重调控机制:一方面通过抑制MYB15转录因子的活性解除其对CBF通路的负调控,另一方面阻断泛素E3泛素连接酶HOS1介导的ICE1泛素化降解,从而维持低温信号通路的持续激活[42].进一步研究发现,SIZ1通过调控SA代谢途径影响拟南芥耐寒性,siz1突变体中SA异常积累,且CBF3/DREB1A及其下游基因表达下降,对低温和冷冻条件的敏感性提高,而降低SA水平可部分恢复其低温耐受性,证实SA信号与SIZ1途径在拟南芥对冷胁迫的响应中存在拮抗作用[43].前人在拟南芥中研究发现,泛素E3连接酶COP1与AtSIZ1相互作用,并通过多泛素化修饰促进其降解,当COP1活性被抑制或缺失时,AtSIZ1蛋白水平显著增加,从而导致植株中蛋白SUMO化水平升高,这种调控机制使得植物对低温、干旱和高盐等逆境条件表现出更强的耐受性;此外,AtSIZ1还可能通过SUMO化修饰反向调控COP1的活性[44],这一发现揭示了泛素化和SUMO化系统之间的交叉调控机制.苹果MdSIZ1通过SUMO化修饰稳定MdMYB1转录因子,促进低温诱导的花青素生物合成[22],MdMYB2作为上游调控因子,可直接结合MdSIZ1启动子并增强其转录活性,从而形成MdMYB2-MdSIZ1-MdMYB1级联调控网络[45].拟南芥HPY2通过对细胞分裂素响应因子ARR1的K236位点进行SUMO化修饰激活其转录活性,在低温或细胞分裂素处理下ARR1 D89位点的磷酸化介导其与HPY2结合,SUMO化的ARR1进一步促进组蛋白H3乙酰化,增强靶基因表达,参与细胞分裂素与低温信号的整合调控[46]. ...
SIZ1, a small ubiquitin-related modifier ligase, controls cold signaling through regulation of salicylic acid accumulation
1
2010
... SIZ1在植物低温胁迫响应中具有关键的调控作用.拟南芥AtSIZ1通过介导ICE1转录因子的SUMO化修饰显著增强其蛋白稳定性及转录活性,进而激活CBF3/DREB1A信号通路,最终促进冷响应基因表达并提高植物抗冻性;SUMO化的ICE1可形成双重调控机制:一方面通过抑制MYB15转录因子的活性解除其对CBF通路的负调控,另一方面阻断泛素E3泛素连接酶HOS1介导的ICE1泛素化降解,从而维持低温信号通路的持续激活[42].进一步研究发现,SIZ1通过调控SA代谢途径影响拟南芥耐寒性,siz1突变体中SA异常积累,且CBF3/DREB1A及其下游基因表达下降,对低温和冷冻条件的敏感性提高,而降低SA水平可部分恢复其低温耐受性,证实SA信号与SIZ1途径在拟南芥对冷胁迫的响应中存在拮抗作用[43].前人在拟南芥中研究发现,泛素E3连接酶COP1与AtSIZ1相互作用,并通过多泛素化修饰促进其降解,当COP1活性被抑制或缺失时,AtSIZ1蛋白水平显著增加,从而导致植株中蛋白SUMO化水平升高,这种调控机制使得植物对低温、干旱和高盐等逆境条件表现出更强的耐受性;此外,AtSIZ1还可能通过SUMO化修饰反向调控COP1的活性[44],这一发现揭示了泛素化和SUMO化系统之间的交叉调控机制.苹果MdSIZ1通过SUMO化修饰稳定MdMYB1转录因子,促进低温诱导的花青素生物合成[22],MdMYB2作为上游调控因子,可直接结合MdSIZ1启动子并增强其转录活性,从而形成MdMYB2-MdSIZ1-MdMYB1级联调控网络[45].拟南芥HPY2通过对细胞分裂素响应因子ARR1的K236位点进行SUMO化修饰激活其转录活性,在低温或细胞分裂素处理下ARR1 D89位点的磷酸化介导其与HPY2结合,SUMO化的ARR1进一步促进组蛋白H3乙酰化,增强靶基因表达,参与细胞分裂素与低温信号的整合调控[46]. ...
COP1 controls abiotic stress responses by modulating AtSIZ1 function through its E3 ubiquitin ligase activity
1
2016
... SIZ1在植物低温胁迫响应中具有关键的调控作用.拟南芥AtSIZ1通过介导ICE1转录因子的SUMO化修饰显著增强其蛋白稳定性及转录活性,进而激活CBF3/DREB1A信号通路,最终促进冷响应基因表达并提高植物抗冻性;SUMO化的ICE1可形成双重调控机制:一方面通过抑制MYB15转录因子的活性解除其对CBF通路的负调控,另一方面阻断泛素E3泛素连接酶HOS1介导的ICE1泛素化降解,从而维持低温信号通路的持续激活[42].进一步研究发现,SIZ1通过调控SA代谢途径影响拟南芥耐寒性,siz1突变体中SA异常积累,且CBF3/DREB1A及其下游基因表达下降,对低温和冷冻条件的敏感性提高,而降低SA水平可部分恢复其低温耐受性,证实SA信号与SIZ1途径在拟南芥对冷胁迫的响应中存在拮抗作用[43].前人在拟南芥中研究发现,泛素E3连接酶COP1与AtSIZ1相互作用,并通过多泛素化修饰促进其降解,当COP1活性被抑制或缺失时,AtSIZ1蛋白水平显著增加,从而导致植株中蛋白SUMO化水平升高,这种调控机制使得植物对低温、干旱和高盐等逆境条件表现出更强的耐受性;此外,AtSIZ1还可能通过SUMO化修饰反向调控COP1的活性[44],这一发现揭示了泛素化和SUMO化系统之间的交叉调控机制.苹果MdSIZ1通过SUMO化修饰稳定MdMYB1转录因子,促进低温诱导的花青素生物合成[22],MdMYB2作为上游调控因子,可直接结合MdSIZ1启动子并增强其转录活性,从而形成MdMYB2-MdSIZ1-MdMYB1级联调控网络[45].拟南芥HPY2通过对细胞分裂素响应因子ARR1的K236位点进行SUMO化修饰激活其转录活性,在低温或细胞分裂素处理下ARR1 D89位点的磷酸化介导其与HPY2结合,SUMO化的ARR1进一步促进组蛋白H3乙酰化,增强靶基因表达,参与细胞分裂素与低温信号的整合调控[46]. ...
The transcription factor MdMYB2 influences cold tolerance and anthocyanin accumulation by activating SUMO E3 ligase MdSIZ1 in apple
1
2022
... SIZ1在植物低温胁迫响应中具有关键的调控作用.拟南芥AtSIZ1通过介导ICE1转录因子的SUMO化修饰显著增强其蛋白稳定性及转录活性,进而激活CBF3/DREB1A信号通路,最终促进冷响应基因表达并提高植物抗冻性;SUMO化的ICE1可形成双重调控机制:一方面通过抑制MYB15转录因子的活性解除其对CBF通路的负调控,另一方面阻断泛素E3泛素连接酶HOS1介导的ICE1泛素化降解,从而维持低温信号通路的持续激活[42].进一步研究发现,SIZ1通过调控SA代谢途径影响拟南芥耐寒性,siz1突变体中SA异常积累,且CBF3/DREB1A及其下游基因表达下降,对低温和冷冻条件的敏感性提高,而降低SA水平可部分恢复其低温耐受性,证实SA信号与SIZ1途径在拟南芥对冷胁迫的响应中存在拮抗作用[43].前人在拟南芥中研究发现,泛素E3连接酶COP1与AtSIZ1相互作用,并通过多泛素化修饰促进其降解,当COP1活性被抑制或缺失时,AtSIZ1蛋白水平显著增加,从而导致植株中蛋白SUMO化水平升高,这种调控机制使得植物对低温、干旱和高盐等逆境条件表现出更强的耐受性;此外,AtSIZ1还可能通过SUMO化修饰反向调控COP1的活性[44],这一发现揭示了泛素化和SUMO化系统之间的交叉调控机制.苹果MdSIZ1通过SUMO化修饰稳定MdMYB1转录因子,促进低温诱导的花青素生物合成[22],MdMYB2作为上游调控因子,可直接结合MdSIZ1启动子并增强其转录活性,从而形成MdMYB2-MdSIZ1-MdMYB1级联调控网络[45].拟南芥HPY2通过对细胞分裂素响应因子ARR1的K236位点进行SUMO化修饰激活其转录活性,在低温或细胞分裂素处理下ARR1 D89位点的磷酸化介导其与HPY2结合,SUMO化的ARR1进一步促进组蛋白H3乙酰化,增强靶基因表达,参与细胞分裂素与低温信号的整合调控[46]. ...
HIGH PLOIDY2-mediated SUMOylation of transcription factor ARR1 controls two-component signaling in Arabidopsis
1
2024
... SIZ1在植物低温胁迫响应中具有关键的调控作用.拟南芥AtSIZ1通过介导ICE1转录因子的SUMO化修饰显著增强其蛋白稳定性及转录活性,进而激活CBF3/DREB1A信号通路,最终促进冷响应基因表达并提高植物抗冻性;SUMO化的ICE1可形成双重调控机制:一方面通过抑制MYB15转录因子的活性解除其对CBF通路的负调控,另一方面阻断泛素E3泛素连接酶HOS1介导的ICE1泛素化降解,从而维持低温信号通路的持续激活[42].进一步研究发现,SIZ1通过调控SA代谢途径影响拟南芥耐寒性,siz1突变体中SA异常积累,且CBF3/DREB1A及其下游基因表达下降,对低温和冷冻条件的敏感性提高,而降低SA水平可部分恢复其低温耐受性,证实SA信号与SIZ1途径在拟南芥对冷胁迫的响应中存在拮抗作用[43].前人在拟南芥中研究发现,泛素E3连接酶COP1与AtSIZ1相互作用,并通过多泛素化修饰促进其降解,当COP1活性被抑制或缺失时,AtSIZ1蛋白水平显著增加,从而导致植株中蛋白SUMO化水平升高,这种调控机制使得植物对低温、干旱和高盐等逆境条件表现出更强的耐受性;此外,AtSIZ1还可能通过SUMO化修饰反向调控COP1的活性[44],这一发现揭示了泛素化和SUMO化系统之间的交叉调控机制.苹果MdSIZ1通过SUMO化修饰稳定MdMYB1转录因子,促进低温诱导的花青素生物合成[22],MdMYB2作为上游调控因子,可直接结合MdSIZ1启动子并增强其转录活性,从而形成MdMYB2-MdSIZ1-MdMYB1级联调控网络[45].拟南芥HPY2通过对细胞分裂素响应因子ARR1的K236位点进行SUMO化修饰激活其转录活性,在低温或细胞分裂素处理下ARR1 D89位点的磷酸化介导其与HPY2结合,SUMO化的ARR1进一步促进组蛋白H3乙酰化,增强靶基因表达,参与细胞分裂素与低温信号的整合调控[46]. ...
SIZ1-mediated SUMOylation during phosphate homeostasis in plants: Looking beyond the tip of the iceberg
1
2018
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
SIZ1 regulation of phosphate starvation-induced root architecture remodeling involves the control of auxin accumulation
1
2011
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
OsSIZ2 exerts regulatory influences on the developmental responses and phosphate homeostasis in rice
1
2017
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
OsSIZ1, a SUMO E3 ligase gene, is involved in the regulation of the responses to phosphate and nitrogen in rice
1
2015
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
Apple SUMO E3 ligase MdSIZ1 is involved in the response to phosphate deficiency
1
2019
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
The SUMO E3 ligase SIZ1 partially regulates STOP1 SUMOylation and stability in Arabidopsis thaliana
2
2021
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
... 在拟南芥中,ALMT1编码一个苹果酸转运子,SIZ1通过STOP1-ALMT1信号枢纽在铝/铁胁迫响应中发挥双向调控功能:一方面,SIZ1通过对STOP1进行SUMO化修饰降低其稳定性,抑制ALMT1介导的苹果酸分泌,从而负调控拟南芥对铝的耐受性;另一方面,铝胁迫通过诱导SIZ1降解形成负反馈回路,精细调控解毒反应[52-53].研究发现,突变体siz1在其根质外体中过度积累铁导致羟自由基(OH•)水平升高,通过STOP1-ALMT1通路加剧根系生长抑制[66].苹果SIZ1通过SUMO化修饰MdbHLH104增强H+-ATPase活性,揭示了SIZ1介导的SUMO化机制在铁吸收调控中具有物种特异性[67]. ...
SIZ1 negatively regulates aluminum resistance by mediating the STOP1-ALMT1 pathway in Arabidopsis
2
2021
... 磷元素对于植物正常生长不可或缺.SIZ1及其同源物调节磷的吸收、转运及根系形态建成等多个生理过程,其在植物磷适应性调控中的作用已被广泛研究[47].拟南芥AtSIZ1通过SUMO化修饰磷信号核心调控因子AtPHR1,抑制AtIPS1、AtRNS1等磷饥饿响应基因的过度激活,其siz1-2突变体在磷充足的条件下仍表现出主根生长受抑、侧根增生及花青素积累等典型的缺磷表型[15].进一步研究表明,siz1突变体在低磷条件下表现出更显著的主根生长抑制和侧根发育增加,并伴随生长素响应基因TIR1和ARF19的表达上调,表明SIZ1可能通过调控生长素的分布模式介导低磷诱导的根系重塑过程[48].水稻OsSIZ1及其同源基因OsSIZ2均参与磷稳态调控,ossiz1突变体在磷充足和磷缺乏条件下均表现出磷积累增加,并影响多个磷饥饿信号通路相关基因的表达,而ossiz2突变体在缺磷时表现出组织磷含量降低,表明二者在磷信号调控中具有不同作用[49-50].苹果MdSIZ1表达受缺磷胁迫诱导,并通过促进SUMO化调节磷饥饿响应基因的表达,过表达MdSIZ1可部分恢复拟南芥siz1-2突变体的缺陷,并改善转基因苹果愈伤组织在缺磷条件下的生长[51].拟南芥STOP1是调控低磷适应性的关键因子,SIZ1介导STOP1在K40和K212位点的SUMO化修饰,从而抑制其DNA结合能力,导致AtALMT1的转录水平下降,siz1突变体中AtALMT1表达上调、苹果酸分泌增加,从而提高根际磷的可利用性,进而增强了植物对低磷环境的适应性[52-53]. ...
... 在拟南芥中,ALMT1编码一个苹果酸转运子,SIZ1通过STOP1-ALMT1信号枢纽在铝/铁胁迫响应中发挥双向调控功能:一方面,SIZ1通过对STOP1进行SUMO化修饰降低其稳定性,抑制ALMT1介导的苹果酸分泌,从而负调控拟南芥对铝的耐受性;另一方面,铝胁迫通过诱导SIZ1降解形成负反馈回路,精细调控解毒反应[52-53].研究发现,突变体siz1在其根质外体中过度积累铁导致羟自由基(OH•)水平升高,通过STOP1-ALMT1通路加剧根系生长抑制[66].苹果SIZ1通过SUMO化修饰MdbHLH104增强H+-ATPase活性,揭示了SIZ1介导的SUMO化机制在铁吸收调控中具有物种特异性[67]. ...
Arabidopsis nitrate reductase activity is stimulated by the E3 SUMO ligase AtSIZ1
2
2011
... 氮元素是植物正常生长发育必不可少的营养元素之一.在拟南芥中,AtSIZ1通过SUMO化修饰硝酸还原酶NR(NIA1/NIA2),显著增强其催化硝酸盐(NO3-)向亚硝酸盐(NO2-)的转化效率,直接调控植株氮素积累与生长发育[54].siz1-2突变体中NR活性降低、硝酸盐异常积累、氮含量下降,而外施铵(NH4+)态氮可部分恢复其表型,证实了SUMO化修饰对氮源利用的选择性调控[54-55].进一步研究发现,拟南芥siz1-2突变体中线粒体交替呼吸途径基因NDA2、AOX1a的表达受到抑制,导致碳氮比失衡及自由糖含量降低,说明SUMO化通过协调氮同化与呼吸代谢维持植株体内氮稳态[56].此外,AtSIZ1还通过表观遗传机制调控氮代谢基因的表达,AtSIZ1通过介导CMT3的SUMO化增强其对目标基因CHG位点的甲基化能力,特异性抑制NIA2甲基化水平而不影响NIA1[57].AtSIZ1还能调控NR的核质转运,使NR从胞质膜重定位至细胞核,该过程受到氮源的影响,硝酸盐促进AtSIZ1从细胞核向细胞质转移,但不影响NR的亚细胞定位,而铵盐处理则降低NR蛋白水平,不改变细胞中AtSIZ1的定位;此外,COP1介导的泛素化与AtSIZ1介导的SUMO化相拮抗,其通过26S蛋白酶体途径调控NIA1/NIA2稳定性,而铵盐处理通过未知E3连接酶加速其降解[55]. ...
... [54-55].进一步研究发现,拟南芥siz1-2突变体中线粒体交替呼吸途径基因NDA2、AOX1a的表达受到抑制,导致碳氮比失衡及自由糖含量降低,说明SUMO化通过协调氮同化与呼吸代谢维持植株体内氮稳态[56].此外,AtSIZ1还通过表观遗传机制调控氮代谢基因的表达,AtSIZ1通过介导CMT3的SUMO化增强其对目标基因CHG位点的甲基化能力,特异性抑制NIA2甲基化水平而不影响NIA1[57].AtSIZ1还能调控NR的核质转运,使NR从胞质膜重定位至细胞核,该过程受到氮源的影响,硝酸盐促进AtSIZ1从细胞核向细胞质转移,但不影响NR的亚细胞定位,而铵盐处理则降低NR蛋白水平,不改变细胞中AtSIZ1的定位;此外,COP1介导的泛素化与AtSIZ1介导的SUMO化相拮抗,其通过26S蛋白酶体途径调控NIA1/NIA2稳定性,而铵盐处理通过未知E3连接酶加速其降解[55]. ...
Nitrate reductases are relocalized to the nucleus by AtSIZ1 and their levels are negatively regulated by COP1 and ammonium
2
2018
... 氮元素是植物正常生长发育必不可少的营养元素之一.在拟南芥中,AtSIZ1通过SUMO化修饰硝酸还原酶NR(NIA1/NIA2),显著增强其催化硝酸盐(NO3-)向亚硝酸盐(NO2-)的转化效率,直接调控植株氮素积累与生长发育[54].siz1-2突变体中NR活性降低、硝酸盐异常积累、氮含量下降,而外施铵(NH4+)态氮可部分恢复其表型,证实了SUMO化修饰对氮源利用的选择性调控[54-55].进一步研究发现,拟南芥siz1-2突变体中线粒体交替呼吸途径基因NDA2、AOX1a的表达受到抑制,导致碳氮比失衡及自由糖含量降低,说明SUMO化通过协调氮同化与呼吸代谢维持植株体内氮稳态[56].此外,AtSIZ1还通过表观遗传机制调控氮代谢基因的表达,AtSIZ1通过介导CMT3的SUMO化增强其对目标基因CHG位点的甲基化能力,特异性抑制NIA2甲基化水平而不影响NIA1[57].AtSIZ1还能调控NR的核质转运,使NR从胞质膜重定位至细胞核,该过程受到氮源的影响,硝酸盐促进AtSIZ1从细胞核向细胞质转移,但不影响NR的亚细胞定位,而铵盐处理则降低NR蛋白水平,不改变细胞中AtSIZ1的定位;此外,COP1介导的泛素化与AtSIZ1介导的SUMO化相拮抗,其通过26S蛋白酶体途径调控NIA1/NIA2稳定性,而铵盐处理通过未知E3连接酶加速其降解[55]. ...
... [55]. ...
Arabidopsis SIZ1 positively regulates alternative respiratory bypass pathways
1
2012
... 氮元素是植物正常生长发育必不可少的营养元素之一.在拟南芥中,AtSIZ1通过SUMO化修饰硝酸还原酶NR(NIA1/NIA2),显著增强其催化硝酸盐(NO3-)向亚硝酸盐(NO2-)的转化效率,直接调控植株氮素积累与生长发育[54].siz1-2突变体中NR活性降低、硝酸盐异常积累、氮含量下降,而外施铵(NH4+)态氮可部分恢复其表型,证实了SUMO化修饰对氮源利用的选择性调控[54-55].进一步研究发现,拟南芥siz1-2突变体中线粒体交替呼吸途径基因NDA2、AOX1a的表达受到抑制,导致碳氮比失衡及自由糖含量降低,说明SUMO化通过协调氮同化与呼吸代谢维持植株体内氮稳态[56].此外,AtSIZ1还通过表观遗传机制调控氮代谢基因的表达,AtSIZ1通过介导CMT3的SUMO化增强其对目标基因CHG位点的甲基化能力,特异性抑制NIA2甲基化水平而不影响NIA1[57].AtSIZ1还能调控NR的核质转运,使NR从胞质膜重定位至细胞核,该过程受到氮源的影响,硝酸盐促进AtSIZ1从细胞核向细胞质转移,但不影响NR的亚细胞定位,而铵盐处理则降低NR蛋白水平,不改变细胞中AtSIZ1的定位;此外,COP1介导的泛素化与AtSIZ1介导的SUMO化相拮抗,其通过26S蛋白酶体途径调控NIA1/NIA2稳定性,而铵盐处理通过未知E3连接酶加速其降解[55]. ...
Arabidopsis CMT3 activity is positively regulated by AtSIZ1-mediated sumoylation
1
2015
... 氮元素是植物正常生长发育必不可少的营养元素之一.在拟南芥中,AtSIZ1通过SUMO化修饰硝酸还原酶NR(NIA1/NIA2),显著增强其催化硝酸盐(NO3-)向亚硝酸盐(NO2-)的转化效率,直接调控植株氮素积累与生长发育[54].siz1-2突变体中NR活性降低、硝酸盐异常积累、氮含量下降,而外施铵(NH4+)态氮可部分恢复其表型,证实了SUMO化修饰对氮源利用的选择性调控[54-55].进一步研究发现,拟南芥siz1-2突变体中线粒体交替呼吸途径基因NDA2、AOX1a的表达受到抑制,导致碳氮比失衡及自由糖含量降低,说明SUMO化通过协调氮同化与呼吸代谢维持植株体内氮稳态[56].此外,AtSIZ1还通过表观遗传机制调控氮代谢基因的表达,AtSIZ1通过介导CMT3的SUMO化增强其对目标基因CHG位点的甲基化能力,特异性抑制NIA2甲基化水平而不影响NIA1[57].AtSIZ1还能调控NR的核质转运,使NR从胞质膜重定位至细胞核,该过程受到氮源的影响,硝酸盐促进AtSIZ1从细胞核向细胞质转移,但不影响NR的亚细胞定位,而铵盐处理则降低NR蛋白水平,不改变细胞中AtSIZ1的定位;此外,COP1介导的泛素化与AtSIZ1介导的SUMO化相拮抗,其通过26S蛋白酶体途径调控NIA1/NIA2稳定性,而铵盐处理通过未知E3连接酶加速其降解[55]. ...
SIZ1-dependent post-translational modification by SUMO modulates sugar signaling and metabolism in Arabidopsis thaliana
1
2015
... 糖不仅能提供植物生长发育中所需的能量,同时也作为信号分子发挥关键作用.SIZ1通过动态调控糖代谢网络参与植物糖信号响应.拟南芥SIZ1介导的SUMO化修饰在高糖条件下显著增强,siz1突变体中蔗糖/淀粉降解基因表达上调,表现出淀粉含量降低及胚后生长停滞(prolonged growth gap arrest, PGGA)等典型糖敏感表型,SIZ1通过抑制糖代谢基因负调控淀粉积累,且可能通过稳定SnRK1激酶AKIN10影响糖信号转导[58].进一步研究发现,SIZ1直接参与调控糖代谢,siz1突变体的萌发延迟表型部分依赖于SA积累,但其PGGA与根毛发育异常等糖响应特征则独立于SA信号[59].拟南芥Hexokinase 1(HXK1)与SIZ1可能通过不同途径调控糖代谢从而影响植株生长,siz1hxk1双突变体表现出叠加的生长缺陷,但HXK1与SIZ1或其他SUMO连接酶的互作关系尚未明确[60].最新研究揭示,HXK1与SIZ1在控制拟南芥对高糖条件的响应方面存在拮抗作用和上位性:hxk1siz1双突变可逆转siz1的萌发抑制表型,说明HXK1可能通过抑制SIZ1下游通路或形成遗传上位性调控网络发挥功能[61]. ...
Sugar signaling regulation by Arabidopsis SIZ1-driven sumoylation is independent of salicylic acid
1
2018
... 糖不仅能提供植物生长发育中所需的能量,同时也作为信号分子发挥关键作用.SIZ1通过动态调控糖代谢网络参与植物糖信号响应.拟南芥SIZ1介导的SUMO化修饰在高糖条件下显著增强,siz1突变体中蔗糖/淀粉降解基因表达上调,表现出淀粉含量降低及胚后生长停滞(prolonged growth gap arrest, PGGA)等典型糖敏感表型,SIZ1通过抑制糖代谢基因负调控淀粉积累,且可能通过稳定SnRK1激酶AKIN10影响糖信号转导[58].进一步研究发现,SIZ1直接参与调控糖代谢,siz1突变体的萌发延迟表型部分依赖于SA积累,但其PGGA与根毛发育异常等糖响应特征则独立于SA信号[59].拟南芥Hexokinase 1(HXK1)与SIZ1可能通过不同途径调控糖代谢从而影响植株生长,siz1hxk1双突变体表现出叠加的生长缺陷,但HXK1与SIZ1或其他SUMO连接酶的互作关系尚未明确[60].最新研究揭示,HXK1与SIZ1在控制拟南芥对高糖条件的响应方面存在拮抗作用和上位性:hxk1siz1双突变可逆转siz1的萌发抑制表型,说明HXK1可能通过抑制SIZ1下游通路或形成遗传上位性调控网络发挥功能[61]. ...
Arabidopsis thaliana growth is independently controlled by the SUMO E3 ligase SIZ1 and Hexokinase 1
1
2020
... 糖不仅能提供植物生长发育中所需的能量,同时也作为信号分子发挥关键作用.SIZ1通过动态调控糖代谢网络参与植物糖信号响应.拟南芥SIZ1介导的SUMO化修饰在高糖条件下显著增强,siz1突变体中蔗糖/淀粉降解基因表达上调,表现出淀粉含量降低及胚后生长停滞(prolonged growth gap arrest, PGGA)等典型糖敏感表型,SIZ1通过抑制糖代谢基因负调控淀粉积累,且可能通过稳定SnRK1激酶AKIN10影响糖信号转导[58].进一步研究发现,SIZ1直接参与调控糖代谢,siz1突变体的萌发延迟表型部分依赖于SA积累,但其PGGA与根毛发育异常等糖响应特征则独立于SA信号[59].拟南芥Hexokinase 1(HXK1)与SIZ1可能通过不同途径调控糖代谢从而影响植株生长,siz1hxk1双突变体表现出叠加的生长缺陷,但HXK1与SIZ1或其他SUMO连接酶的互作关系尚未明确[60].最新研究揭示,HXK1与SIZ1在控制拟南芥对高糖条件的响应方面存在拮抗作用和上位性:hxk1siz1双突变可逆转siz1的萌发抑制表型,说明HXK1可能通过抑制SIZ1下游通路或形成遗传上位性调控网络发挥功能[61]. ...
Complex genetic interaction between glucose sensor HXK1 and E3 SUMO ligase SIZ1 in regulating plant morphogenesis
1
2024
... 糖不仅能提供植物生长发育中所需的能量,同时也作为信号分子发挥关键作用.SIZ1通过动态调控糖代谢网络参与植物糖信号响应.拟南芥SIZ1介导的SUMO化修饰在高糖条件下显著增强,siz1突变体中蔗糖/淀粉降解基因表达上调,表现出淀粉含量降低及胚后生长停滞(prolonged growth gap arrest, PGGA)等典型糖敏感表型,SIZ1通过抑制糖代谢基因负调控淀粉积累,且可能通过稳定SnRK1激酶AKIN10影响糖信号转导[58].进一步研究发现,SIZ1直接参与调控糖代谢,siz1突变体的萌发延迟表型部分依赖于SA积累,但其PGGA与根毛发育异常等糖响应特征则独立于SA信号[59].拟南芥Hexokinase 1(HXK1)与SIZ1可能通过不同途径调控糖代谢从而影响植株生长,siz1hxk1双突变体表现出叠加的生长缺陷,但HXK1与SIZ1或其他SUMO连接酶的互作关系尚未明确[60].最新研究揭示,HXK1与SIZ1在控制拟南芥对高糖条件的响应方面存在拮抗作用和上位性:hxk1siz1双突变可逆转siz1的萌发抑制表型,说明HXK1可能通过抑制SIZ1下游通路或形成遗传上位性调控网络发挥功能[61]. ...
Arabidopsis SUMO E3 ligase SIZ1 is involved in excess copper tolerance
2
2011
... SUMO E3连接酶SIZ1通过动态调控金属转运与解毒网络介导植物对重金属胁迫的响应.在拟南芥中,SIZ1介导的SUMO化修饰可抑制YellowStripe-like1/3(YSL1/YSL3)的表达,从而限制铜在植株地上部的异常积累[62];进一步研究发现,SIZ1通过稳定Cu/Zn超氧化物歧化酶CSD1的蛋白水平,缓解铜诱导的氧化损伤[63],同时通过调控SA信号通路抑制YSL3的过度表达,突变体siz1因SA过量积累而表现出对铜敏感加剧[64].去SUMO化酶OTS1与SIZ1形成协同调控轴,ots1突变体中SUMO化蛋白异常积累导致YSL3表达失调,证实了SUMO修饰动态平衡在维持铜稳态中的核心作用[65]. ...
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
Effect of Cu content on the activity of Cu/ZnSOD1 in the Arabidopsis SUMO E3 ligase siz1 mutant
1
2011
... SUMO E3连接酶SIZ1通过动态调控金属转运与解毒网络介导植物对重金属胁迫的响应.在拟南芥中,SIZ1介导的SUMO化修饰可抑制YellowStripe-like1/3(YSL1/YSL3)的表达,从而限制铜在植株地上部的异常积累[62];进一步研究发现,SIZ1通过稳定Cu/Zn超氧化物歧化酶CSD1的蛋白水平,缓解铜诱导的氧化损伤[63],同时通过调控SA信号通路抑制YSL3的过度表达,突变体siz1因SA过量积累而表现出对铜敏感加剧[64].去SUMO化酶OTS1与SIZ1形成协同调控轴,ots1突变体中SUMO化蛋白异常积累导致YSL3表达失调,证实了SUMO修饰动态平衡在维持铜稳态中的核心作用[65]. ...
Alternative functions of Arabidopsis Yellow STRIPE-LIKE3: from metal translocation to pathogen defense
2
2014
... SUMO E3连接酶SIZ1通过动态调控金属转运与解毒网络介导植物对重金属胁迫的响应.在拟南芥中,SIZ1介导的SUMO化修饰可抑制YellowStripe-like1/3(YSL1/YSL3)的表达,从而限制铜在植株地上部的异常积累[62];进一步研究发现,SIZ1通过稳定Cu/Zn超氧化物歧化酶CSD1的蛋白水平,缓解铜诱导的氧化损伤[63],同时通过调控SA信号通路抑制YSL3的过度表达,突变体siz1因SA过量积累而表现出对铜敏感加剧[64].去SUMO化酶OTS1与SIZ1形成协同调控轴,ots1突变体中SUMO化蛋白异常积累导致YSL3表达失调,证实了SUMO修饰动态平衡在维持铜稳态中的核心作用[65]. ...
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
OTS1-dependent deSUMOylation increases tolerance to high copper levels in Arabidopsis
1
2018
... SUMO E3连接酶SIZ1通过动态调控金属转运与解毒网络介导植物对重金属胁迫的响应.在拟南芥中,SIZ1介导的SUMO化修饰可抑制YellowStripe-like1/3(YSL1/YSL3)的表达,从而限制铜在植株地上部的异常积累[62];进一步研究发现,SIZ1通过稳定Cu/Zn超氧化物歧化酶CSD1的蛋白水平,缓解铜诱导的氧化损伤[63],同时通过调控SA信号通路抑制YSL3的过度表达,突变体siz1因SA过量积累而表现出对铜敏感加剧[64].去SUMO化酶OTS1与SIZ1形成协同调控轴,ots1突变体中SUMO化蛋白异常积累导致YSL3表达失调,证实了SUMO修饰动态平衡在维持铜稳态中的核心作用[65]. ...
SIZ1 regulates phosphate deficiency-induced inhibition of primary root growth of Arabidopsis by modulating Fe accumulation and ROS production in its roots
1
2021
... 在拟南芥中,ALMT1编码一个苹果酸转运子,SIZ1通过STOP1-ALMT1信号枢纽在铝/铁胁迫响应中发挥双向调控功能:一方面,SIZ1通过对STOP1进行SUMO化修饰降低其稳定性,抑制ALMT1介导的苹果酸分泌,从而负调控拟南芥对铝的耐受性;另一方面,铝胁迫通过诱导SIZ1降解形成负反馈回路,精细调控解毒反应[52-53].研究发现,突变体siz1在其根质外体中过度积累铁导致羟自由基(OH•)水平升高,通过STOP1-ALMT1通路加剧根系生长抑制[66].苹果SIZ1通过SUMO化修饰MdbHLH104增强H+-ATPase活性,揭示了SIZ1介导的SUMO化机制在铁吸收调控中具有物种特异性[67]. ...
The SUMO E3 ligase MdSIZ1 targets MdbHLH104 to regulate plasma membrane H+-ATPase activity and iron homeostasis
1
2019
... 在拟南芥中,ALMT1编码一个苹果酸转运子,SIZ1通过STOP1-ALMT1信号枢纽在铝/铁胁迫响应中发挥双向调控功能:一方面,SIZ1通过对STOP1进行SUMO化修饰降低其稳定性,抑制ALMT1介导的苹果酸分泌,从而负调控拟南芥对铝的耐受性;另一方面,铝胁迫通过诱导SIZ1降解形成负反馈回路,精细调控解毒反应[52-53].研究发现,突变体siz1在其根质外体中过度积累铁导致羟自由基(OH•)水平升高,通过STOP1-ALMT1通路加剧根系生长抑制[66].苹果SIZ1通过SUMO化修饰MdbHLH104增强H+-ATPase活性,揭示了SIZ1介导的SUMO化机制在铁吸收调控中具有物种特异性[67]. ...
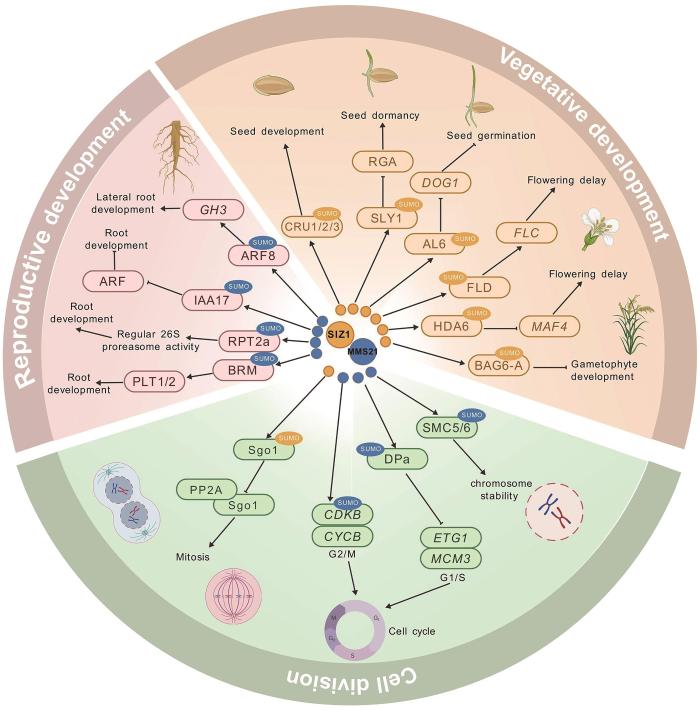
Sumoylation of ABI5 by the Arabidopsis SUMO E3 ligase SIZ1 negatively regulates abscisic acid signaling
1
2009
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
A SUMO ligase AtMMS21 regulates the stability of the chromatin remodeler BRAHMA in root development
1
2017
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
A SUMO ligase AtMMS21 regulates activity of the 26S proteasome in root development
1
2019
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
Apple SUMO E3 ligase MdSIZ1 facilitates SUMOylation of MdARF8 to regulate lateral root formation
1
2021
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
A SUMO ligase OsMMS21 regulates rice development and auxin response
2
2021
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
... SUMO E3连接酶介导的PTM在植物逆境响应和生长发育的多个过程中发挥重要作用,因此被视为改良作物性状的新途径.SUMO E3连接酶的功能鉴定及相关机制的研究突破为利用SUMO通路对作物进行遗传改良提供了新的切入点.育种家们可以将SUMO通路相关基因作为育种或基因编辑的目标,例如,通过基因工程适度上调关键的SUMO E3连接酶(如提高水稻OsSIZ1或OsMMS21的表达)[25, 72],有望增强作物对干旱、盐碱等胁迫的耐受性,促进作物在逆境条件下的稳产.SUMO化修饰在植物根系发育调控中的重要作用也为作物遗传改良提供了新的思路.通过有针对性对调控根系发育的SUMO相关基因进行改造,有望培育出更能适应贫瘠土壤、具备较高产量潜力的作物新种质. ...
SUMO E3 ligase AtMMS21-dependent SUMOylation of AUXIN/INDOLE-3-ACETIC ACID 17 regulates auxin signaling
1
2023
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
Sumoylation of BRI1-EMS-SUPPRESSOR 1 (BES1) by the SUMO E3 ligase SIZ1 negatively regulates brassinosteroids signaling in Arabidopsis thaliana
1
2019
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
Inhibition of SIZ1-mediated SUMOylation of HOOKLESS1 promotes light-induced apical hook opening in Arabidopsis
1
2023
... SUMO E3连接酶在植物营养生长期的生长发育中发挥广泛作用,参与了对根、顶端弯钩、叶片等发育过程的调控.SIZ1与MMS21通过介导靶蛋白SUMO化,在调控根系结构建成方面发挥重要作用.Kenji[68]发现AtSIZ1介导ABI5的SUMO化修饰从而负调控ABA信号转导途径,siz1突变体对ABA表现出超敏反应,导致初生根生长受到抑制.AtMMS21可通过对染色质重塑因子BRAHMA(BRM)进行SUMO化修饰增强其蛋白稳定性,从而维持根尖分生组织活性,mms21-1突变体在根发育方面表现出明显缺陷,过表达BRM可部分恢复其表型,表明二者在同一通路中发挥作用[69].此外,AtMMS21还可以通过介导26S蛋白酶体亚基RPT2a的SUMO化修饰维持蛋白酶体复合物的功能稳定性,从而保证拟南芥根部正常发育[70].苹果MdSIZ1可以通过增强MdARF8的SUMO化水平提高其蛋白稳定性,从而促进MdGH3表达,最终促进侧根形成[71].水稻突变体osmms21表现出短根与矮化表型,对生长素处理敏感性降低,同时生长素信号相关基因OsIAA9和OsIAA13表达下调,表明OsMMS21可能通过调控生长素信号介导根发育过程[72].近期研究发现,AtMMS21通过SUMO化IAA17的K41位点增强其稳定性,维持IAA17在根尖的积累,从而抑制生长素响应并精细调控根的发育;而高浓度生长素可降低IAA17的SUMO化水平,促进其降解,最终促进根生长[73].AtSIZ1介导BES1的SUMO化修饰负调控BR信号通路,突变体siz1在下胚轴伸长过程中对BR具有超敏反应[74].HOOKLESS1(HLS1)是拟南芥顶钩形成的关键调控因子,SIZ1通过维持HLS1蛋白在黑暗条件下的SUMO化修饰状态促进其寡聚化,进而维持顶钩结构;而在光照下,随着SIZ1表达下降,PhyB与HY5协同解除HLS1的SUMO化修饰,启动顶钩展开过程,这一调控机制揭示了SIZ1在光形态建成中的动态调节功能[75]. ...
BAG6-A from Fragaria viridis pollen modulates gametophyte development in diploid strawberry
1
2023
... FviBAG6-A是绿草莓(Fragariaviridis)中一个关键的生殖发育调控因子,其与E3 SUMO连接酶FviSIZ1互作并发生SUMO化修饰,在调控配子体发育中发挥重要作用,在绿草莓中过表达FviBAG6-A导致雄性绒毡层和中层提前降解、花粉败育,雌性胚珠母细胞发育异常,种子败育率达80%以上[76].在拟南芥种子发育过程中,SIZ1直接介导种子储存蛋白CRU1/2/3的SUMO化修饰从而维持其稳定性,siz1-2突变体的种子发育有缺陷、种子数量减少,但种子中总蛋白和氨基酸的含量增加,表明AtSIZ1通过调控贮藏蛋白的积累和稳定性影响种子发育[77].SIZ1还通过调控赤霉素(gibberellic acid, GA)信号精确协调拟南芥生长,其直接结合并SUMO化修饰F-box蛋白SLY1,增强其对DELLA蛋白RGA的泛素化降解,从而解除DELLA对生长的抑制[78].该过程形成GA诱导的SLY1SUMO化正反馈环路进一步强化信号,在种子萌发阶段,SIZ1缺失导致SLY1稳定性下降,休眠相关基因(DOG1、ABI3)及DELLA基因(RGA、GAI)表达上调,引发种子过度休眠,而外源GA或后熟处理可部分恢复种子萌发能力[79].最新研究发现,AtSIZ1通过介导转录因子AL6的SUMO化修饰,负调控种子休眠和热抑制的关键调控因子基因DOG1的表达,SUMO化的AL6能够结合DOG1启动子并避免DOG1蛋白被泛素化降解,高温条件下AL6的SUMO化水平下降,导致AL6蛋白水平降低、DOG1表达上升并抑制种子萌发,说明AL6的可逆SUMO化能够精细调控DOG1表达,有助于精确建立拟南芥种子休眠和抑制高温下的种子萌发[80]. ...
E3 SUMO ligase AtSIZ1 regulates the cruciferin content of Arabidopsis seeds
1
2019
... FviBAG6-A是绿草莓(Fragariaviridis)中一个关键的生殖发育调控因子,其与E3 SUMO连接酶FviSIZ1互作并发生SUMO化修饰,在调控配子体发育中发挥重要作用,在绿草莓中过表达FviBAG6-A导致雄性绒毡层和中层提前降解、花粉败育,雌性胚珠母细胞发育异常,种子败育率达80%以上[76].在拟南芥种子发育过程中,SIZ1直接介导种子储存蛋白CRU1/2/3的SUMO化修饰从而维持其稳定性,siz1-2突变体的种子发育有缺陷、种子数量减少,但种子中总蛋白和氨基酸的含量增加,表明AtSIZ1通过调控贮藏蛋白的积累和稳定性影响种子发育[77].SIZ1还通过调控赤霉素(gibberellic acid, GA)信号精确协调拟南芥生长,其直接结合并SUMO化修饰F-box蛋白SLY1,增强其对DELLA蛋白RGA的泛素化降解,从而解除DELLA对生长的抑制[78].该过程形成GA诱导的SLY1SUMO化正反馈环路进一步强化信号,在种子萌发阶段,SIZ1缺失导致SLY1稳定性下降,休眠相关基因(DOG1、ABI3)及DELLA基因(RGA、GAI)表达上调,引发种子过度休眠,而外源GA或后熟处理可部分恢复种子萌发能力[79].最新研究发现,AtSIZ1通过介导转录因子AL6的SUMO化修饰,负调控种子休眠和热抑制的关键调控因子基因DOG1的表达,SUMO化的AL6能够结合DOG1启动子并避免DOG1蛋白被泛素化降解,高温条件下AL6的SUMO化水平下降,导致AL6蛋白水平降低、DOG1表达上升并抑制种子萌发,说明AL6的可逆SUMO化能够精细调控DOG1表达,有助于精确建立拟南芥种子休眠和抑制高温下的种子萌发[80]. ...
E3 SUMO ligase AtSIZ1 positively regulates SLY1-mediated GA signalling and plant development
1
2015
... FviBAG6-A是绿草莓(Fragariaviridis)中一个关键的生殖发育调控因子,其与E3 SUMO连接酶FviSIZ1互作并发生SUMO化修饰,在调控配子体发育中发挥重要作用,在绿草莓中过表达FviBAG6-A导致雄性绒毡层和中层提前降解、花粉败育,雌性胚珠母细胞发育异常,种子败育率达80%以上[76].在拟南芥种子发育过程中,SIZ1直接介导种子储存蛋白CRU1/2/3的SUMO化修饰从而维持其稳定性,siz1-2突变体的种子发育有缺陷、种子数量减少,但种子中总蛋白和氨基酸的含量增加,表明AtSIZ1通过调控贮藏蛋白的积累和稳定性影响种子发育[77].SIZ1还通过调控赤霉素(gibberellic acid, GA)信号精确协调拟南芥生长,其直接结合并SUMO化修饰F-box蛋白SLY1,增强其对DELLA蛋白RGA的泛素化降解,从而解除DELLA对生长的抑制[78].该过程形成GA诱导的SLY1SUMO化正反馈环路进一步强化信号,在种子萌发阶段,SIZ1缺失导致SLY1稳定性下降,休眠相关基因(DOG1、ABI3)及DELLA基因(RGA、GAI)表达上调,引发种子过度休眠,而外源GA或后熟处理可部分恢复种子萌发能力[79].最新研究发现,AtSIZ1通过介导转录因子AL6的SUMO化修饰,负调控种子休眠和热抑制的关键调控因子基因DOG1的表达,SUMO化的AL6能够结合DOG1启动子并避免DOG1蛋白被泛素化降解,高温条件下AL6的SUMO化水平下降,导致AL6蛋白水平降低、DOG1表达上升并抑制种子萌发,说明AL6的可逆SUMO化能够精细调控DOG1表达,有助于精确建立拟南芥种子休眠和抑制高温下的种子萌发[80]. ...
The E3 SUMO ligase AtSIZ1 functions in seed germination in Arabidopsis
1
2016
... FviBAG6-A是绿草莓(Fragariaviridis)中一个关键的生殖发育调控因子,其与E3 SUMO连接酶FviSIZ1互作并发生SUMO化修饰,在调控配子体发育中发挥重要作用,在绿草莓中过表达FviBAG6-A导致雄性绒毡层和中层提前降解、花粉败育,雌性胚珠母细胞发育异常,种子败育率达80%以上[76].在拟南芥种子发育过程中,SIZ1直接介导种子储存蛋白CRU1/2/3的SUMO化修饰从而维持其稳定性,siz1-2突变体的种子发育有缺陷、种子数量减少,但种子中总蛋白和氨基酸的含量增加,表明AtSIZ1通过调控贮藏蛋白的积累和稳定性影响种子发育[77].SIZ1还通过调控赤霉素(gibberellic acid, GA)信号精确协调拟南芥生长,其直接结合并SUMO化修饰F-box蛋白SLY1,增强其对DELLA蛋白RGA的泛素化降解,从而解除DELLA对生长的抑制[78].该过程形成GA诱导的SLY1SUMO化正反馈环路进一步强化信号,在种子萌发阶段,SIZ1缺失导致SLY1稳定性下降,休眠相关基因(DOG1、ABI3)及DELLA基因(RGA、GAI)表达上调,引发种子过度休眠,而外源GA或后熟处理可部分恢复种子萌发能力[79].最新研究发现,AtSIZ1通过介导转录因子AL6的SUMO化修饰,负调控种子休眠和热抑制的关键调控因子基因DOG1的表达,SUMO化的AL6能够结合DOG1启动子并避免DOG1蛋白被泛素化降解,高温条件下AL6的SUMO化水平下降,导致AL6蛋白水平降低、DOG1表达上升并抑制种子萌发,说明AL6的可逆SUMO化能够精细调控DOG1表达,有助于精确建立拟南芥种子休眠和抑制高温下的种子萌发[80]. ...
SUMOylation of AL6 regulates seed dormancy and thermoinhibition in Arabidopsis
1
2025
... FviBAG6-A是绿草莓(Fragariaviridis)中一个关键的生殖发育调控因子,其与E3 SUMO连接酶FviSIZ1互作并发生SUMO化修饰,在调控配子体发育中发挥重要作用,在绿草莓中过表达FviBAG6-A导致雄性绒毡层和中层提前降解、花粉败育,雌性胚珠母细胞发育异常,种子败育率达80%以上[76].在拟南芥种子发育过程中,SIZ1直接介导种子储存蛋白CRU1/2/3的SUMO化修饰从而维持其稳定性,siz1-2突变体的种子发育有缺陷、种子数量减少,但种子中总蛋白和氨基酸的含量增加,表明AtSIZ1通过调控贮藏蛋白的积累和稳定性影响种子发育[77].SIZ1还通过调控赤霉素(gibberellic acid, GA)信号精确协调拟南芥生长,其直接结合并SUMO化修饰F-box蛋白SLY1,增强其对DELLA蛋白RGA的泛素化降解,从而解除DELLA对生长的抑制[78].该过程形成GA诱导的SLY1SUMO化正反馈环路进一步强化信号,在种子萌发阶段,SIZ1缺失导致SLY1稳定性下降,休眠相关基因(DOG1、ABI3)及DELLA基因(RGA、GAI)表达上调,引发种子过度休眠,而外源GA或后熟处理可部分恢复种子萌发能力[79].最新研究发现,AtSIZ1通过介导转录因子AL6的SUMO化修饰,负调控种子休眠和热抑制的关键调控因子基因DOG1的表达,SUMO化的AL6能够结合DOG1启动子并避免DOG1蛋白被泛素化降解,高温条件下AL6的SUMO化水平下降,导致AL6蛋白水平降低、DOG1表达上升并抑制种子萌发,说明AL6的可逆SUMO化能够精细调控DOG1表达,有助于精确建立拟南芥种子休眠和抑制高温下的种子萌发[80]. ...
Rice SIZ1, a SUMO E3 ligase, controls spikelet fertility through regulation of anther dehiscence
1
2011
... 水稻SIZ1在调控花药开裂中发挥重要作用,siz1突变体及RNAi株系成熟小穗中高分子量蛋白的SUMO化程度显著降低,次生壁形成相关基因的表达下降,花药内皮层和隔膜裂解受阻导致花粉释放受限,从而影响花药开裂和植株结实率[81].拟南芥siz1突变体在花序纤维细胞中表现出次生细胞壁(secondary cell wall, SCW)缺陷,AtSIZ1通过对LBD30 K226位点的SUMO化激活SND1/NST1介导的转录调控网络,激活控制SCW合成的基因,从而促进SCW纤维素与木质素合成[82]. ...
SUMO modification of LBD30 by SIZ1 regulates secondary cell wall formation in Arabidopsis thaliana
1
2019
... 水稻SIZ1在调控花药开裂中发挥重要作用,siz1突变体及RNAi株系成熟小穗中高分子量蛋白的SUMO化程度显著降低,次生壁形成相关基因的表达下降,花药内皮层和隔膜裂解受阻导致花粉释放受限,从而影响花药开裂和植株结实率[81].拟南芥siz1突变体在花序纤维细胞中表现出次生细胞壁(secondary cell wall, SCW)缺陷,AtSIZ1通过对LBD30 K226位点的SUMO化激活SND1/NST1介导的转录调控网络,激活控制SCW合成的基因,从而促进SCW纤维素与木质素合成[82]. ...
FLC-mediated flowering repression is positively regulated by sumoylation
1
2014
... FLC是植物开花转变的核心抑制因子,SIZ1通过多层次调控FLC延迟植物开花:一方面,SIZ1直接结合FLC并抑制其K154位点的SUMO化修饰,延缓FLC被蛋白酶体降解,维持其稳定性[83];另一方面,SIZ1通过SUMO化修饰组蛋白去乙酰化酶FLD,解除FLD对FLC的表观沉默,从而促进FLC转录[84].拟南芥MMS21/HYP2也可介导FLC的SUMO化修饰从而增强其稳定性,hpy2-2突变体的开花时间提前[85].此外,SIZ1还能与HDA6相互作用,抑制其与靶基因FLC和MAF4的结合,提高组蛋白H3乙酰化水平,从而增强FLC和MAF4的表达,进而延迟开花[86]. ...
The SUMO E3 ligase, AtSIZ1, regulates flowering by controlling a salicylic acid-mediated floral promotion pathway and through affects on FLC chromatin structure
1
2008
... FLC是植物开花转变的核心抑制因子,SIZ1通过多层次调控FLC延迟植物开花:一方面,SIZ1直接结合FLC并抑制其K154位点的SUMO化修饰,延缓FLC被蛋白酶体降解,维持其稳定性[83];另一方面,SIZ1通过SUMO化修饰组蛋白去乙酰化酶FLD,解除FLD对FLC的表观沉默,从而促进FLC转录[84].拟南芥MMS21/HYP2也可介导FLC的SUMO化修饰从而增强其稳定性,hpy2-2突变体的开花时间提前[85].此外,SIZ1还能与HDA6相互作用,抑制其与靶基因FLC和MAF4的结合,提高组蛋白H3乙酰化水平,从而增强FLC和MAF4的表达,进而延迟开花[86]. ...
Arabidopsis HIGH PLOIDY2 sumoylates and stabilizes flowering locus C through its E3 ligase activity
1
2016
... FLC是植物开花转变的核心抑制因子,SIZ1通过多层次调控FLC延迟植物开花:一方面,SIZ1直接结合FLC并抑制其K154位点的SUMO化修饰,延缓FLC被蛋白酶体降解,维持其稳定性[83];另一方面,SIZ1通过SUMO化修饰组蛋白去乙酰化酶FLD,解除FLD对FLC的表观沉默,从而促进FLC转录[84].拟南芥MMS21/HYP2也可介导FLC的SUMO化修饰从而增强其稳定性,hpy2-2突变体的开花时间提前[85].此外,SIZ1还能与HDA6相互作用,抑制其与靶基因FLC和MAF4的结合,提高组蛋白H3乙酰化水平,从而增强FLC和MAF4的表达,进而延迟开花[86]. ...
Arabidopsis SUMO E3 ligase SIZ1 interacts with HDA6 and negatively regulates HDA6 function during flowering
1
2021
... FLC是植物开花转变的核心抑制因子,SIZ1通过多层次调控FLC延迟植物开花:一方面,SIZ1直接结合FLC并抑制其K154位点的SUMO化修饰,延缓FLC被蛋白酶体降解,维持其稳定性[83];另一方面,SIZ1通过SUMO化修饰组蛋白去乙酰化酶FLD,解除FLD对FLC的表观沉默,从而促进FLC转录[84].拟南芥MMS21/HYP2也可介导FLC的SUMO化修饰从而增强其稳定性,hpy2-2突变体的开花时间提前[85].此外,SIZ1还能与HDA6相互作用,抑制其与靶基因FLC和MAF4的结合,提高组蛋白H3乙酰化水平,从而增强FLC和MAF4的表达,进而延迟开花[86]. ...
AtMMS21, an SMC5/6 complex subunit, is involved in stem cell niche maintenance and DNA damage responses in Arabidopsis roots
1
2013
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
SUMO E3 ligase AtMMS21 is required for normal meiosis and gametophyte development in Arabidopsis
1
2014
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
The SUMO ligase MMS21 profoundly influences maize development through its impact on genome activity and stability
1
2021
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
Generation of viable hypomorphic and null mutant plants via CRISPR-Cas9 targeting mRNA splicing sites
1
2025
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
The Arabidopsis SUMO E3 ligase AtMMS21 dissociates the E2Fa/DPa complex in cell cycle regulation
1
2016
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
SUMOylation stabilizes sister kinetochore biorientation to allow timely anaphase
1
2021
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
The SUMO E3 ligase MdSIZ1 sumoylates a cell number regulator MdCNR8 to control organ size
1
2022
... 植物SUMO E3连接酶通过多靶点修饰构建起精密的细胞周期调控网络,在维持染色体稳定性、分生组织稳态及细胞周期时序等过程中发挥关键作用.AtMMS21/HPY2作用于分生组织形成关键调控转录因子PLT的下游,正调控细胞分裂素通路(CRE1-ARR1/ARR3/4/5/7)及细胞分裂标志蛋白CYCB1的活性以促进分生区细胞增殖,AtMMS21/HPY2的活性还同时受到细胞分裂素的抑制从而促进细胞分化,进而在根系发育中维持细胞增殖与分化的平衡,其突变体mms21-1因CYCB1活性降低、CRE1/ARRs表达受抑,导致细胞周期停滞和分生区细胞减少[16].SMC5/6复合体作为真核生物中高度保守的染色体维持因子,在DNA修复、基因组完整性维持及细胞分裂中发挥关键作用,拟南芥AtMMS21是SMC5/6复合体的关键亚基,能够维持根部干细胞小体的结构与功能,AtMMS21突变导致拟南芥干细胞定位紊乱、细胞死亡增加、对DNA损伤敏感性增强[87].在减数分裂阶段,AtMMS21介导SMC5/6复合物的SUMO化修饰,调控染色体稳定性和同源重组修复过程,其突变体mms21-1中出现染色体断裂、四分体形成异常的现象,并伴随RAD51、SPO11-1等减数分裂基因表达紊乱,导致配子体发育异常和植株半不育[88].玉米MMS21/HPY2/NSE2介导的SMC5/6复合物和其他靶点的SUMO化通过影响染色质结构来实现适当的基因表达,mms21突变体呈现生长迟缓、根短、叶片减少及种子发育延迟等表型,突变体植株中种子贮藏蛋白mRNA及DNA修复相关基因异常表达[89].最近,Yoshimura等[90]利用CRISPR-Cas9靶向剪接位点构建了HPY2的多个突变体,其中功能缺失型突变体hpy2-cr1和hpy2-cr2的幼苗致死,而低活性等位基因突变体hpy2-cr3可存活至生殖期,该等位基因产生异常剪接但保留部分功能区,从而保留了一定生物活性,该研究进一步确立了HPY2/AtMMS21在细胞周期调控和维持染色体稳定性中的核心作用,也展示了通过剪接位点编辑策略获得可生存突变体、解析必需基因功能的可行性.Liu等[91]研究发现AtMMS21通过竞争和SUMO化的双重分子机制抑制E2Fa/DPa复合物功能从而调控细胞周期,其N端直接干扰DPa二聚化域与E2Fa互作,C端则催化DpaK139/K140/K146位点SUMO化,降低复合物核定位效率并抑制ETG1、MCM3等G1/S转换基因的表达,与CDKB/CYCB介导的G2/M停滞形成协同调控网络.拟南芥Siz1/Siz2通过SUMO化修饰定位于着丝粒周围的染色体保护蛋白Shugoshin 1(Sgo1)调控有丝分裂进程,SUMO化修饰削弱Sgo1与PP2A的结合能力,降低CPC在着丝粒上的锚定活性,促进双定向染色体分离,并通过调控Sgo1从染色质释放及Bir1亚基稳定性实现中期到后期的精准转换,保障胞质分裂完整性[92].MdSIZ1通过SUMO化修饰膜定位蛋白MdCNR8促使其向细胞核中易位,过表达MdSIZ1使细胞增殖加速、器官尺寸增加,而其功能缺失则导致相反表型[93]. ...
PTI-ETI crosstalk: an integrative view of plant immunity
1
2021
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
Crosstalk between ubiquitination and other post-translational protein modifications in plant immunity
1
2020
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
Systemic acquired resistance: turning local infection into global defense
1
2013
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
Sumoylation E3 ligase SIZ1 modulates plant immunity partly through the immune receptor gene SNC1 in Arabidopsis
1
2017
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
SIZ1-mediated SUMOylation of TPR1 suppresses plant immunity in Arabidopsis
2
2019
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
... 尽管SUMO通路在作物性状改良中具有应用潜力,但仍面临技术瓶颈.其关键挑战在于实现特异性调控:SUMO E3连接酶具有多种底物,过度表达或敲除关键基因可能产生多重效应,既可能增强抗逆性,也可能扰乱激素平衡、影响生长发育.因此,需采用组织特异或诱导型策略,在特定时空精确调控SUMO化水平.此外,需要建立简单易行的检测体系:相较于传统育种性状,植物中蛋白的SUMO化水平难以直接检测.目前植物种蛋白SUMO化水平的检测主要依赖实验室条件下的分子分析,未来或可借助标记报告系统,在植物体内指示SUMO通路的活性状态,加速育种筛选进程.三是SUMO化通路的成员组成和作用机制在不同植物物种中可能存在差异,还需深入研究阐明.有趣的是,一些跨物种研究证实SUMO途径在植物中具有较强的保守性,说明不同作物中的SUMO调控网络在整体框架上可能存在共性[106-107].植物SIZ1和MMS21/HPY2型SUMO E3连接酶均包含进化上高度保守的SP-RING核心催化结构域,保障其在调控植物发育、胁迫应答和维持基因组稳定性中的基本功能;然而,非结构域区域的序列特征和某些植物中的基因家族扩张也赋予了SUMO E3连接酶在底物识别与调控途径方面的特异性调控能力和功能分化潜力.例如,拟南芥中的AtSIZ1在免疫抑制方面具有独特作用,如抑制TPR1-HDA19复合物的活性以阻止过度免疫反应[98],而水稻中的OsSIZ1对干旱、高温和盐胁迫表现出更强的适应性[25],可能涉及不同SUMO化底物的选择.这种保守性与多样性的结合使得SUMO E3连接酶能够灵活参与并调节复杂的信号网络,以适应环境变化与植物自身的生长需求.因此,结合比较基因组学与模式植物研究,系统鉴定各主要农作物中特有的SUMO E3及其功能靶标,将有助于在保守机制基础上制定更具作物特异性的分子育种策略.未来,随着研究的不断深入,SUMO E3连接酶有望被应用于作物抗逆性和发育相关性状的遗传改良,为培育适应复杂环境条件的作物新品种提供新的途径. ...
NUA positively regulates plant immunity by coordination with ESD4 to deSUMOylate TPR1 in Arabidopsis
1
2024
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
Virulence strategies of an insect herbivore and oomycete plant pathogen converge on host E3 SUMO ligase SIZ1
1
2022
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
Damage-associated molecular pattern-triggered immunity in plants
1
2019
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
SUMOylation controls peptide processing to generate damage-associated molecular patterns in Arabidopsis
1
2025
... 与哺乳动物依赖特化的免疫细胞进行防御的机制不同,植物进化形成的两层免疫系统——由病原相关分子模式触发的免疫(pathogen-associated molecular pattern-triggered immunity, PTI)和效应蛋白诱导的免疫(effector-triggered immunity, ETI)构成了抵御病原体侵染的核心防线[94],这两个免疫层级的调控枢纽(包括PTI中的模式识别受体和ETI中的R蛋白复合体)及许多关键的免疫信号传导组分都受到PTM的动态调控,以实现细胞内稳态过程中的细胞迅速重新编程[95].其中,SUMO E3连接酶在协调免疫平衡中的调控机制已成为植物免疫研究领域的重要方向.系统获得抗性(systemic acquired resistance, SAR)是由SA系统性积累触发,通过诱导病程相关蛋白(pathogenesis-related proteins, PR)表达激活的一种广谱抗性的植物免疫机制[96].Lee等[23]研究发现,siz1突变体表现出SAR,植株体内SA水平显著上升、PR基因表达增加,以及对丁香假单胞菌番茄致病变种Pst DC3000的抗性显著增强,首次在分子层面证实SIZ1作为关键抑制因子调控植物免疫稳态.Chen等[62,64]发现SIZ1依赖的SUMO化修饰动态调控金属转运蛋白YSL3的表达,在拟南芥受到Pst DC3000感染时,YSL3通过NPR1被SA信号激活,并受JA信号拮抗调控,从而介导植物对病原菌的系统性防御.Gou等[97]发现拟南芥SIZ1通过负调控NB-LRR型免疫受体SNC1参与植物免疫应答,siz1-3突变体因SNC1异常激活引发温度依赖性自身免疫反应,表现为生长抑制和抗病性增强,而SIZ1过表达通过降低SNC1转录水平及蛋白稳定性抑制免疫过度激活,SNC1蛋白在体内被SUMO1和SUMO2修饰,且其SUMO化与泛素化降解通路关键因子CPR1相关.Hammoudi等[35]在此基础上系统解析了拟南芥中SIZ1介导的免疫-生长平衡机制,SIZ1持续抑制SNC1介导的自身免疫信号,即使在高温(28 ℃)下仍维持部分抗性,这种效应依赖EDS1/SNC1通路.研究发现,SIZ1通过SUMO化修饰转录共抑制因子TPR1抑制其活性,而关键修饰位点K282和K721的突变阻断其SUMO化,使得TPR1的转录共抑制活性增强,且与组蛋白去乙酰化酶HDA19的互作增加,导致抗病负调控基因DND1/DND2表达下调,从而激活免疫反应,此外,SUMO化还可能通过增强TPR1与免疫受体SNC1的互作,或协同泛素化降解通路动态平衡免疫信号[98].最近的研究发现,核孔锚定蛋白NUA可与SUMO特异性蛋白酶ESD4协同介导对TPR1的去SUMO化,从而恢复TPR1介导的DND1/2等免疫相关基因表达并增强植物对多种病原的抗性,nua突变体中TPR1的SUMO化水平升高,病程相关基因表达下降,对细菌、真菌和卵菌的敏感性增强[99].此外,SIZ1还能通过独立于SNC1、EDS1/PAD4的机制调控植物的广谱抗性,拟南芥siz1突变体对蚜虫(Myzuspersicae)和卵菌(Phytophthoracapsici)抗性显著增强,蚜虫效应蛋白Mp64通过稳定SIZ1蛋白水平增强其功能,而卵菌效应蛋白CRN83_152通过提高SIZ1的SUMO E3连接酶活性促进SUMO化修饰,二者通过不同机制增强植物感病性[100].在植物免疫系统中,损伤相关分子模式(damage associated molecular pattern, DAMP)的及时释放对于快速诱导防御反应至关重要,植物激发子肽(plant elicitor peptide,Pep)是一种具有代表性的DAMP,经过原蛋白酶切割从其前体PROPEP中释放出来[101].近期研究发现,拟南芥PROPEP家族成员PROPEP1可在SIZ1的作用下发生SUMO化修饰,增强与其特异蛋白酶MC4(含SUMO互作基序SIM)的结合效率,促进其裂解并释放成熟的Pep1肽,从而显著激活PDF1.2等防御相关基因的表达,进而增强植物对细胞壁受损的免疫响应能力[102]. ...
SUMO conjugation to the pattern recognition receptor FLS2 triggers intracellular signalling in plant innate immunity
1
2018
... SUMO E3连接酶对底物蛋白调控的作用机制仍有诸多未知之处.尽管体外条件下部分底物可在没有E3的情况下被SUMO化[103],但植物体内尚未发现E3非依赖性修饰的证据,暗示着E3在细胞环境中可能对底物识别与修饰的特异性与效率具有关键调控作用.然而,不同E3如何识别特定底物,以及其如何介导底物上单一SUMO或多聚SUMO链的连接,仍未得到完全阐释.此外,虽然动物和真菌中存在SIZ1/PIAS类、RanBP2类和Pc2类3种SUMO E3,但植物中尚未发现与Pc2类具有同源性和相似功能的蛋白,植物是否含有Pc2类E3的功能同源物仍待探究.近年来研究者们在生物界鉴定出越来越多新的SUMO E3连接酶,其中包括一些病毒蛋白,例如人类腺病毒(Human adenovirus)的E4-ORF3,但它们发挥SUMO E3连接酶活性的结构基础尚不清楚[104].因此,SUMO E3连接酶家族在植物中可能还存在未被发现的成员,该家族现有成员的底物谱和作用原理亦有待深入研究.目前,植物中研究较为深入的SUMO E3连接酶主要包括SIZ1和MMS21,它们的底物涉及多种重要转录调控因子、胁迫响应蛋白和关键代谢酶等.其主要通过以下机制对底物发挥调控作用:一是提高SUMO化修饰效率和特异性,促进E2与底物的高效结合;二是通过SUMO化修饰调节底物蛋白的稳定性,影响其降解速率;三是改变底物蛋白的亚细胞定位或转录活性,从而精细调控相关的发育或胁迫响应过程.明确SUMO E3连接酶的底物对于揭示SUMO E3连接酶调控的生物学过程具有重要意义,然而,由于SUMO化在植物中的丰度较低且动态性高,鉴定SUMO化蛋白和位点极具挑战性.目前用于鉴定SUMO化蛋白的方法主要包括免疫学方法和质谱分析法.利用酵母双杂交和免疫共沉淀等传统方法仅能鉴定到少量底物,近期,南方科技大学研究团队建立了一种两步赖氨酸缺失SUMO富集方法进行质谱分析,大幅提高了检测灵敏度,在拟南芥中鉴定到1 300多个SUMO化蛋白[105].其他植物中的SUMO化蛋白尚未得到系统性鉴定.显然,与动物中的研究相比,植物中SUMO化底物蛋白鉴定仍然存在很大的局限性.在未来的研究中,可以尝试结合冷冻电镜技术解析瞬态SUMO化复合物结构,以及借助人工智能预测SUMO化热点区域,优先确定高概率修饰位点. ...
The role of SUMO E3 ligases in signaling pathway of cancer cells
1
2022
... SUMO E3连接酶对底物蛋白调控的作用机制仍有诸多未知之处.尽管体外条件下部分底物可在没有E3的情况下被SUMO化[103],但植物体内尚未发现E3非依赖性修饰的证据,暗示着E3在细胞环境中可能对底物识别与修饰的特异性与效率具有关键调控作用.然而,不同E3如何识别特定底物,以及其如何介导底物上单一SUMO或多聚SUMO链的连接,仍未得到完全阐释.此外,虽然动物和真菌中存在SIZ1/PIAS类、RanBP2类和Pc2类3种SUMO E3,但植物中尚未发现与Pc2类具有同源性和相似功能的蛋白,植物是否含有Pc2类E3的功能同源物仍待探究.近年来研究者们在生物界鉴定出越来越多新的SUMO E3连接酶,其中包括一些病毒蛋白,例如人类腺病毒(Human adenovirus)的E4-ORF3,但它们发挥SUMO E3连接酶活性的结构基础尚不清楚[104].因此,SUMO E3连接酶家族在植物中可能还存在未被发现的成员,该家族现有成员的底物谱和作用原理亦有待深入研究.目前,植物中研究较为深入的SUMO E3连接酶主要包括SIZ1和MMS21,它们的底物涉及多种重要转录调控因子、胁迫响应蛋白和关键代谢酶等.其主要通过以下机制对底物发挥调控作用:一是提高SUMO化修饰效率和特异性,促进E2与底物的高效结合;二是通过SUMO化修饰调节底物蛋白的稳定性,影响其降解速率;三是改变底物蛋白的亚细胞定位或转录活性,从而精细调控相关的发育或胁迫响应过程.明确SUMO E3连接酶的底物对于揭示SUMO E3连接酶调控的生物学过程具有重要意义,然而,由于SUMO化在植物中的丰度较低且动态性高,鉴定SUMO化蛋白和位点极具挑战性.目前用于鉴定SUMO化蛋白的方法主要包括免疫学方法和质谱分析法.利用酵母双杂交和免疫共沉淀等传统方法仅能鉴定到少量底物,近期,南方科技大学研究团队建立了一种两步赖氨酸缺失SUMO富集方法进行质谱分析,大幅提高了检测灵敏度,在拟南芥中鉴定到1 300多个SUMO化蛋白[105].其他植物中的SUMO化蛋白尚未得到系统性鉴定.显然,与动物中的研究相比,植物中SUMO化底物蛋白鉴定仍然存在很大的局限性.在未来的研究中,可以尝试结合冷冻电镜技术解析瞬态SUMO化复合物结构,以及借助人工智能预测SUMO化热点区域,优先确定高概率修饰位点. ...
Highly sensitive site-specific SUMOylation proteomics in Arabidopsis
1
2024
... SUMO E3连接酶对底物蛋白调控的作用机制仍有诸多未知之处.尽管体外条件下部分底物可在没有E3的情况下被SUMO化[103],但植物体内尚未发现E3非依赖性修饰的证据,暗示着E3在细胞环境中可能对底物识别与修饰的特异性与效率具有关键调控作用.然而,不同E3如何识别特定底物,以及其如何介导底物上单一SUMO或多聚SUMO链的连接,仍未得到完全阐释.此外,虽然动物和真菌中存在SIZ1/PIAS类、RanBP2类和Pc2类3种SUMO E3,但植物中尚未发现与Pc2类具有同源性和相似功能的蛋白,植物是否含有Pc2类E3的功能同源物仍待探究.近年来研究者们在生物界鉴定出越来越多新的SUMO E3连接酶,其中包括一些病毒蛋白,例如人类腺病毒(Human adenovirus)的E4-ORF3,但它们发挥SUMO E3连接酶活性的结构基础尚不清楚[104].因此,SUMO E3连接酶家族在植物中可能还存在未被发现的成员,该家族现有成员的底物谱和作用原理亦有待深入研究.目前,植物中研究较为深入的SUMO E3连接酶主要包括SIZ1和MMS21,它们的底物涉及多种重要转录调控因子、胁迫响应蛋白和关键代谢酶等.其主要通过以下机制对底物发挥调控作用:一是提高SUMO化修饰效率和特异性,促进E2与底物的高效结合;二是通过SUMO化修饰调节底物蛋白的稳定性,影响其降解速率;三是改变底物蛋白的亚细胞定位或转录活性,从而精细调控相关的发育或胁迫响应过程.明确SUMO E3连接酶的底物对于揭示SUMO E3连接酶调控的生物学过程具有重要意义,然而,由于SUMO化在植物中的丰度较低且动态性高,鉴定SUMO化蛋白和位点极具挑战性.目前用于鉴定SUMO化蛋白的方法主要包括免疫学方法和质谱分析法.利用酵母双杂交和免疫共沉淀等传统方法仅能鉴定到少量底物,近期,南方科技大学研究团队建立了一种两步赖氨酸缺失SUMO富集方法进行质谱分析,大幅提高了检测灵敏度,在拟南芥中鉴定到1 300多个SUMO化蛋白[105].其他植物中的SUMO化蛋白尚未得到系统性鉴定.显然,与动物中的研究相比,植物中SUMO化底物蛋白鉴定仍然存在很大的局限性.在未来的研究中,可以尝试结合冷冻电镜技术解析瞬态SUMO化复合物结构,以及借助人工智能预测SUMO化热点区域,优先确定高概率修饰位点. ...
Defining the SUMO system in maize: SUMOylation is up-regulated during endosperm development and rapidly induced by stress
1
2016
... 尽管SUMO通路在作物性状改良中具有应用潜力,但仍面临技术瓶颈.其关键挑战在于实现特异性调控:SUMO E3连接酶具有多种底物,过度表达或敲除关键基因可能产生多重效应,既可能增强抗逆性,也可能扰乱激素平衡、影响生长发育.因此,需采用组织特异或诱导型策略,在特定时空精确调控SUMO化水平.此外,需要建立简单易行的检测体系:相较于传统育种性状,植物中蛋白的SUMO化水平难以直接检测.目前植物种蛋白SUMO化水平的检测主要依赖实验室条件下的分子分析,未来或可借助标记报告系统,在植物体内指示SUMO通路的活性状态,加速育种筛选进程.三是SUMO化通路的成员组成和作用机制在不同植物物种中可能存在差异,还需深入研究阐明.有趣的是,一些跨物种研究证实SUMO途径在植物中具有较强的保守性,说明不同作物中的SUMO调控网络在整体框架上可能存在共性[106-107].植物SIZ1和MMS21/HPY2型SUMO E3连接酶均包含进化上高度保守的SP-RING核心催化结构域,保障其在调控植物发育、胁迫应答和维持基因组稳定性中的基本功能;然而,非结构域区域的序列特征和某些植物中的基因家族扩张也赋予了SUMO E3连接酶在底物识别与调控途径方面的特异性调控能力和功能分化潜力.例如,拟南芥中的AtSIZ1在免疫抑制方面具有独特作用,如抑制TPR1-HDA19复合物的活性以阻止过度免疫反应[98],而水稻中的OsSIZ1对干旱、高温和盐胁迫表现出更强的适应性[25],可能涉及不同SUMO化底物的选择.这种保守性与多样性的结合使得SUMO E3连接酶能够灵活参与并调节复杂的信号网络,以适应环境变化与植物自身的生长需求.因此,结合比较基因组学与模式植物研究,系统鉴定各主要农作物中特有的SUMO E3及其功能靶标,将有助于在保守机制基础上制定更具作物特异性的分子育种策略.未来,随着研究的不断深入,SUMO E3连接酶有望被应用于作物抗逆性和发育相关性状的遗传改良,为培育适应复杂环境条件的作物新品种提供新的途径. ...
Understanding SUMO-mediated adaptive responses in plants to improve crop productivity
1
2022
... 尽管SUMO通路在作物性状改良中具有应用潜力,但仍面临技术瓶颈.其关键挑战在于实现特异性调控:SUMO E3连接酶具有多种底物,过度表达或敲除关键基因可能产生多重效应,既可能增强抗逆性,也可能扰乱激素平衡、影响生长发育.因此,需采用组织特异或诱导型策略,在特定时空精确调控SUMO化水平.此外,需要建立简单易行的检测体系:相较于传统育种性状,植物中蛋白的SUMO化水平难以直接检测.目前植物种蛋白SUMO化水平的检测主要依赖实验室条件下的分子分析,未来或可借助标记报告系统,在植物体内指示SUMO通路的活性状态,加速育种筛选进程.三是SUMO化通路的成员组成和作用机制在不同植物物种中可能存在差异,还需深入研究阐明.有趣的是,一些跨物种研究证实SUMO途径在植物中具有较强的保守性,说明不同作物中的SUMO调控网络在整体框架上可能存在共性[106-107].植物SIZ1和MMS21/HPY2型SUMO E3连接酶均包含进化上高度保守的SP-RING核心催化结构域,保障其在调控植物发育、胁迫应答和维持基因组稳定性中的基本功能;然而,非结构域区域的序列特征和某些植物中的基因家族扩张也赋予了SUMO E3连接酶在底物识别与调控途径方面的特异性调控能力和功能分化潜力.例如,拟南芥中的AtSIZ1在免疫抑制方面具有独特作用,如抑制TPR1-HDA19复合物的活性以阻止过度免疫反应[98],而水稻中的OsSIZ1对干旱、高温和盐胁迫表现出更强的适应性[25],可能涉及不同SUMO化底物的选择.这种保守性与多样性的结合使得SUMO E3连接酶能够灵活参与并调节复杂的信号网络,以适应环境变化与植物自身的生长需求.因此,结合比较基因组学与模式植物研究,系统鉴定各主要农作物中特有的SUMO E3及其功能靶标,将有助于在保守机制基础上制定更具作物特异性的分子育种策略.未来,随着研究的不断深入,SUMO E3连接酶有望被应用于作物抗逆性和发育相关性状的遗传改良,为培育适应复杂环境条件的作物新品种提供新的途径. ...