







生物技术通报 ›› 2025, Vol. 41 ›› Issue (12): 214-224.doi: 10.13560/j.cnki.biotech.bull.1985.2025-0490
杜雨晴1( ), 蒋路园1, 汪奕衡2, 吴宸炜1, 杨梦露1, 刘旭升3, 王晓君4, 邱德有1, 樊玮5(
), 蒋路园1, 汪奕衡2, 吴宸炜1, 杨梦露1, 刘旭升3, 王晓君4, 邱德有1, 樊玮5( ), 杨艳芳1(
), 杨艳芳1( )
)
收稿日期:2025-05-13
出版日期:2025-12-26
发布日期:2026-01-06
通讯作者:
杨艳芳,女,博士,研究员,研究方向 :林木次生代谢工程与调控;E-mail: echoyyf@caf.ac.cn作者简介:杜雨晴,女,硕士研究生,研究方向 :林木次生代谢;E-mail: dyq991115@163.com
基金资助:
DU Yu-qing1( ), JIANG Lu-yuan1, WANG Yi-heng2, WU Chen-wei1, YANG Meng-lu1, LIU Xu-sheng3, WANG Xiao-jun4, QIU De-you1, FAN Wei5(
), JIANG Lu-yuan1, WANG Yi-heng2, WU Chen-wei1, YANG Meng-lu1, LIU Xu-sheng3, WANG Xiao-jun4, QIU De-you1, FAN Wei5( ), YANG Yan-fang1(
), YANG Yan-fang1( )
)
Received:2025-05-13
Published:2025-12-26
Online:2026-01-06
摘要:
目的 红豆杉(Taxus spp.)为国家一级濒危保护植物,阐明太行山地区南方红豆杉种群遗传多样性和遗传变异,可为南方红豆杉遗传多样性保护及培育新品种提供指导。 方法 基于前期筛选出的8对稳定性和多态性良好的SSR引物,对太行山区的9个南方红豆杉种群以及3个来自浙江、湖南和福建3省的南方红豆杉种群(共106个体)的遗传多样性和遗传变异进行分析。 结果 12个南方红豆杉种群的平均观测等位基因数(Na)为2.781,平均有效等位基因数(Ne)为2.074,Shannon多样性指数(I)为0.679,平均观测杂合度Ho=0.390,平均期望杂合度He=0.381。通过对种群间遗传分化指数(Fst)和基因流强度(Nm)进行分析发现,太行山区南方红豆杉种群间大多为中低水平的遗传分化(Fst<0.15)且存在较为频繁的基因交流(Nm>>1),而太行山区的南方红豆杉种群与来自浙江、湖南和福建3省的南方红豆杉种群的分化程度较高(Fst>0.25),基因交流水平低(Nm<1)。此外,AMOVA分析显示,南方红豆杉的遗传变异主要发生在个体内,种群间和个体间的遗传变异占比较低。STRUCTURE分析和聚类分析表明,106份材料可以分成2大类,其中福建、湖南和浙江省份种源聚为一类,太行山地区的南方红豆杉聚为一类,且大多数样品间彼此相互混杂,具有较为丰富的遗传背景。 结论 南方红豆杉种群整体具有较丰富的遗传多样性,但太行山区种群的遗传多样性相对偏低。该区域内种群间呈现中低水平的遗传分化,基因交流程度较高;然而,太行山区种群与低纬度地区种群间则表现出较高的遗传分化。
杜雨晴, 蒋路园, 汪奕衡, 吴宸炜, 杨梦露, 刘旭升, 王晓君, 邱德有, 樊玮, 杨艳芳. 太行山地区南方红豆杉SSR遗传多样性分析[J]. 生物技术通报, 2025, 41(12): 214-224.
DU Yu-qing, JIANG Lu-yuan, WANG Yi-heng, WU Chen-wei, YANG Meng-lu, LIU Xu-sheng, WANG Xiao-jun, QIU De-you, FAN Wei, YANG Yan-fang. Genetic Diversity of Taxus chinensis var. mairei in Taihang Mountains Based on SSR Molecular Markers[J]. Biotechnology Bulletin, 2025, 41(12): 214-224.
种群编号 Population code | 产地 Resource | 个体数 Number of individuals |
|---|---|---|
| 19 | 河南三门峡市卢氏县 Lushi County, Sanmenxia City, Henan Province | 10 |
| 20 | 河南南阳市南召县 Nanzhao County, Nanyang City, Henan Province | 10 |
| 52 | 河南洛阳市汝阳县 Ruyang County, Luoyang City, Henan Province | 10 |
| LS | 山西省晋城市沁水县 Qinshui County, Jincheng City, Shanxi Province | 10 |
| MH | 河南省济源市 Jiyuan City, Henan Province | 11 |
| SD | 山西省晋城市陵川县 Lingchuan County, Jincheng City, Shanxi Province | 10 |
| SM | 山西省晋城市陵川县 Lingchuan County, Jincheng City, Shanxi Province | 10 |
| TT | 山西省晋城市阳城县 Yangcheng County, Jincheng City, Shanxi Province | 11 |
| YTH | 山西省晋城市陵川县 Lingchuan County, Jincheng City, Shanxi Province | 10 |
| FJNF | 福建省南平市 Nanping City, Fujian Province | 5 |
| HNNF | 湖南省永州市 Yongzhou City, Hunan Province | 5 |
| ZJNF | 浙江省丽水市 Lishui City, Zhejiang Province | 5 |
表1 南方红豆杉种群采样信息表
Table 1 Sampling information of T. chinensis var. mairei populations
种群编号 Population code | 产地 Resource | 个体数 Number of individuals |
|---|---|---|
| 19 | 河南三门峡市卢氏县 Lushi County, Sanmenxia City, Henan Province | 10 |
| 20 | 河南南阳市南召县 Nanzhao County, Nanyang City, Henan Province | 10 |
| 52 | 河南洛阳市汝阳县 Ruyang County, Luoyang City, Henan Province | 10 |
| LS | 山西省晋城市沁水县 Qinshui County, Jincheng City, Shanxi Province | 10 |
| MH | 河南省济源市 Jiyuan City, Henan Province | 11 |
| SD | 山西省晋城市陵川县 Lingchuan County, Jincheng City, Shanxi Province | 10 |
| SM | 山西省晋城市陵川县 Lingchuan County, Jincheng City, Shanxi Province | 10 |
| TT | 山西省晋城市阳城县 Yangcheng County, Jincheng City, Shanxi Province | 11 |
| YTH | 山西省晋城市陵川县 Lingchuan County, Jincheng City, Shanxi Province | 10 |
| FJNF | 福建省南平市 Nanping City, Fujian Province | 5 |
| HNNF | 湖南省永州市 Yongzhou City, Hunan Province | 5 |
| ZJNF | 浙江省丽水市 Lishui City, Zhejiang Province | 5 |
引物名称 Primer name | 引物序列 Primer sequences (5′-3′) | 退火温度 Annealing temperature (℃) | 荧光标签 Fluorescent labels |
|---|---|---|---|
| HDS10 | F: | 55 | FAM |
| R: CCTCAGCAACAGACACAGGA | |||
| HDS52 | F: | 55 | VIC |
| R: GTAAACATCGCCTTCCTTGC | |||
| HDS54 | F: | 55 | VIC |
| R: TTCACCTGCCAATCTAAGGG | |||
| HDS76 | F: | 55 | NED |
| R: GCTTCAACATTGCAAAACGA | |||
| HDS82 | F: | 54 | NED |
| R: GTGGATTCGGTCACTTTGGT | |||
| HDS84 | F: | 55 | NED |
| R: AAGCATAAAAGGCGAGAGCA | |||
| HDS86 | F: | 55 | NED |
| R: CGTTTCAAGAAAAACGGGAA | |||
| HDS88 | F: | 53 | NED |
| R: TGCCCAATAAACTATCATCTCC |
表2 多态引物信息表
Table 2 Information of polymorphic primers
引物名称 Primer name | 引物序列 Primer sequences (5′-3′) | 退火温度 Annealing temperature (℃) | 荧光标签 Fluorescent labels |
|---|---|---|---|
| HDS10 | F: | 55 | FAM |
| R: CCTCAGCAACAGACACAGGA | |||
| HDS52 | F: | 55 | VIC |
| R: GTAAACATCGCCTTCCTTGC | |||
| HDS54 | F: | 55 | VIC |
| R: TTCACCTGCCAATCTAAGGG | |||
| HDS76 | F: | 55 | NED |
| R: GCTTCAACATTGCAAAACGA | |||
| HDS82 | F: | 54 | NED |
| R: GTGGATTCGGTCACTTTGGT | |||
| HDS84 | F: | 55 | NED |
| R: AAGCATAAAAGGCGAGAGCA | |||
| HDS86 | F: | 55 | NED |
| R: CGTTTCAAGAAAAACGGGAA | |||
| HDS88 | F: | 53 | NED |
| R: TGCCCAATAAACTATCATCTCC |
引物名称 Primer name | 等位基因数 Na | 有效等位基因数 Ne | Shannon’s指数 I | 观测杂合度 Ho | 期望杂合度 He |
|---|---|---|---|---|---|
| HDS 10 | 2.417 | 1.786 | 0.612 | 0.449 | 0.377 |
| HDS 52 | 1.500 | 1.307 | 0.251 | 0.067 | 0.158 |
| HDS 54 | 1.917 | 1.474 | 0.423 | 0.302 | 0.261 |
| HDS 76 | 5.583 | 4.038 | 1.441 | 0.549 | 0.705 |
| HDS 82 | 4.083 | 2.632 | 1.070 | 0.701 | 0.576 |
| HDS 84 | 2.750 | 2.314 | 0.794 | 0.561 | 0.469 |
| HDS 86 | 1.500 | 1.092 | 0.136 | 0.070 | 0.071 |
| HDS 88 | 2.500 | 1.951 | 0.702 | 0.423 | 0.433 |
| Total | 22.250 | 16.594 | 5.429 | 3.122 | 3.050 |
| Average | 2.781 | 2.074 | 0.679 | 0.390 | 0.381 |
表3 八对多态SSR引物的多态性信息表
Table 3 Polymorphism information of 8 pairs of SSR primers
引物名称 Primer name | 等位基因数 Na | 有效等位基因数 Ne | Shannon’s指数 I | 观测杂合度 Ho | 期望杂合度 He |
|---|---|---|---|---|---|
| HDS 10 | 2.417 | 1.786 | 0.612 | 0.449 | 0.377 |
| HDS 52 | 1.500 | 1.307 | 0.251 | 0.067 | 0.158 |
| HDS 54 | 1.917 | 1.474 | 0.423 | 0.302 | 0.261 |
| HDS 76 | 5.583 | 4.038 | 1.441 | 0.549 | 0.705 |
| HDS 82 | 4.083 | 2.632 | 1.070 | 0.701 | 0.576 |
| HDS 84 | 2.750 | 2.314 | 0.794 | 0.561 | 0.469 |
| HDS 86 | 1.500 | 1.092 | 0.136 | 0.070 | 0.071 |
| HDS 88 | 2.500 | 1.951 | 0.702 | 0.423 | 0.433 |
| Total | 22.250 | 16.594 | 5.429 | 3.122 | 3.050 |
| Average | 2.781 | 2.074 | 0.679 | 0.390 | 0.381 |
种群编号 Population code | 等位基因数 Na | 有效等位基因数 Ne | Shannon’s指数 I | 观测杂合度 Ho | 期望杂合度 He |
|---|---|---|---|---|---|
| 19 | 2.250 | 1.897 | 0.542 | 0.306 | 0.310 |
| 20 | 2.125 | 1.681 | 0.496 | 0.388 | 0.301 |
| 52 | 2.250 | 1.795 | 0.543 | 0.452 | 0.316 |
| LS | 3.000 | 2.107 | 0.755 | 0.357 | 0.433 |
| MH | 2.875 | 2.088 | 0.747 | 0.356 | 0.421 |
| SD | 3.250 | 2.407 | 0.739 | 0.396 | 0.395 |
| SM | 3.500 | 2.108 | 0.744 | 0.363 | 0.389 |
| TT | 3.125 | 2.247 | 0.690 | 0.378 | 0.348 |
| YTH | 2.875 | 2.031 | 0.689 | 0.313 | 0.403 |
| Total 1 | 25.250 | 18.360 | 5.946 | 3.306 | 3.316 |
| Mean 1 | 2.806 | 2.040 | 0.661 | 0.367 | 0.368 |
| FJNF | 2.375 | 1.806 | 0.576 | 0.400 | 0.333 |
| HHNF | 2.500 | 2.256 | 0.720 | 0.475 | 0.428 |
| ZJNF | 3.250 | 2.470 | 0.902 | 0.500 | 0.500 |
| Total 2 | 33.375 | 24.893 | 8.143 | 4.684 | 4.577 |
| Mean 2 | 2.781 | 2.074 | 0.679 | 0.390 | 0.381 |
表4 南方红豆杉的遗传多样性参数
Table 4 Genetic diversity parameters of T. chinensis var. mairei
种群编号 Population code | 等位基因数 Na | 有效等位基因数 Ne | Shannon’s指数 I | 观测杂合度 Ho | 期望杂合度 He |
|---|---|---|---|---|---|
| 19 | 2.250 | 1.897 | 0.542 | 0.306 | 0.310 |
| 20 | 2.125 | 1.681 | 0.496 | 0.388 | 0.301 |
| 52 | 2.250 | 1.795 | 0.543 | 0.452 | 0.316 |
| LS | 3.000 | 2.107 | 0.755 | 0.357 | 0.433 |
| MH | 2.875 | 2.088 | 0.747 | 0.356 | 0.421 |
| SD | 3.250 | 2.407 | 0.739 | 0.396 | 0.395 |
| SM | 3.500 | 2.108 | 0.744 | 0.363 | 0.389 |
| TT | 3.125 | 2.247 | 0.690 | 0.378 | 0.348 |
| YTH | 2.875 | 2.031 | 0.689 | 0.313 | 0.403 |
| Total 1 | 25.250 | 18.360 | 5.946 | 3.306 | 3.316 |
| Mean 1 | 2.806 | 2.040 | 0.661 | 0.367 | 0.368 |
| FJNF | 2.375 | 1.806 | 0.576 | 0.400 | 0.333 |
| HHNF | 2.500 | 2.256 | 0.720 | 0.475 | 0.428 |
| ZJNF | 3.250 | 2.470 | 0.902 | 0.500 | 0.500 |
| Total 2 | 33.375 | 24.893 | 8.143 | 4.684 | 4.577 |
| Mean 2 | 2.781 | 2.074 | 0.679 | 0.390 | 0.381 |
种群编号 Population code | 19 | 20 | 52 | LS | MH | SD | SM | TT | YTH | ZJNF | FJNF | HNNF |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 19 | - | 0.89 | 12.25 | 2.02 | 1.31 | 1.54 | 1.31 | 1.22 | 0.89 | 0.51 | 0.38 | 0.31 |
| 20 | 0.22 | - | 1.07 | 2.25 | 3.32 | 3.92 | 3.92 | 2.88 | 0.79 | 0.61 | 0.27 | 0.32 |
| 52 | 0.02 | 0.19 | - | 2.25 | 1.54 | 1.83 | 1.67 | 1.42 | 1.00 | 0.56 | 0.36 | 0.33 |
| LS | 0.11 | 0.10 | 0.10 | - | 3.92 | 6.00 | 3.32 | 2.53 | 1.42 | 0.71 | 0.46 | 0.43 |
| MH | 0.16 | 0.07 | 0.14 | 0.06 | - | 8.08 | 4.75 | 2.88 | 1.07 | 0.68 | 0.46 | 0.41 |
| SD | 0.14 | 0.06 | 0.12 | 0.04 | 0.03 | - | 4.75 | 3.32 | 1.22 | 0.68 | 0.38 | 0.39 |
| SM | 0.16 | 0.06 | 0.13 | 0.07 | 0.05 | 0.05 | - | 3.32 | 1.07 | 0.71 | 0.38 | 0.39 |
| TT | 0.17 | 0.08 | 0.15 | 0.09 | 0.08 | 0.07 | 0.07 | - | 0.84 | 0.56 | 0.31 | 0.31 |
| YTH | 0.22 | 0.24 | 0.20 | 0.15 | 0.19 | 0.17 | 0.19 | 0.23 | - | 0.75 | 0.46 | 0.46 |
| ZJNF | 0.33 | 0.29 | 0.31 | 0.26 | 0.27 | 0.27 | 0.26 | 0.31 | 0.25 | - | 0.79 | 2.25 |
| FJNF | 0.40 | 0.48 | 0.41 | 0.35 | 0.35 | 0.40 | 0.40 | 0.45 | 0.35 | 0.24 | - | 0.75 |
| HNNF | 0.45 | 0.44 | 0.43 | 0.37 | 0.38 | 0.39 | 0.39 | 0.45 | 0.35 | 0.10 | 0.25 | - |
表5 遗传分化系数Fst(下三角)与基因流Nm(上三角)分析
Table 5 Analysis of genetic differentiation coefficient Fst (below triangle of the table) and Nm (above triangle of the table)
种群编号 Population code | 19 | 20 | 52 | LS | MH | SD | SM | TT | YTH | ZJNF | FJNF | HNNF |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 19 | - | 0.89 | 12.25 | 2.02 | 1.31 | 1.54 | 1.31 | 1.22 | 0.89 | 0.51 | 0.38 | 0.31 |
| 20 | 0.22 | - | 1.07 | 2.25 | 3.32 | 3.92 | 3.92 | 2.88 | 0.79 | 0.61 | 0.27 | 0.32 |
| 52 | 0.02 | 0.19 | - | 2.25 | 1.54 | 1.83 | 1.67 | 1.42 | 1.00 | 0.56 | 0.36 | 0.33 |
| LS | 0.11 | 0.10 | 0.10 | - | 3.92 | 6.00 | 3.32 | 2.53 | 1.42 | 0.71 | 0.46 | 0.43 |
| MH | 0.16 | 0.07 | 0.14 | 0.06 | - | 8.08 | 4.75 | 2.88 | 1.07 | 0.68 | 0.46 | 0.41 |
| SD | 0.14 | 0.06 | 0.12 | 0.04 | 0.03 | - | 4.75 | 3.32 | 1.22 | 0.68 | 0.38 | 0.39 |
| SM | 0.16 | 0.06 | 0.13 | 0.07 | 0.05 | 0.05 | - | 3.32 | 1.07 | 0.71 | 0.38 | 0.39 |
| TT | 0.17 | 0.08 | 0.15 | 0.09 | 0.08 | 0.07 | 0.07 | - | 0.84 | 0.56 | 0.31 | 0.31 |
| YTH | 0.22 | 0.24 | 0.20 | 0.15 | 0.19 | 0.17 | 0.19 | 0.23 | - | 0.75 | 0.46 | 0.46 |
| ZJNF | 0.33 | 0.29 | 0.31 | 0.26 | 0.27 | 0.27 | 0.26 | 0.31 | 0.25 | - | 0.79 | 2.25 |
| FJNF | 0.40 | 0.48 | 0.41 | 0.35 | 0.35 | 0.40 | 0.40 | 0.45 | 0.35 | 0.24 | - | 0.75 |
| HNNF | 0.45 | 0.44 | 0.43 | 0.37 | 0.38 | 0.39 | 0.39 | 0.45 | 0.35 | 0.10 | 0.25 | - |
变异来源 Source of variance | 自由度 df | 平方和 SS | 平均方差 MS | 估算差异值 Est. var. | 变异百分比 Variation percentage (%) | P | |
|---|---|---|---|---|---|---|---|
| 所有红豆杉种群 | 种群间 Among populations | 11 | 141.831 | 12.894 | 0.605 | 24 | <0.001 |
| 个体间 Among indiv. | 94 | 213.282 | 2.269 | 0.399 | 16 | <0.001 | |
| 个体内 Within indiv. | 106 | 156.000 | 1.472 | 1.472 | 60 | <0.001 | |
| 总计 Total | 211 | 511.113 | - | 2.475 | 100 | - | |
| 太行山区红豆杉种群 | 种群间 Among populations | 8 | 69.400 | 8.675 | 0.315 | 15 | <0.001 |
| 个体间 Among indiv. | 82 | 188.782 | 2.302 | 0.445 | 20 | <0.001 | |
| 个体内 Within indiv. | 91 | 128.500 | 1.412 | 1.412 | 65 | <0.001 | |
| 总计 Total | 181 | 386.681 | - | 2.172 | 100 | - | |
表6 基于SSR标记的南方红豆杉种群的分子方差分析
Table 6 Analysis of molecular variance (AMOVA) of T. chinensis var. mairei populations based on SSR markers
变异来源 Source of variance | 自由度 df | 平方和 SS | 平均方差 MS | 估算差异值 Est. var. | 变异百分比 Variation percentage (%) | P | |
|---|---|---|---|---|---|---|---|
| 所有红豆杉种群 | 种群间 Among populations | 11 | 141.831 | 12.894 | 0.605 | 24 | <0.001 |
| 个体间 Among indiv. | 94 | 213.282 | 2.269 | 0.399 | 16 | <0.001 | |
| 个体内 Within indiv. | 106 | 156.000 | 1.472 | 1.472 | 60 | <0.001 | |
| 总计 Total | 211 | 511.113 | - | 2.475 | 100 | - | |
| 太行山区红豆杉种群 | 种群间 Among populations | 8 | 69.400 | 8.675 | 0.315 | 15 | <0.001 |
| 个体间 Among indiv. | 82 | 188.782 | 2.302 | 0.445 | 20 | <0.001 | |
| 个体内 Within indiv. | 91 | 128.500 | 1.412 | 1.412 | 65 | <0.001 | |
| 总计 Total | 181 | 386.681 | - | 2.172 | 100 | - | |
种群编号 Population code | 19 | 20 | 52 | LS | MH | SD | SM | TT | YTH | ZJNF | FJNF | HNNF |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 19 | 0.000 | |||||||||||
| 20 | 0.260 | 0.000 | ||||||||||
| 52 | 0.015 | 0.219 | 0.000 | |||||||||
| LS | 0.151 | 0.147 | 0.141 | 0.000 | ||||||||
| MH | 0.224 | 0.104 | 0.199 | 0.089 | 0.000 | |||||||
| SD | 0.178 | 0.082 | 0.147 | 0.060 | 0.047 | 0.000 | ||||||
| SM | 0.201 | 0.074 | 0.175 | 0.108 | 0.079 | 0.056 | 0.000 | |||||
| TT | 0.229 | 0.094 | 0.205 | 0.146 | 0.116 | 0.096 | 0.100 | 0.000 | ||||
| YTH | 0.371 | 0.449 | 0.353 | 0.343 | 0.418 | 0.325 | 0.370 | 0.464 | 0.000 | |||
| ZJNF | 1.016 | 0.810 | 0.906 | 0.908 | 0.944 | 0.841 | 0.839 | 1.072 | 0.770 | 0.000 | ||
| FJNF | 0.969 | 1.334 | 1.021 | 0.990 | 1.197 | 1.136 | 1.323 | 1.307 | 1.089 | 0.607 | 0.000 | |
| HNNF | 1.610 | 1.536 | 1.517 | 1.571 | 1.550 | 1.467 | 1.491 | 2.097 | 1.192 | 0.242 | 0.572 | 0.000 |
表7 南方红豆杉种群间的遗传距离分析
Table 7 Genetic distance among populations of T. chinensis var. mairei
种群编号 Population code | 19 | 20 | 52 | LS | MH | SD | SM | TT | YTH | ZJNF | FJNF | HNNF |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 19 | 0.000 | |||||||||||
| 20 | 0.260 | 0.000 | ||||||||||
| 52 | 0.015 | 0.219 | 0.000 | |||||||||
| LS | 0.151 | 0.147 | 0.141 | 0.000 | ||||||||
| MH | 0.224 | 0.104 | 0.199 | 0.089 | 0.000 | |||||||
| SD | 0.178 | 0.082 | 0.147 | 0.060 | 0.047 | 0.000 | ||||||
| SM | 0.201 | 0.074 | 0.175 | 0.108 | 0.079 | 0.056 | 0.000 | |||||
| TT | 0.229 | 0.094 | 0.205 | 0.146 | 0.116 | 0.096 | 0.100 | 0.000 | ||||
| YTH | 0.371 | 0.449 | 0.353 | 0.343 | 0.418 | 0.325 | 0.370 | 0.464 | 0.000 | |||
| ZJNF | 1.016 | 0.810 | 0.906 | 0.908 | 0.944 | 0.841 | 0.839 | 1.072 | 0.770 | 0.000 | ||
| FJNF | 0.969 | 1.334 | 1.021 | 0.990 | 1.197 | 1.136 | 1.323 | 1.307 | 1.089 | 0.607 | 0.000 | |
| HNNF | 1.610 | 1.536 | 1.517 | 1.571 | 1.550 | 1.467 | 1.491 | 2.097 | 1.192 | 0.242 | 0.572 | 0.000 |
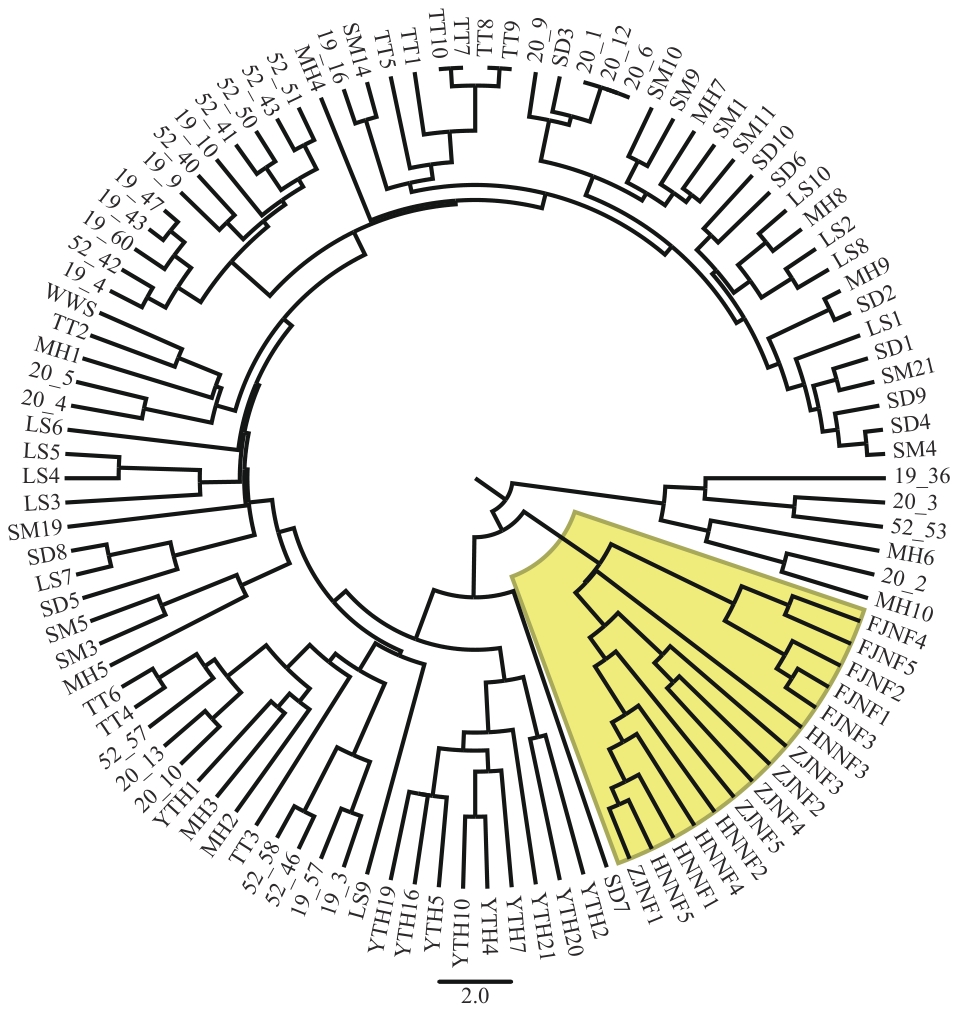
图1 106份南方红豆杉材料聚类分析图黄色部分代表FJNF、HNNF和ZJNF种群
Fig. 1 Cluster analysis of 106 T. chinensis var. mairei materialsThe yellow section indicates the FJNF, HNNF, and ZJNF populations
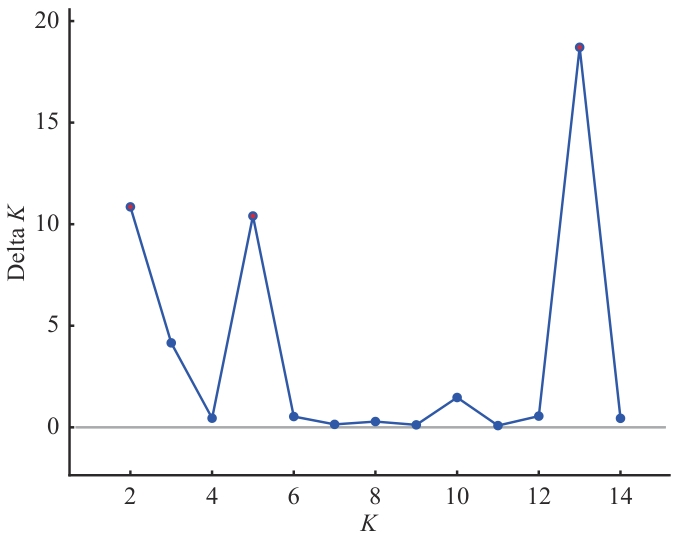
图2 不同分组的delta K曲线图横坐标K代表分组,纵坐标delta K表示Structure软件对分组的判定,delta K越高分组越合理
Fig. 2 The delta K curve graph for different groupsThe horizontal coordinate K indicates the number of clusters, while the vertical coordinate delta K indicates the determination of group division by the Structure software. A higher delta K value suggests a more meaningful grouping
| [1] | 李朝阳, 陈玲, 刘世彪, 等. 古丈县南方红豆杉天然群体的遗传多样性研究 [J]. 中国野生植物资源, 2007, 26(6): 58-60. |
| Li ZY, Chen L, Liu SB, et al. Study on the genetic diversity of natural population of Taxus chinensis in Guzhang in Hunan province [J]. Chin Wild Plant Resour, 2007, 26(6): 58-60. | |
| [2] | 茹文明, 秦永燕, 张桂萍, 等. 濒危植物南方红豆杉遗传多样性的RAPD分析 [J]. 植物研究, 2008, 28(6): 698-704. |
| Ru WM, Qin YY, Zhang GP, et al. Genetic diversity of rare and endangered plant Taxus chinensis var. mairei [J]. Bull Bot Res, 2008, 28(6): 698-704. | |
| [3] | 张蕊, 周志春, 金国庆, 等. 南方红豆杉种源遗传多样性和遗传分化 [J]. 林业科学, 2009, 45(1): 50-56. |
| Zhang R, Zhou ZC, Jin GQ, et al. Genetic diversity and genetic differentiation of Taxus wallichiana var. mairei provenances [J]. Sci Silvae Sin, 2009, 45(1): 50-56. | |
| [4] | 李乃伟, 束晓春, 何树兰, 等. 南方红豆杉的ISSR遗传多样性分析 [J]. 西北植物学报, 2010, 30(12): 2536-2541. |
| Li NW, Shu XC, He SL, et al. ISSR analysis of genetic diversity of Taxus chinensis var. mairei [J]. Acta Bot Boreali Occidentalia Sin, 2010, 30(12): 2536-2541. | |
| [5] | 谢宛余. 元宝山地区红豆杉分类地位及其遗传变异分析 [D]. 长沙: 中南林业科技大学, 2021. |
| Xie WY. Taxonomic status and intraspecified genetic variation of Taxus in Yuanbaoshan region [D]. Changsha: Central South University of Forestry & Technology, 2021. | |
| [6] | 丁桂生. 南方红豆杉种源遗传多样性和遗传分化 [J]. 中国林副特产, 2011(3): 7-11. |
| Ding GS. Provenance genetic diversity and genetic differentiation of Taxus wallichiana var. mairei [J]. For Prod Speciality China, 2011(3): 7-11. | |
| [7] | 徐雯, 瞿印权, 张玲玲, 等. 基于RAPD的福建产南方红豆杉遗传多样性研究 [J]. 中草药, 2017, 48(14): 2943-2949. |
| Xu W, Qu YQ, Zhang LL, et al. Genetic diversity of Taxus chinensis var. mairei from Fujian based on RAPD markers [J]. Chin Tradit Herb Drugs, 2017, 48(14): 2943-2949. | |
| [8] | 罗芊芊, 李峰卿, 肖德卿, 等. 两个南方红豆杉天然居群的交配系统分析 [J]. 南京林业大学学报: 自然科学版, 2023, 47(5): 80-86. |
| Luo QQ, Li FQ, Xiao DQ, et al. Mating system analyses of two natural populations of Taxus wallichiana var. mairei [J]. J Nanjing For Univ Nat Sci Ed, 2023, 47(5): 80-86. | |
| [9] | Cheng J, Wang X, Liu XN, et al. Chromosome-level genome of Himalayan yew provides insights into the origin and evolution of the paclitaxel biosynthetic pathway [J]. Mol Plant, 2021, 14(7): 1199-1209. |
| [10] | Xiong XY, Gou JB, Liao QG, et al. The Taxus genome provides insights into paclitaxel biosynthesis [J]. Nat Plants, 2021, 7(8): 1026-1036. |
| [11] | Song C, Fu FF, Yang LL, et al. Taxus yunnanensis genome offers insights into gymnosperm phylogeny and taxol production [J]. Commun Biol, 2021, 4(1): 1203. |
| [12] | 李炎林, 杨星星, 张家银, 等. 南方红豆杉转录组SSR挖掘及分子标记的研究 [J]. 园艺学报, 2014, 41(4): 735-745. |
| Li YL, Yang XX, Zhang JY, et al. Studies on SSR molecular markers based on transcriptome of Taxus chinensis var. mairei [J]. Acta Hortic Sin, 2014, 41(4): 735-745. | |
| [13] | 申响保, 朱妍洁, 徐刚标. 密叶红豆杉SSR位点分布特征及分子标记开发 [J]. 中南林业科技大学学报, 2021, 41(4): 139-147. |
| Shen XB, Zhu YJ, Xu GB. Distribution characteristics of SSR loci and development of molecular markers in Taxus fuana [J]. J Cent South Univ For Technol, 2021, 41(4): 139-147. | |
| [14] | 蒋路园, 吴文丽, 张恺恺, 等. 南方红豆杉转录组SSR位点分析及其分子标记开发 [J]. 中草药, 2024, 55(3): 928-936. |
| Jiang LY, Wu WL, Zhang KK, et al. Transcriptome sequencing and development of SSR molecular markers of Taxus chinensis var. mairei [J]. Chin Tradit Herb Drugs, 2024, 55(3): 928-936. | |
| [15] | 李孝伟, 孟丽, 许桂芳. 河南太行山南方红豆杉濒危原因分析 [J]. 现代农业科技, 2007(13): 235-237. |
| Li XW, Meng L, Xu GF. Analysis of endangered causes of Taxus mairei in Taihang Mountain, Henan province [J]. Anhui Agric, 2007(13): 235-237. | |
| [16] | 李乃伟, 贺善安, 束晓春, 等. 基于ISSR标记的南方红豆杉野生种群和迁地保护种群的遗传多样性和遗传结构分析 [J]. 植物资源与环境学报, 2011, 20(1): 25-30. |
| Li NW, He SA, Shu XC, et al. Genetic diversity and structure analyses of wild and ex-situ conservation populations of Taxus chinensis var. mairei based on ISSR marker [J]. J Plant Resour Environ, 2011, 20(1): 25-30. | |
| [17] | 吴顺, 刘坤, 罗冬艳, 等. 海拔对贵州雷公山地区南方红豆杉种群遗传多样性的影响 [J]. 中药材, 2018, 41(2): 303-307. |
| Wu S, Liu K, Luo DY, et al. Effects of altitude on genetic diversity for Taxus chinensis var. mairei at Leigongshan area in Guizhou [J]. J Chin Med Mater, 2018, 41(2): 303-307. | |
| [18] | 廉敏. 山西南方红豆杉群落谱系结构及其数量分类与排序研究 [D]. 临汾: 山西师范大学, 2020. |
| Lian M. The phylogenetic structure and quantitative analysis and ordination of Taxus chinensis var.mairei community, in Shanxi province [D]. Linfen: Shanxi Normal University, 2020. | |
| [19] | 吴长桥, 蒋路园, 杨艳芳, 等. 红豆杉属植物中紫杉烷化合物含量比较与分析 [J]. 中草药, 2021, 52(2): 538-543. |
| Wu CQ, Jiang LY, Yang YF, et al. Comparative, regression and cluster analysis on contents of six taxanes in Taxus spp [J]. Chin Tradit Herb Drugs, 2021, 52(2): 538-543. | |
| [20] | Su JY, Yan Y, Song J, et al. Recent fragmentation may not alter genetic patterns in endangered long-lived species: evidence from Taxus cuspidata [J]. Front Plant Sci, 2018, 9: 1571. |
| [21] | Poudel RC, Möller M, Liu J, et al. Low genetic diversity and high inbreeding of the endangered yews in Central Himalaya: implications for conservation of their highly fragmented populations [J]. Divers Distrib, 2014, 20(11): 1270-1284. |
| [22] | 郑超, 别庆铃, 夏冰, 等. 4种红豆杉属植物遗传多样性和遗传关系的RAPD分析 [J]. 植物资源与环境学报, 2013, 22(3): 58-62. |
| Zheng C, Bie QL, Xia B, et al. RAPD analysis of genetic diversity and genetic relationship of four species in Taxus Linn [J]. J Plant Resour Environ, 2013, 22(3): 58-62. | |
| [23] | 吴杰, 汤欢, 黄林芳, 等. 红豆杉属植物全球生态适宜性分析研究 [J]. 药学学报, 2017, 52(7): 1186-1195. |
| Wu J, Tang H, Huang LF, et al. Research and analysis of globally ecological suitability for Taxus plants [J]. Acta Pharm Sin, 2017, 52(7): 1186-1195. | |
| [24] | 程蓓蓓. 中国红豆杉属分子谱系地理学与遗传多样性研究 [D]. 北京: 中国林业科学研究院, 2016. |
| Cheng BB. Molecular phylogeographic and genetic diversity of Taxus L. (taxaeae) in China [D]. Beijing: Chinese Academy of Forestry, 2016. | |
| [25] | Zhang DQ, Zhou N. Genetic diversity and population structure of the endangered conifer Taxus wallichiana var. mairei (Taxaceae) revealed by Simple Sequence Repeat (SSR) markers [J]. Biochem Syst Ecol, 2013, 49: 107-114. |
| [26] | 张宏意, 陈月琴, 廖文波. 南方红豆杉不同居群遗传多样性的RAPD研究 [J]. 西北植物学报, 2003, 23(11): 1993-1996. |
| Zhang HY, Chen YQ, Liao WB. RAPD analysis of population genetic diversity of Taxus mairei [J]. Acta Bot Boreali Occidentalia Sin, 2003, 23(11): 1993-1996. | |
| [27] | 张玲玲. 南方红豆杉DNA指纹图谱技术研究 [D]. 福州: 福建农林大学, 2009. |
| Zhang LL. Studies on the DNA fingerprinting of Taxus Chinensis var. mairei [D]. Fuzhou: Fujian Agriculture and Forestry University, 2009. | |
| [28] | 武庭维. 中国漆树与越南红豆杉遗传多样性的研究 [D]. 杨凌: 西北农林科技大学, 2018. |
| Wu TW. Assessing the genetic diversity of economically tree species in China and Vietnam (Chinese lacquer and Vietnam conifer) [D]. Yangling: Northwest A & F University, 2018. | |
| [29] | 张原, 李鑫玉, 文亚峰, 等. 南岭山地南方红豆杉天然林与人工干扰群体的遗传变异分析 [J]. 中南林业科技大学学报, 2020, 40(6): 105-110, 131. |
| Zhang Y, Li XY, Wen YF, et al. Genetic variation analysis of natural and interference populations of Maire Yew in Nanling mountains [J]. J Cent South Univ For Technol, 2020, 40(6): 105-110, 131. | |
| [30] | Slatkin M. Gene flow and the geographic structure of natural populations [J]. Science, 1987, 236(4803): 787-792. |
| [31] | 李艳红, 张立娟, 朱文博, 等. 全球变化背景下南方红豆杉地域分布变化 [J]. 自然资源学报, 2021, 36(3): 783-792. |
| Li YH, Zhang LJ, Zhu WB, et al. Changes of Taxus chinensis var. mairei habitat distribution under global climate change [J]. J Nat Resour, 2021, 36(3): 783-792. |
| [1] | 卢瑶, 袁平平, 金鑫, 毛向红, 范向斌, 白小东. 基于SSR标记的马铃薯野生种和地方种遗传多样性分析和指纹图谱构建[J]. 生物技术通报, 2025, 41(9): 94-104. |
| [2] | 裴红霞, 汪露瑶, 李生梅, 高晶霞. 基于SCoT、SRAP和SSR分子标记的220份辣椒种质资源遗传多样性分析[J]. 生物技术通报, 2025, 41(8): 165-174. |
| [3] | 段敏杰, 李怡斐, 王春萍, 黄任中, 黄启中, 张世才. 辣椒果实颜色性状与SSR分子标记的关联分析及指纹图谱构建[J]. 生物技术通报, 2025, 41(7): 81-94. |
| [4] | 段永红, 杨欣, 于冠群, 夏俊俊, 宋陆帅, 白小东, 彭锁堂. 125份马铃薯种质资源遗传多样性及主成分分析[J]. 生物技术通报, 2025, 41(6): 130-143. |
| [5] | 程诗袁, 何佳丽, 韩福彬, 杨娜娜, 杨利平, 符勇耀. 麝香百合和‘雪皇后’的形态特征、核型及ISSR标记分析[J]. 生物技术通报, 2025, 41(3): 219-229. |
| [6] | 岳园园, 胡哲源, 何琪, 黄晨阳, 郑素月, 赵梦然. 基于全基因组SNP位点和性状特征解析我国野生肺形侧耳的遗传多样性[J]. 生物技术通报, 2025, 41(3): 282-293. |
| [7] | 于静, 于桂爽, 孙昊杰, 姜春姣, 苑广迪, 杨珍, 王志伟, 王超, 王传堂. 花生种用品质影响因素及相关标记研究[J]. 生物技术通报, 2025, 41(2): 284-294. |
| [8] | 贺涵, 刘传和, 喻梦凡, 袁梦萍, 魏岳荣, 杨敏, 邝瑞彬, 周陈平, 吴夏明, 徐泽. 基于重测序的菠萝基因组InDel标记的开发[J]. 生物技术通报, 2025, 41(2): 65-76. |
| [9] | 宋英培, 王灿, 周会汶, 孔可可, 许孟歌, 王瑞凯. 基于全基因组关联分析和遗传多样性的大豆裂荚性状解析[J]. 生物技术通报, 2025, 41(2): 97-106. |
| [10] | 毛向红, 卢瑶, 范向斌, 杜培兵, 白小东. 基于SSR荧光标记毛细管电泳的马铃薯品种遗传多样性分析及分子身份证构建[J]. 生物技术通报, 2024, 40(9): 131-140. |
| [11] | 李思琪, 张文臣, 杨柳, 付庆新, 洪新, 张海旺. 基于SSR标记的文冠果遗传多样性分析及指纹图谱构建[J]. 生物技术通报, 2024, 40(5): 74-83. |
| [12] | 陈凯凌, 武涛, 徐逸群, 高佳, 张美俊, 李欣, 贾举庆. 燕麦全基因组SSR位点鉴定及多态性标记开发[J]. 生物技术通报, 2024, 40(2): 120-129. |
| [13] | 李晴, 石雨荷, 朱珏, 李晓玲, 侯超文, 童巧珍. 基于SCoT分子标记分析白术种质资源遗传多样性及DNA指纹图谱构建[J]. 生物技术通报, 2024, 40(11): 142-151. |
| [14] | 张道磊, 甘雨军, 乐亮, 普莉. 玉米产量性状的表观遗传调控机制和育种应用[J]. 生物技术通报, 2023, 39(8): 31-42. |
| [15] | 安苗, 王彤彤, 付逸婷, 夏俊俊, 彭锁堂, 段永红. 52个马铃薯遗传多样性分析及SSR分子身份证构建[J]. 生物技术通报, 2023, 39(12): 136-147. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||