







生物技术通报 ›› 2021, Vol. 37 ›› Issue (9): 106-113.doi: 10.13560/j.cnki.biotech.bull.1985.2021-0287
胡子曜( ), 代培红, 刘超, 玛迪娜·木拉提, 王倩, 吾尕力汗·阿不都维力, 赵燚, 孙玲, 徐诗佳, 李月(
), 代培红, 刘超, 玛迪娜·木拉提, 王倩, 吾尕力汗·阿不都维力, 赵燚, 孙玲, 徐诗佳, 李月( )
)
收稿日期:2021-03-09
出版日期:2021-09-26
发布日期:2021-10-25
作者简介:胡子曜,男,硕士,研究方向:作物逆境分子生物学;E-mail: 基金资助:
HU Zi-yao( ), DAI Pei-hong, LIU Chao, Madina Mulati, WANG Qian, Wugalihan Abuduwili, ZHAO Yi, SUN Ling, XU Shi-jia, LI Yue(
), DAI Pei-hong, LIU Chao, Madina Mulati, WANG Qian, Wugalihan Abuduwili, ZHAO Yi, SUN Ling, XU Shi-jia, LI Yue( )
)
Received:2021-03-09
Published:2021-09-26
Online:2021-10-25
摘要:
通过对陆地棉小GTP结合蛋白(small GTP-binding protein)基因GhROP3在不同逆境胁迫下的响应表达模式进行研究分析,为棉花抗逆相关基因克隆及棉花抗逆分子机制研究奠定基础。利用同源克隆的方法克隆GhROP3,利用生物信息学的方法分析该基因的理化性质,通过荧光定量PCR(real-time quantitative polymerase chain reaction,qRT-PCR)方法分析GhROP3在不同逆境诱导条件下的表达模式及组织表达特异性。同时构建了该基因的VIGS沉默载体,并利用农杆菌介导法转化棉花,qRT-PCR检测沉默效率。结果表明,以陆地棉的cDNA为模板克隆得到GhROP3,其开放阅读框(ORF)为591 bp,编码一个含196个氨基酸的碱性亲水性蛋白,相对分子质量为21.75 kD。qRT-PCR分析结果表明,GhROP3在棉花幼苗根、茎、叶、子叶和下胚轴中均有表达,且在茎中表达水平最高;GhROP3对高盐、干旱、低温和棉花黄萎病菌处理都有一定程度的响应。构建该基因的VIGS沉默载体转化棉花,qRT-PCR检测GhROP3在棉花的叶片和根部沉默,证明VIGS沉默载体构建成功并可以在植物体内正常工作。GhROP3可能在陆地棉的抗逆境胁迫反应中发挥着重要的作用。
胡子曜, 代培红, 刘超, 玛迪娜·木拉提, 王倩, 吾尕力汗·阿不都维力, 赵燚, 孙玲, 徐诗佳, 李月. 陆地棉小GTP结合蛋白基因GhROP3的克隆、表达及VIGS载体的构建[J]. 生物技术通报, 2021, 37(9): 106-113.
HU Zi-yao, DAI Pei-hong, LIU Chao, Madina Mulati, WANG Qian, Wugalihan Abuduwili, ZHAO Yi, SUN Ling, XU Shi-jia, LI Yue. Molecular Cloning,Expression and VIGS Construction of a Small GTP-binding Protein Gene GhROP3 in Gossypium hirsutum[J]. Biotechnology Bulletin, 2021, 37(9): 106-113.
| 基因 Gene | 序列 Sequence(5'-3') | 用途 Application |
|---|---|---|
| GhROP3 | F-ATGAGCACTGCAAGATTTATC R-TCAAAGGAAAGCACATGTTCT | 基因克隆 |
| qGhROP3 | F-GGATCCCAGAGCTAAGACAT R-CTCCCTGAGATGTTGATATTGGTG | qRT-PCR |
| qGhUBQ7 | F-GAAGGCATTCCACCTGACCAAC R-CTTGACCTTCTTCTTCTTGTGCTTG | qRT-PCR |
| VIGS-GhROP3 | F-TCTAGAGGATCCCAGAGCTAAGACATTA R-CCCGGGTTGCAAGGCTTTCTCTTTGG | VIGS载 体构建 |
| VIGS-qGhCLA1 | F-GCCCTTTGTGCATCTTC R-CTCTAGGGGCATTGAAG | qRT-PCR |
| VIGS-qGhROP3 | F-TTCTCCTTTTGCAAGGCTTTCTC R-TGACAGACCATCCTAACGCA | qRT-PCR |
表1 引物及用途
Table 1 Primer and application
| 基因 Gene | 序列 Sequence(5'-3') | 用途 Application |
|---|---|---|
| GhROP3 | F-ATGAGCACTGCAAGATTTATC R-TCAAAGGAAAGCACATGTTCT | 基因克隆 |
| qGhROP3 | F-GGATCCCAGAGCTAAGACAT R-CTCCCTGAGATGTTGATATTGGTG | qRT-PCR |
| qGhUBQ7 | F-GAAGGCATTCCACCTGACCAAC R-CTTGACCTTCTTCTTCTTGTGCTTG | qRT-PCR |
| VIGS-GhROP3 | F-TCTAGAGGATCCCAGAGCTAAGACATTA R-CCCGGGTTGCAAGGCTTTCTCTTTGG | VIGS载 体构建 |
| VIGS-qGhCLA1 | F-GCCCTTTGTGCATCTTC R-CTCTAGGGGCATTGAAG | qRT-PCR |
| VIGS-qGhROP3 | F-TTCTCCTTTTGCAAGGCTTTCTC R-TGACAGACCATCCTAACGCA | qRT-PCR |
| 基因 Gene | 开放阅读框 ORF/bp | 氨基酸 Amino acid | 相对分子质量 Relative molecular quality/kD | 理论等电点 Theoretical isoelectric point | 平均疏水性 Average hydrophobicity | 信号肽 Signal peptide | 亚细胞定位 Subcellular localization | 脂肪系数 Fat coefficient |
|---|---|---|---|---|---|---|---|---|
| GhROP3 | 591 | 196 | 21.75 | 9.45 | -0.138 | 无None | 细胞膜Cytomembrane | 86.02 |
表2 棉花GhROP3的生物信息学分析
Table 2 Bioinformatics analysis of GhROP3 in cotton
| 基因 Gene | 开放阅读框 ORF/bp | 氨基酸 Amino acid | 相对分子质量 Relative molecular quality/kD | 理论等电点 Theoretical isoelectric point | 平均疏水性 Average hydrophobicity | 信号肽 Signal peptide | 亚细胞定位 Subcellular localization | 脂肪系数 Fat coefficient |
|---|---|---|---|---|---|---|---|---|
| GhROP3 | 591 | 196 | 21.75 | 9.45 | -0.138 | 无None | 细胞膜Cytomembrane | 86.02 |
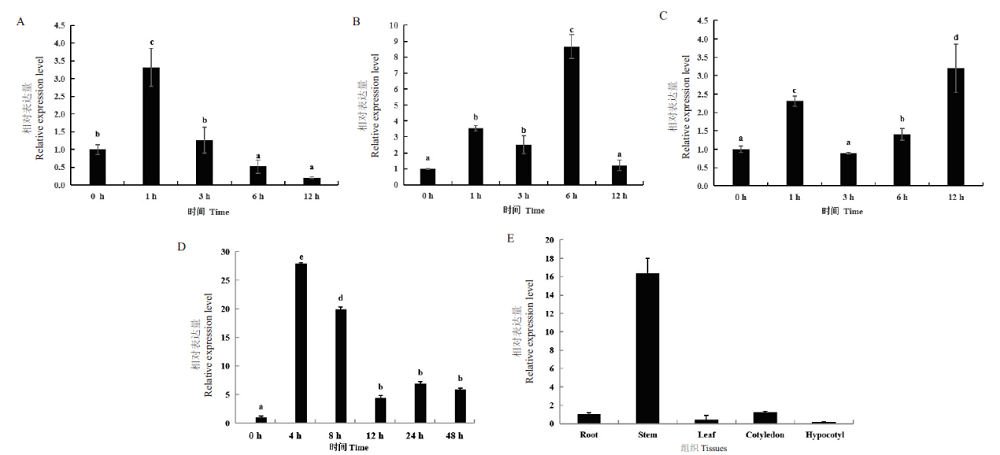
图2 GhROP3的表达谱分析 A:盐处理;B:干旱处理;C:低温处理;D:棉花黄萎病菌处理;E:GhROP3在不同组织中的表达分析。以未处理(0 h)的表达量为参照,不同胁迫处理时间不同小写字母之间表示差异显著(P<0.05)
Fig. 2 Expression pattern analysis of GhROP3 A:Salt stress. B:Drought stress. C:Cold stress. D:Verticillium wilt. E:Expression of GhROP3 in different tissues of cotton. The difference between the lowercase letters showed significant difference in different sample time at the 0.05 probability level

图3 GhROP3的VIGS载体的构建 A:GhROP3目标片段的PCR扩增;B:TRV﹕GhROP3 VIGS载体的酶切鉴定;M:Trans 2K Plus Ⅱ DNA Marker
Fig. 3 Construction of VIGS vector of GhROP3 A:PCR amplification of target fragment of GhROP3;B:identification of TRV﹕GhROP3 VIGS vectors by restriction enzyme digestion. M:Trans 2K Plus Ⅱ DNA marker
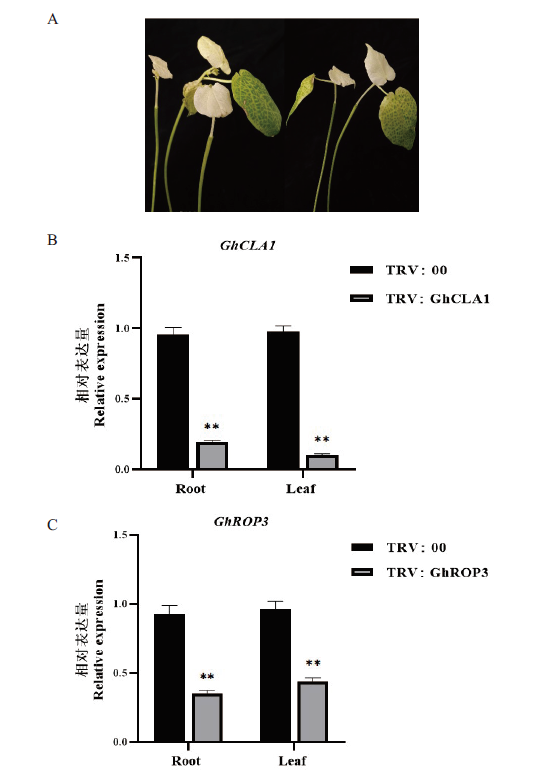
图4 VIGS技术体系验证 A:在棉花中沉默GhCLA1表型;B:GhCLA1的沉默效率检测;C:GhROP3的沉默效率检测。**:在0.01水平上存在显著差异
Fig. 4 Verification of VIGS system A:The phenotype of silenced GhCLA1 in cotton;B:silencing efficiency of GhCLA1; C:silencing efficiency of GhROP3. **:Significant difference at the 0.01 probability
| [1] |
Ono E, Wong HL, Kawasaki T, et al. Essential role of the small GTPase Rac in disease resistance of rice[J]. PNAS, 2001, 98(2):759-764.
pmid: 11149940 |
| [2] |
Assmann SM. Heterotrimeric and unconventional GTP binding proteins in plant cell signaling[J]. Plant Cell, 2002, 14(Suppl):S355-S373.
doi: 10.1105/tpc.001792 URL |
| [3] |
Takai Y, Sasaki T, Matozaki T. Small GTP-binding proteins[J]. Physiol Rev, 2001, 81(1):153-208.
pmid: 11152757 |
| [4] |
Brembu T, Winge P, Bones AM, et al. A RHOse by any other name:a comparative analysis of animal and plant Rho GTPases[J]. Cell Res, 2006, 16(5):435-445.
pmid: 16699539 |
| [5] | Eliáš M, Klimeš V. Rho GTPases:deciphering the evolutionary history of a complex protein family[J]. Methods Mol Biol, 2012, 827:13-34. |
| [6] |
Berken A, Wittinghofer A. Structure and function of Rho-type molecular switches in plants[J]. Plant Physiol Biochem, 2008, 46(3):380-393.
doi: 10.1016/j.plaphy.2007.12.008 URL |
| [7] |
Feiguelman G, Fu Y, Yalovsky S. ROP GTPases structure-function and signaling pathways[J]. Plant Physiol, 2018, 176(1):57-79.
doi: 10.1104/pp.17.01415 pmid: 29150557 |
| [8] |
Jin WW, Xu CJ, et al. Expression of ROP/RAC GTPase genes in postharvest loquat fruit in association with senescence and cold regulated lignification[J]. Postharvest Biol Technol, 2009, 54(1):9-14.
doi: 10.1016/j.postharvbio.2009.05.009 URL |
| [9] | 李先碧, 肖月华, 罗明, 等. 两个棉花Rac蛋白基因的克隆与表达分析[J]. 遗传学报, 2005, 32(1):72-78. |
| Li XB, Xiao YH, Luo M, et al. Cloning and expression analysis of two rac genes from cotton(Gossypium hirsutum L.)[J]. Acta Genet Sin, 2005, 32(1):72-78. | |
| [10] |
Delmer DP, Pear JR, Andrawis A, et al. Genes encoding small GTP-binding proteins analogous to mammalian rac are preferentially expressed in developing cotton fibers[J]. Mol Gen Genet MGG, 1995, 248(1):43-51.
doi: 10.1007/BF02456612 URL |
| [11] | 王钰静. GhROP6在棉花抗黄萎病中的功能研究[D]. 武汉:华中农业大学, 2017. |
| Wang YJ. Functional analysis of GhROP6 in cotton responsive to Verticillium dahliae[D]. Wuhan:Huazhong Agricultural University, 2017. | |
| [12] | 李月, 吾尕力汗·阿不都维力, 周垚均, 等. 陆地棉小GTP结合蛋白基因GhRop4的克隆及其表达分析[J]. 棉花学报, 2020, 32(1):21-29. |
| Li Y, Wugalihan Abuduweili, Zhou YJ, et al. Molecular cloning and expression analysis of a small GTP-binding protein gene gh Rop4 in Gossypium hirsutum L.[J]. Cotton Sci, 2020, 32(1):21-29. | |
| [13] | 李姣, 于宗霞, 冯宝民. 植物中病毒诱导基因沉默技术的研究与应用进展[J]. 分子植物育种, 2019, 17(5):1537-1542. |
| Li J, Yu ZX, Feng BM. Advances in research and application of virus induced gene silencing in plants[J]. Mol Plant Breed, 2019, 17(5):1537-1542. | |
| [14] | 王娜, 赵资博, 等. 陆地棉盐胁迫应答基因GhPEAMT1的克隆及功能分析[J]. 中国农业科学, 2021, 54(2):248-260. |
| Wang N, Zhao ZB, et al. Cloning and functional analysis of salt stress response gene GhPEAMT1 in upland cotton[J]. Sci Agric Sin, 2021, 54(2):248-260. | |
| [15] | 李秀青, 李月, 刘超, 等. 棉花黄萎病相关基因GhAAT的克隆与功能鉴定[J]. 分子植物育种, 2020, 18(4):1048-1053. |
| Li XQ, Li Y, Liu C, et al. Cloning and functional identification of cotton Verticillium wilt related gene GhAAT[J]. Mol Plant Breed, 2020, 18(4):1048-1053. | |
| [16] |
袁伟, 万红建, 杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择[J]. 植物学报, 2012, 47(4):427-436.
doi: 10.3724/SP.J.1259.2012.00427 |
| Yuan W, Wan HJ, Yang YJ. Characterization and selection of reference genes for real-time quantitative RT-PCR of plants[J]. Chin Bull Bot, 2012, 47(4):427-436. | |
| [17] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T))method[J]. Methods, 2001, 25(4):402-408.
pmid: 11846609 |
| [18] | 郑巨云, 王俊铎, 梁亚军, 等. 新疆棉花种业现状及展望[J]. 中国棉花, 2020, 47(6):1-8. |
| Zheng JY, Wang JD, Liang YJ, et al. The present status and prospect of cotton seed industry in Xinjiang[J]. China Cotton, 2020, 47(6):1-8. | |
| [19] |
Kawchuk LM, Hachey J, Lynch DR, et al. Tomato Ve disease resistance genes encode cell surface-like receptors[J]. PNAS, 2001, 98(11):6511-6515.
pmid: 11331751 |
| [20] | 陈捷胤. 海岛棉LRR-TM类抗病基因GbaVd1和GbaVd2的克隆与功能研究[D]. 北京:中国农业科学院, 2010. |
| Chen JY. Cloning and functional analysis of GbaVd1 and Gbavd2, LRR-TM like, disease resistance genes in Gossypium bar-badense[D]. Beijing:Chinese Academy of Agricultural Sciences, 2010. | |
| [21] |
Zhang B, Yang Y, Chen T, et al. Island cotton Gbve1 gene encoding a receptor-like protein confers resistance to both defoliating and non-defoliating isolates of Verticillium dahliae[J]. PLoS One, 2012, 7(12):e51091.
doi: 10.1371/journal.pone.0051091 URL |
| [22] | 张坤. 棉花转录因子GhWRKY40-like和GhWRKY70在抗黄萎病中的功能研究[D]. 武汉:华中农业大学, 2015. |
| Zhang K. Functional analysis of GhWRKY40-like and GhWRKY70 in cotton responsive to Verticillium dahliae[D]. Wuhan:Huazhong Agricultural University, 2015. | |
| [23] | 雷建峰, 徐新霞, 李月, 等. CRISPR/Cas9介导靶向敲除拟南芥GGB基因突变体的鉴定[J]. 西北植物学报, 2016, 36(5):857-864. |
| Lei JF, Xu XX, Li Y, et al. Identification of GGB mutant caused by CRISPR/Cas9 in Arabidopsis[J]. Acta Bot Boreali Occidentalia Sin, 2016, 36(5):857-864. | |
| [24] |
Huang XY, Chao DY, Gao JP, et al. A previously unknown zinc finger protein, DST, regulates drought and salt tolerance in rice via stomatal aperture control[J]. Genes Dev, 2009, 23(15):1805-1817.
doi: 10.1101/gad.1812409 URL |
| [25] |
Yang S, Vanderbeld B, Wan J, et al. Narrowing down the targets:towards successful genetic engineering of drought-tolerant crops[J]. Mol Plant, 2010, 3(3):469-490.
doi: 10.1093/mp/ssq016 URL |
| [26] |
Li ZX, Kang J, Sui N, et al. ROP11 GTPase is a negative regulator of multiple ABA responses in Arabidopsis[J]. J Integr Plant Biol, 2012, 54(3):169-179.
doi: 10.1111/j.1744-7909.2012.01100.x URL |
| [27] |
Lemichez E, Wu Y, Sanchez JP, et al. Inactivation of AtRac1 by abscisic acid is essential for stomatal closure[J]. Genes Dev, 2001, 15(14):1808-1816.
doi: 10.1101/gad.900401 URL |
| [28] |
Li C, Lu H, Li W, et al. A ROP2-RIC1 pathway fine-tunes microtubule reorganization for salt tolerance in Arabidopsis[J]. Plant Cell Environ, 2017, 40(7):1127-1142.
doi: 10.1111/pce.12905 URL |
| [1] | 娄慧, 朱金成, 杨洋, 张薇. 抗、感品种棉花根系分泌物对尖孢镰刀菌生长及基因表达的影响[J]. 生物技术通报, 2023, 39(9): 156-167. |
| [2] | 吕秋谕, 孙培媛, 冉彬, 王佳蕊, 陈庆富, 李洪有. 苦荞转录因子基因FtbHLH3的克隆、亚细胞定位及表达分析[J]. 生物技术通报, 2023, 39(8): 194-203. |
| [3] | 王佳蕊, 孙培媛, 柯瑾, 冉彬, 李洪有. 苦荞糖基转移酶基因FtUGT143的克隆及表达分析[J]. 生物技术通报, 2023, 39(8): 204-212. |
| [4] | 孙明慧, 吴琼, 刘丹丹, 焦小雨, 王文杰. 茶树CsTMFs的克隆与表达分析[J]. 生物技术通报, 2023, 39(7): 151-159. |
| [5] | 赵雪婷, 高利燕, 王俊刚, 沈庆庆, 张树珍, 李富生. 甘蔗AP2/ERF转录因子基因ShERF3的克隆、表达及其编码蛋白的定位[J]. 生物技术通报, 2023, 39(6): 208-216. |
| [6] | 杨洋, 朱金成, 娄慧, 韩泽刚, 张薇. 海岛棉与枯萎病菌的互作转录组分析[J]. 生物技术通报, 2023, 39(6): 259-273. |
| [7] | 姜晴春, 杜洁, 王嘉诚, 余知和, 王允, 柳忠玉. 虎杖转录因子PcMYB2的表达特性和功能分析[J]. 生物技术通报, 2023, 39(5): 217-223. |
| [8] | 姚姿婷, 曹雪颖, 肖雪, 李瑞芳, 韦小妹, 邹承武, 朱桂宁. 火龙果溃疡病菌实时荧光定量PCR内参基因的筛选[J]. 生物技术通报, 2023, 39(5): 92-102. |
| [9] | 王艺清, 王涛, 韦朝领, 戴浩民, 曹士先, 孙威江, 曾雯. 茶树SMAS基因家族的鉴定及互作分析[J]. 生物技术通报, 2023, 39(4): 246-258. |
| [10] | 刘思佳, 王浩楠, 付宇辰, 闫文欣, 胡增辉, 冷平生. ‘西伯利亚’百合LiCMK基因克隆及功能分析[J]. 生物技术通报, 2023, 39(3): 196-205. |
| [11] | 王涛, 漆思雨, 韦朝领, 王艺清, 戴浩民, 周喆, 曹士先, 曾雯, 孙威江. CsPPR和CsCPN60-like在茶树白化叶片中的表达分析及互作蛋白验证[J]. 生物技术通报, 2023, 39(3): 218-231. |
| [12] | 庞强强, 孙晓东, 周曼, 蔡兴来, 张文, 王亚强. 菜心BrHsfA3基因克隆及其对高温胁迫的响应[J]. 生物技术通报, 2023, 39(2): 107-115. |
| [13] | 苗淑楠, 高宇, 李昕儒, 蔡桂萍, 张飞, 薛金爱, 季春丽, 李润植. 大豆GmPDAT1参与油脂合成和非生物胁迫应答的功能分析[J]. 生物技术通报, 2023, 39(2): 96-106. |
| [14] | 葛雯冬, 王腾辉, 马天意, 范震宇, 王玉书. 结球甘蓝PRX基因家族全基因组鉴定与逆境条件下的表达分析[J]. 生物技术通报, 2023, 39(11): 252-260. |
| [15] | 杨旭妍, 赵爽, 马天意, 白玉, 王玉书. 三个甘蓝WRKY基因的克隆及其对非生物胁迫的表达[J]. 生物技术通报, 2023, 39(11): 261-269. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||