







生物技术通报 ›› 2022, Vol. 38 ›› Issue (4): 163-173.doi: 10.13560/j.cnki.biotech.bull.1985.2021-1272
马馨馨( ), 许洋, 赵欢欢, 火兆燕, 王树彬, 钟凤林(
), 许洋, 赵欢欢, 火兆燕, 王树彬, 钟凤林( )
)
收稿日期:2021-10-09
出版日期:2022-04-26
发布日期:2022-05-06
通讯作者:
钟凤林,男,博士,教授,研究方向:蔬菜种质资源创制与新品种选育;E-mail: zhong591@fafu.edu.cn作者简介:马馨馨,女,硕士,研究方向:蔬菜育种与生物技术;E-mail: 1126691073@qq.com
基金资助:
MA Xin-xin( ), XU Yang, ZHAO Huan-huan, HUO Zhao-yan, WANG Shu-bin, ZHONG Feng-lin(
), XU Yang, ZHAO Huan-huan, HUO Zhao-yan, WANG Shu-bin, ZHONG Feng-lin( )
)
Received:2021-10-09
Published:2022-04-26
Online:2022-05-06
摘要:
探究番茄4CL基因家族的基本理化性质、分子进化特性和其在不同浓度氮素下的表达模式。对番茄4CL基因家族成员进行全基因组鉴定,采用生物信息学方法对其进行分析,并在不同浓度氮素处理下进行qPCR验证。在番茄中共鉴定出6个4CL基因家族成员,全部成员均为稳定蛋白,有5个成员呈酸性,编码的氨基酸数在538-957之间,二级结构以无规则卷曲为主,三级结构除Sl4CL04以5bsw.1.A为模型,其他成员均以5bsr.1.A为模型。番茄4CL基因家族内含子数量在5-11之间,12个Motif在Sl4CL中排序及位置都非常保守,具有较高的同源性。启动子顺式作用元件分析发现MYB作用元件的分布最多,在不同浓度氮素处理下,6个基因表达量均有显著差异。对Sl4CL03构建过表达载体,亚细胞定位结果显示位于过氧化物酶体、叶绿体和细胞质。番茄4CL家族进化具有保守性和特异性,能够在氮素处理时进行相关机理调节。
马馨馨, 许洋, 赵欢欢, 火兆燕, 王树彬, 钟凤林. 番茄4CL基因家族鉴定和氮素处理下的表达分析[J]. 生物技术通报, 2022, 38(4): 163-173.
MA Xin-xin, XU Yang, ZHAO Huan-huan, HUO Zhao-yan, WANG Shu-bin, ZHONG Feng-lin. Identification of Tomato 4CL Gene Family and Expression Analysis Under Nitrogen Treatment[J]. Biotechnology Bulletin, 2022, 38(4): 163-173.
| 类型 Type | 基因ID Gene ID | 基因名称Gene name | 正向引物Forward(5'-3') | 反向引物Reverse(5'-3') |
|---|---|---|---|---|
| RT-qPCR | Solyc03g097030 | Sl4CL01 | TTTGAGGAGAGGACATGTATTATCG | GACGATGCTAGAATGGTAAA |
| Solyc03g117870 | Sl4CL02 | ATGTTGACACACAAAGGCT | ATTGCTGCTCCGACTCTC | |
| Solyc06g068650 | Sl4CL03 | TGTCTTCCTTGGTGCGTC | GCATAATCCTTCACCTTGTTC | |
| Solyc08g076300 | Sl4CL04 | TAAGTCAGCAAGATACAGCG | GAGAGAAAGCCCATACACAT | |
| Solyc11g069050 | Sl4CL05 | TCAACCTATGAGTATCTTTGCTGC | AAAACCGTTGTGCCGTGC | |
| Solyc12g042460 | Sl4CL06 | AGTAAGGATAGTCATTTCTGGG | GCTAAGCACATTGTTATAGGTG | |
| Actin | GTCCTCTTCCAGCCATCCAT | ACCACTGAGCACAATGTTACCG | ||
| RT-PCR | Solyc06g068650 | Sl4CL03 | ATGACAAATGCAACGATGGAG | ATTTGGA TATCCAGCAG CT |
| pCAMBIA1302 | GTGGATTGATGTGATATCTCC | CTGACAGAAAATTTGTGCCC |
表1 PCR所用到的引物
Table 1 Primers for PCR
| 类型 Type | 基因ID Gene ID | 基因名称Gene name | 正向引物Forward(5'-3') | 反向引物Reverse(5'-3') |
|---|---|---|---|---|
| RT-qPCR | Solyc03g097030 | Sl4CL01 | TTTGAGGAGAGGACATGTATTATCG | GACGATGCTAGAATGGTAAA |
| Solyc03g117870 | Sl4CL02 | ATGTTGACACACAAAGGCT | ATTGCTGCTCCGACTCTC | |
| Solyc06g068650 | Sl4CL03 | TGTCTTCCTTGGTGCGTC | GCATAATCCTTCACCTTGTTC | |
| Solyc08g076300 | Sl4CL04 | TAAGTCAGCAAGATACAGCG | GAGAGAAAGCCCATACACAT | |
| Solyc11g069050 | Sl4CL05 | TCAACCTATGAGTATCTTTGCTGC | AAAACCGTTGTGCCGTGC | |
| Solyc12g042460 | Sl4CL06 | AGTAAGGATAGTCATTTCTGGG | GCTAAGCACATTGTTATAGGTG | |
| Actin | GTCCTCTTCCAGCCATCCAT | ACCACTGAGCACAATGTTACCG | ||
| RT-PCR | Solyc06g068650 | Sl4CL03 | ATGACAAATGCAACGATGGAG | ATTTGGA TATCCAGCAG CT |
| pCAMBIA1302 | GTGGATTGATGTGATATCTCC | CTGACAGAAAATTTGTGCCC |
| 基因名称Gene name | 氨基酸个数 Number of amino acids | 分子量 Molecular weight/kD | 等电点 Isoelectric point/pI | 不稳定指数Instability index | 总平均亲水系数Grand average of hydropathicity | 原子总数Total number of atoms | 亚细胞定位Subcellular localization |
|---|---|---|---|---|---|---|---|
| Sl4CL01 | 570 | 62080.54 | 5.34 | 34.92 | 0.106 | 8820 | Peroxisome Chloroplast |
| Sl4CL02 | 699 | 77723.64 | 5.64 | 37.7 | -0.126 | 10926 | Peroxisome |
| Sl4CL03 | 560 | 61700.2 | 5.49 | 33.69 | -0.028 | 8735 | Peroxisome |
| Sl4CL04 | 957 | 102255.02 | 5.45 | 31.47 | 0.476 | 14550 | Peroxisome |
| Sl4CL05 | 538 | 58930.46 | 8.49 | 30.69 | 0.07 | 8369 | Peroxisome |
| Sl4CL06 | 548 | 60394.02 | 5.9 | 32.36 | 0.045 | 8590 | Peroxisome |
表2 番茄4CL基因家族蛋白理化性质
Table 2 Physico-chemical properties of 4CL family proteins in tomato(Solanum lycopersicum)
| 基因名称Gene name | 氨基酸个数 Number of amino acids | 分子量 Molecular weight/kD | 等电点 Isoelectric point/pI | 不稳定指数Instability index | 总平均亲水系数Grand average of hydropathicity | 原子总数Total number of atoms | 亚细胞定位Subcellular localization |
|---|---|---|---|---|---|---|---|
| Sl4CL01 | 570 | 62080.54 | 5.34 | 34.92 | 0.106 | 8820 | Peroxisome Chloroplast |
| Sl4CL02 | 699 | 77723.64 | 5.64 | 37.7 | -0.126 | 10926 | Peroxisome |
| Sl4CL03 | 560 | 61700.2 | 5.49 | 33.69 | -0.028 | 8735 | Peroxisome |
| Sl4CL04 | 957 | 102255.02 | 5.45 | 31.47 | 0.476 | 14550 | Peroxisome |
| Sl4CL05 | 538 | 58930.46 | 8.49 | 30.69 | 0.07 | 8369 | Peroxisome |
| Sl4CL06 | 548 | 60394.02 | 5.9 | 32.36 | 0.045 | 8590 | Peroxisome |
| 基因名称 Gene name | α-螺旋α-helix/% | 延伸链 Extended strand/% | β-转角 β-turn/% | 无规则卷曲 Random coil/% |
|---|---|---|---|---|
| Sl4CL01 | 32.28 | 18.6 | 6.84 | 42.28 |
| Sl4CL02 | 33.19 | 18.6 | 9.01 | 39.2 |
| Sl4CL03 | 30.89 | 20.71 | 7.5 | 40.89 |
| Sl4CL04 | 41.69 | 15.57 | 7.94 | 34.8 |
| Sl4CL05 | 30.67 | 20.07 | 8.36 | 40.89 |
| Sl4CL06 | 31.93 | 19.53 | 8.58 | 39.96 |
表3 番茄4CL基因家族二级结构
Table 3 Secondary structure of tomato 4CL gene family
| 基因名称 Gene name | α-螺旋α-helix/% | 延伸链 Extended strand/% | β-转角 β-turn/% | 无规则卷曲 Random coil/% |
|---|---|---|---|---|
| Sl4CL01 | 32.28 | 18.6 | 6.84 | 42.28 |
| Sl4CL02 | 33.19 | 18.6 | 9.01 | 39.2 |
| Sl4CL03 | 30.89 | 20.71 | 7.5 | 40.89 |
| Sl4CL04 | 41.69 | 15.57 | 7.94 | 34.8 |
| Sl4CL05 | 30.67 | 20.07 | 8.36 | 40.89 |
| Sl4CL06 | 31.93 | 19.53 | 8.58 | 39.96 |
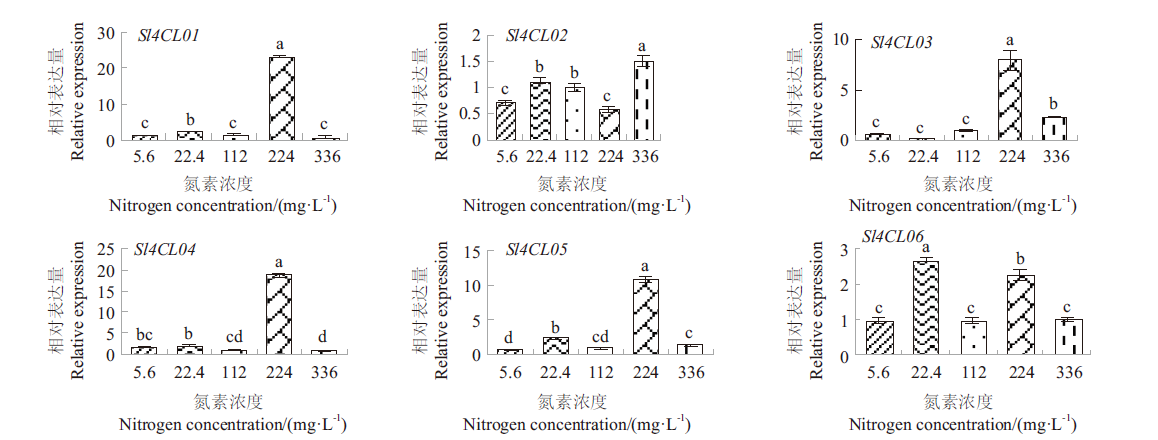
图6 番茄4CL基因在不同浓度氮素下的表达模式 不同柱上不同字母表示显著差异(P<0.05)
Fig.6 Expression patterns of tomato 4CL genes under different concentrations of nitrogen Histograms with different letters indicate significant difference at the 0.05 level
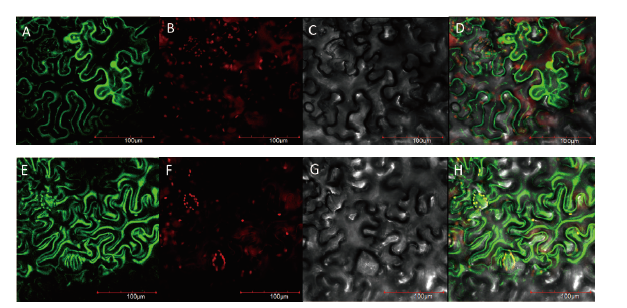
图8 Sl4CL03蛋白在烟草叶片细胞的亚细胞定位 A-D:分别为pCAMBIA1302空载的荧光通道、叶绿体荧光通道、明场、叠加图;E-H:分别为Sl4CL03蛋白的荧光通道、叶绿体荧光通道、明场、叠加图
Fig.8 Subcellular localization of Sl4CL03 in Nicotiana tabacum leaf cells A-D:pCAMBIA1302 empty vector fluorescence,chloroplast fluorescence,bright,and merged.E-H:Sl4CL03 protein fluorescence,chloroplast fluorescence,bright,and merged
| [1] | 杜红豆. 氮素对飞机草(Eupatorium odoratum)4CL 酶活性和基因表达影响的研究[D]. 哈尔滨:哈尔滨师范大学, 2012. |
| Du HD. Influence of nitrogen on activity of 4-coumarate:coenzyme a ligase(4CL)and its gene expression in Eupatorium odoratum[D]. Harbin:Harbin Normal University, 2012. | |
| [2] | 李晓瑞. 干旱胁迫下小麦苯丙烷类代谢及MYB转录因子TaMpc1-D4响应机制研究[D]. 杨凌:西北农林科技大学, 2020. |
| Li XR. The mechanisms of the phenylpropanoid pathway and MYB transcription factor TaMpc1-D4 in responses to drought stress in wheat[D]. Yangling:Northwest A&F University, 2020. | |
| [3] | 常胜合, 孙威, 许桂莺, 等. 利用4-香豆酸:CoA连接酶基因Mu4CL15提高香蕉植株的倒伏抗性[J]. 分子植物育种, 2017, 15(12):4905-4911. |
| Chang SH, Sun W, Xu GY, et al. Enhancing lodging resistance of banana plant by transforming 4-coumarate:CoA ligase gene Mu4CL15[J]. Mol Plant Breed, 2017, 15(12):4905-4911. | |
| [4] |
Sun SC, Xiong XP, Zhang XL, et al. Correction to:Characterization of the Gh4CL gene family reveals a role of Gh4CL7 in drought tolerance[J]. BMC Plant Biol, 2021, 21(1):65.
doi: 10.1186/s12870-021-02834-9 URL |
| [5] | Zhang MY, Wang DJ, Gao XX, et al. Exogenous caffeic acid and epicatechin enhance resistance against Botrytis cinerea through activation of the phenylpropanoid pathway in apples[J]. Sci Hortic, 2020, 268:109348. |
| [6] | Zhang CH, Ma T, Luo WC, et al. Identification of 4CL genes in desert poplars and their changes in expression in response to salt stress[J]. Genes:Basel, 2015, 6(3):901-917. |
| [7] |
Wei HY, Rao GD, Wang YK, et al. Cloning and analysis of a new 4CL-like gene in Populus tomentosa[J]. For Sci Pract, 2013, 15(2):98-104.
doi: 10.1007/s11632-013-0204-z URL |
| [8] | Liu XY, Cui XM, Ji DC, et al. Luteolin-induced activation of the phenylpropanoid metabolic pathway contributes to quality maintenance and disease resistance of sweet cherry[J]. Food Chem, 2021, 342:128309. |
| [9] |
Dong NQ, Lin HX. Contribution of phenylpropanoid metabolism to plant development and plant-environment interactions[J]. J Integr Plant Biol, 2021, 63(1):180-209.
doi: 10.1111/jipb.13054 URL |
| [10] |
Lavhale SG, Kalunke RM, Giri AP. Structural, functional and evolutionary diversity of 4-coumarate-CoA ligase in plants[J]. Planta, 2018, 248(5):1063-1078.
doi: 10.1007/s00425-018-2965-z URL |
| [11] | Li SS, Li Y, Sun LL, et al. Identification and expression analysis of 4-Coumarate:Coenzyme A ligase gene family in Dryopteris Fragrans[J]. Cell Mol Biol:Noisy-Le-Grand, 2015, 61(4):25-33. |
| [12] |
Badhan S, Ball AS, Mantri N. First report of CRISPR/Cas9 mediated DNA-free editing of 4CL and RVE7 genes in chickpea protoplasts[J]. Int J Mol Sci, 2021, 22(1):396.
doi: 10.3390/ijms22010396 URL |
| [13] |
王惠, 张顺斌, 金贺, 等. 4-香豆酸辅酶A连接酶响应大豆孢囊线虫胁迫的潜在功能[J]. 生物技术通报, 2021, 37(7):71-80.
doi: 10.13560/j.cnki.biotech.bull.1985.2021-0378 |
| Wang H, Zhang SB, Jin H, et al. Potential function of 4-coumaric acid-CoA ligase in response to soybean cyst nematode stress[J]. Biotechnol Bull, 2021, 37(7):71-80. | |
| [14] | 杨克彬, 单雪萌, 史晶晶, 等. 毛竹4-香豆酸辅酶A连接酶基因家族鉴定及表达分析[J]. 核农学报, 2021, 35(1):72-82. |
| Yang KB, Shan XM, Shi JJ, et al. Identification and expression analysis of 4CL gene family in Phyllostachys edulis[J]. J Nucl Agric Sci, 2021, 35(1):72-82. | |
| [15] | 洪平静, 徐小萍, 王静宇, 等. 龙眼体胚发生早期4CL基因家族鉴定与功能分析[J]. 热带作物学报, 2021, 42(4):909-919. |
| Hong PJ, Xu XP, Wang JY, et al. Identification and functional analysis of 4CL gene family during early somatic embryogenesis in Dimocarpus longan lour[J]. Chin J Trop Crop, 2021, 42(4):909-919. | |
| [16] | 唐映红. 苎麻4CL和CCR基因家族成员的克隆、表达及功能研究[D]. 长沙:湖南农业大学, 2018. |
| Tang YH. Study on the cloning, expression and functional of Boehmeria nivea 4CL and CCR gene family members[D]. Changsha:Hunan Agricultural University, 2018. | |
| [17] | 申晚霞, 王志彬, 薛杨, 等. 柑橘4CL基因家族的结构及其功能分析[J]. 园艺学报, 2019, 46(6):1068-1078. |
| Shen WX, Wang ZB, Xue Y, et al. Characterization of 4-coumarate:CoA ligase(4CL)gene family in Citrus[J]. Acta Hortic Sin, 2019, 46(6):1068-1078. | |
| [18] | 陈夏晔, 彭宣翔. 普通烟草Nt4CL基因家族的生物信息学分析[J]. 贵州农业科学, 2018, 46(8):11-15. |
| Chen XY, Peng XX. Bioinformatics analysis on tobacco Nt4CL gene family[J]. Guizhou Agric Sci, 2018, 46(8):11-15. | |
| [19] | 曹运鹏, 方志, 李姝妹, 等. 砀山酥梨4CL基因家族的全基因组鉴定与分析[J]. 遗传, 2015, 37(7):711-719. |
| Cao YP, Fang Z, Li SM, et al. Genome-wide identification and analyses of 4CL gene families in Pyrus bretschneideri Rehd[J]. Hereditas, 2015, 37(7):711-719. | |
| [20] |
Meng J, Li CN, Zhao ML, et al. Lignin biosynjournal regulated by the antisense 4CL gene in alfalfa[J]. Czech J Genet Plant Breed, 2018, 54(No. 1):26-29.
doi: 10.17221/23/2017-CJGPB URL |
| [21] | 孙士超. 绿色棉纤维转录组分析及4CL基因家族的鉴定[D]. 石河子:石河子大学, 2020. |
| Sun SC. Transcriptome analysis of green cotton fiber and identification of 4CL gene family[D]. Shihezi:Shihezi University, 2020. | |
| [22] | 杨婷, 潘翔, 饶国栋, 等. 植物4CL基因家族结构功能与表达特性研究进展[J]. 成都大学学报:自然科学版, 2011, 30(1):4-7. |
| Yang T, Pan X, Rao GD, et al. Research progress in structural function and expression characteristic of 4CL’s gene family in plants[J]. J Chengdu Univ:Nat Sci Ed, 2011, 30(1):4-7. | |
| [23] |
田晓明, 颜立红, 向光锋, 等. 植物4香豆酸:辅酶A连接酶研究进展[J]. 生物技术通报, 2017, 33(4):19-26.
doi: 10.13560/j.cnki.biotech.bull.1985.2017.04.003 |
| Tian XM, Yan LH, Xiang GF, et al. Research progress on 4-coumarate:coenzyme A ligase(4CL)in plants[J]. Biotechnol Bull, 2017, 33(4):19-26. | |
| [24] | 乔中全, 王晓明, 曾慧杰, 等. 灰毡毛忍冬Lm4CL基因克隆及表达分析[J]. 中南林业科技大学学报, 2021, 41(5):122-132. |
| Qiao ZQ, Wang XM, Zeng HJ, et al. Clone and expression analysis of Lm4CL in Lonicera macranthoides Hand-Mazz[J]. J Central South Univ For Technol, 2021, 41(5):122-132. | |
| [25] |
Sun H, Guo K, Feng S, et al. Positive selection drives adaptive diversification of the 4-coumarate:CoA ligase(4CL)gene in angiosperms[J]. Ecol Evol, 2015, 5(16):3413-3420.
doi: 10.1002/ece3.1613 URL |
| [26] | 汤崴, 白云赫, 胡雨晴, 等. 葡萄木质素相关基因PAL、4CL和CAD特征鉴定及其应答赤霉素在果实发育过程中的作用[J]. 南京农业大学学报, 2019, 42(3):430-439. |
| Tang W, Bai YH, Hu YQ, et al. Characterization of grape lignin-related genes PAL, 4CL, CAD and the role of GA in the berry development[J]. J Nanjing Agric Univ, 2019, 42(3):430-439. | |
| [27] | 王星淇, 孙雪丽, 车婧如, 等. 两个非洲菊4CL基因的克隆及表达[J]. 应用与环境生物学报, 2020, 26(2):272-279. |
| Wang XQ, Sun XL, Che JR, et al. Cloning and expression analysis of two 4CL genes in Gerbera[J]. Chin J Appl Environ Biol, 2020, 26(2):272-279. | |
| [28] |
Park JJ, Yoo CG, Flanagan A, et al. Defined Tetra-allelic gene disruption of the 4-coumarate:coenzyme A ligase 1(Pv4CL1)gene by CRISPR/Cas9 in switchgrass results in lignin reduction and improved sugar release[J]. Biotechnol Biofuels, 2017, 10:284.
doi: 10.1186/s13068-017-0972-0 URL |
| [29] | Muna Alariqi. 陆地棉基因GhLDOX和Gh4CL3的功能解析[D]. 武汉:华中农业大学, 2020. |
| Muna Alariqi. Functional studies of GhLDOX & Gh4CL3 genes in upland cotton Gossypium hirsutum[D]. Wuhan:Huazhong Agricultural University, 2020. | |
| [30] | 熊显鹏. Gh4CL30和GhWRKY70D13在棉花抗黄萎病中的功能研究[D]. 石河子:石河子大学, 2020. |
| Xiong XP. Functional analysis of Gh4CL30 and GhWRKY70D13 in Cotton Resistance to Verticillium Wilt[D]. Shihezi:Shihezi University, 2020. | |
| [31] |
Kienow L, Schneider K, Bartsch M, et al. Jasmonates meet fatty acids:functional analysis of a new acyl-coenzyme A synthetase family from Arabidopsis thaliana[J]. J Exp Bot, 2008, 59(2):403-419.
doi: 10.1093/jxb/erm325 pmid: 18267944 |
| [1] | 王佳蕊, 孙培媛, 柯瑾, 冉彬, 李洪有. 苦荞糖基转移酶基因FtUGT143的克隆及表达分析[J]. 生物技术通报, 2023, 39(8): 204-212. |
| [2] | 刘珍银, 段郅臻, 彭婷, 王童欣, 王健. 基于三角梅的病毒诱导基因沉默体系的建立与优化[J]. 生物技术通报, 2023, 39(7): 123-130. |
| [3] | 孙明慧, 吴琼, 刘丹丹, 焦小雨, 王文杰. 茶树CsTMFs的克隆与表达分析[J]. 生物技术通报, 2023, 39(7): 151-159. |
| [4] | 赵雪婷, 高利燕, 王俊刚, 沈庆庆, 张树珍, 李富生. 甘蔗AP2/ERF转录因子基因ShERF3的克隆、表达及其编码蛋白的定位[J]. 生物技术通报, 2023, 39(6): 208-216. |
| [5] | 姜晴春, 杜洁, 王嘉诚, 余知和, 王允, 柳忠玉. 虎杖转录因子PcMYB2的表达特性和功能分析[J]. 生物技术通报, 2023, 39(5): 217-223. |
| [6] | 史建磊, 宰文珊, 苏世闻, 付存念, 熊自立. 番茄青枯病抗性相关miRNA的鉴定与表达分析[J]. 生物技术通报, 2023, 39(5): 233-242. |
| [7] | 车永梅, 郭艳苹, 刘广超, 叶青, 李雅华, 赵方贵, 刘新. 菌株C8和B4的分离鉴定及其耐盐促生效果和机制[J]. 生物技术通报, 2023, 39(5): 276-285. |
| [8] | 姚姿婷, 曹雪颖, 肖雪, 李瑞芳, 韦小妹, 邹承武, 朱桂宁. 火龙果溃疡病菌实时荧光定量PCR内参基因的筛选[J]. 生物技术通报, 2023, 39(5): 92-102. |
| [9] | 胡明月, 杨宇, 郭仰东, 张喜春. 低温胁迫下番茄SlMYB96的功能分析[J]. 生物技术通报, 2023, 39(4): 236-245. |
| [10] | 王艺清, 王涛, 韦朝领, 戴浩民, 曹士先, 孙威江, 曾雯. 茶树SMAS基因家族的鉴定及互作分析[J]. 生物技术通报, 2023, 39(4): 246-258. |
| [11] | 刘思佳, 王浩楠, 付宇辰, 闫文欣, 胡增辉, 冷平生. ‘西伯利亚’百合LiCMK基因克隆及功能分析[J]. 生物技术通报, 2023, 39(3): 196-205. |
| [12] | 王涛, 漆思雨, 韦朝领, 王艺清, 戴浩民, 周喆, 曹士先, 曾雯, 孙威江. CsPPR和CsCPN60-like在茶树白化叶片中的表达分析及互作蛋白验证[J]. 生物技术通报, 2023, 39(3): 218-231. |
| [13] | 申云鑫, 施竹凤, 周旭东, 李铭刚, 张庆, 冯路遥, 陈齐斌, 杨佩文. 三株具生防功能芽孢杆菌的分离鉴定及其生物活性研究[J]. 生物技术通报, 2023, 39(3): 267-277. |
| [14] | 庞强强, 孙晓东, 周曼, 蔡兴来, 张文, 王亚强. 菜心BrHsfA3基因克隆及其对高温胁迫的响应[J]. 生物技术通报, 2023, 39(2): 107-115. |
| [15] | 苗淑楠, 高宇, 李昕儒, 蔡桂萍, 张飞, 薛金爱, 季春丽, 李润植. 大豆GmPDAT1参与油脂合成和非生物胁迫应答的功能分析[J]. 生物技术通报, 2023, 39(2): 96-106. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||