







生物技术通报 ›› 2024, Vol. 40 ›› Issue (9): 198-211.doi: 10.13560/j.cnki.biotech.bull.1985.2024-0269
吴慧琴( ), 王延宏, 刘涵, 司政, 刘雪晴, 王静, 阳宜, 成妍(
), 王延宏, 刘涵, 司政, 刘雪晴, 王静, 阳宜, 成妍( )
)
收稿日期:2024-03-19
出版日期:2024-09-26
发布日期:2024-10-12
通讯作者:
成妍,女,博士,副研究员,研究方向:辣椒栽培育种;E-mail: chengyan820620@163.com作者简介:吴慧琴,女,硕士研究生,研究方向:辣椒遗传育种与分子生物学;E-mail: 1993148688@qq.com
基金资助:
WU Hui-qin( ), WANG Yan-hong, LIU Han, SI Zheng, LIU Xue-qing, WANG Jing, YANG Yi, CHENG Yan(
), WANG Yan-hong, LIU Han, SI Zheng, LIU Xue-qing, WANG Jing, YANG Yi, CHENG Yan( )
)
Received:2024-03-19
Published:2024-09-26
Online:2024-10-12
摘要:
【目的】鉴定分析辣椒UGT基因家族成员的结构和功能,为解析辣椒多糖糖基化分子机制奠定基础。【方法】利用BioEdit、BLASTP和Pfam比对搜索辣椒UGT成员;利用CDD和HMMER数据库验证保守结构域;利用ExPASy、Cell PLOC、MAGA X、MG2C、GSDS、STRING等分析预测蛋白理化性质、系统发育、染色体定位、基因结构和蛋白互作;利用转录组数据和实时荧光定量PCR分析辣椒UGT基因在各器官发育过程中的表达模式。【结果】从辣椒基因家族中鉴定到的140个辣椒UGT家族成员,编码的氨基酸数量介于253-534之间,等电点在4.7-8.45之间;CaUGTs蛋白大多数定位于细胞外,少部分定位于质膜和叶绿体中;分布在17个组中,定位于12条染色体上,其中25个成员定位在第12条染色体(Chromosome 12,chr.12);所有成员均包含保守结构域motif 1、motif 3;响应与植物生长发育、胁迫有关的作用元件。辣椒UGT基因在不同组织、逆境胁迫及激素应答下的表达模式分析表明,UGT基因具有组织特异性表达差异,在花、果实发育阶段表达相对较高;在ABA、GA3、高温以及低温胁迫条件下,表达被显著诱导增加或者降低,在发育的特定阶段CaUGTs成员可能发挥不同的作用,很可能参与了调控辣椒逆境胁迫条件下的防御应答反应。【结论】140个辣椒UGT成员在分布及结构上存在多样性,而组内成员之间高度相似,CaUGTs基因可能在辣椒植株生长发育过程中响应非生物胁迫。
吴慧琴, 王延宏, 刘涵, 司政, 刘雪晴, 王静, 阳宜, 成妍. 辣椒UGT基因家族的鉴定及表达分析[J]. 生物技术通报, 2024, 40(9): 198-211.
WU Hui-qin, WANG Yan-hong, LIU Han, SI Zheng, LIU Xue-qing, WANG Jing, YANG Yi, CHENG Yan. Identification and Expression Analysis of UGT Gene Family in Pepper[J]. Biotechnology Bulletin, 2024, 40(9): 198-211.
| 基因 Gene | 正向引物Forward primer(5'-3') | 正向引物Forward primer(5'-3') |
|---|---|---|
| CaUGT31 | GCTACAAAGCTGATGCAGGC | GCGAAGACACTCGTACCGAA |
| CaUGT39 | AAGGAGGCCATCGACACAAG | TAGGCAGGATCAAGCTGTGC |
| CaUGT47 | GGGCCTTCTTCCACGAATTT | CGATCCCAAGTCCTCCAAGTT |
| CaUGT53 | GGTTCTTGACTCACAGCGGA | GCGAAAAATGGCCAGCAGAT |
| CaUGT63 | TTAGCTTTGGGAGTGTCGCC | ATCCGCGGGAATCTTGGTTT |
| CaUGT74 | GCTCCTCTTCCCTCTTCCAA | CAAACCGGCGAGACAAAAGA |
| CaUGT82 | CTGAGATGGCATGGGGACTT | GGCAAATTTACAACGTCGTCC |
| CaUGT92 | TGCCGTGTCAATTCGAAAGG | GCAACTTCAACAGCCCATGA |
| CaUGT127 | ATGTCCAAACCCAACTTCACA | CACGGCAGCACCAAAAGTTA |
| CaUGT128 | CCAATGAATGCCAGGTTGGTAG | TGGCCCTCAAATTTTCCCCCAT |
| CaUGT132 | GCCCCTCAAGCAACAATTCT | CACTGGTTGGTCAATGTGCA |
| CaActin | GGTGACGAGGCTCAATCCAA | CTCTGGAGCCACACGAAGTT |
表1 CaUGTs家族的RT-qPCR引物序列
Table 1 RT-qPCR primer sequences of CaUGTs
| 基因 Gene | 正向引物Forward primer(5'-3') | 正向引物Forward primer(5'-3') |
|---|---|---|
| CaUGT31 | GCTACAAAGCTGATGCAGGC | GCGAAGACACTCGTACCGAA |
| CaUGT39 | AAGGAGGCCATCGACACAAG | TAGGCAGGATCAAGCTGTGC |
| CaUGT47 | GGGCCTTCTTCCACGAATTT | CGATCCCAAGTCCTCCAAGTT |
| CaUGT53 | GGTTCTTGACTCACAGCGGA | GCGAAAAATGGCCAGCAGAT |
| CaUGT63 | TTAGCTTTGGGAGTGTCGCC | ATCCGCGGGAATCTTGGTTT |
| CaUGT74 | GCTCCTCTTCCCTCTTCCAA | CAAACCGGCGAGACAAAAGA |
| CaUGT82 | CTGAGATGGCATGGGGACTT | GGCAAATTTACAACGTCGTCC |
| CaUGT92 | TGCCGTGTCAATTCGAAAGG | GCAACTTCAACAGCCCATGA |
| CaUGT127 | ATGTCCAAACCCAACTTCACA | CACGGCAGCACCAAAAGTTA |
| CaUGT128 | CCAATGAATGCCAGGTTGGTAG | TGGCCCTCAAATTTTCCCCCAT |
| CaUGT132 | GCCCCTCAAGCAACAATTCT | CACTGGTTGGTCAATGTGCA |
| CaActin | GGTGACGAGGCTCAATCCAA | CTCTGGAGCCACACGAAGTT |
| 项目 Item | 蛋白长度 Protein length/aa | 分子量 Molecular weight/kD | 等电点 pI | 不稳定指数 Instability index | 疏水指数 GRAVY | 带负电残基数 Asp + Glu | 带正电残基数 Arg + Lys |
|---|---|---|---|---|---|---|---|
| 分布范围 | 253-534 | 20.71×103-59.65×103 | 4.7-8.45 | 26.52-59.99 | -0.334-0.054 | 29-73 | 20-64 |
| 平均值 | 450 | 50.61×103 | 5.79 | 43.35 | -0.139 | 55.53 | 45.86 |
| 亲水蛋白数 | — | — | — | — | 131 | — | — |
| 疏水蛋白数 | — | — | — | — | 9 | — | — |
| 酸性蛋白数 | — | — | 134 | — | — | — | — |
| 碱性蛋白数 | — | — | 6 | — | — | — | — |
| 稳定蛋白 | — | — | — | 40 | — | — | — |
| 不稳定蛋白 | — | — | — | 100 | — | — | — |
| 最小值成员 | CaUGT43 | CaUGT43 | CaUGT101 | CaUGT102 | CaUGT67 | CaUGT15 | CaUGT137 |
| 最大值成员 | CaUGT99 | CaUGT99 | CaUGT137 | CaUGT61 | CaUGT49 | CaUGT91 | CaUGT49 |
表2 CaUGTs蛋白理化性质
Table 2 Physical and chemical properties of CaUGTs protein
| 项目 Item | 蛋白长度 Protein length/aa | 分子量 Molecular weight/kD | 等电点 pI | 不稳定指数 Instability index | 疏水指数 GRAVY | 带负电残基数 Asp + Glu | 带正电残基数 Arg + Lys |
|---|---|---|---|---|---|---|---|
| 分布范围 | 253-534 | 20.71×103-59.65×103 | 4.7-8.45 | 26.52-59.99 | -0.334-0.054 | 29-73 | 20-64 |
| 平均值 | 450 | 50.61×103 | 5.79 | 43.35 | -0.139 | 55.53 | 45.86 |
| 亲水蛋白数 | — | — | — | — | 131 | — | — |
| 疏水蛋白数 | — | — | — | — | 9 | — | — |
| 酸性蛋白数 | — | — | 134 | — | — | — | — |
| 碱性蛋白数 | — | — | 6 | — | — | — | — |
| 稳定蛋白 | — | — | — | 40 | — | — | — |
| 不稳定蛋白 | — | — | — | 100 | — | — | — |
| 最小值成员 | CaUGT43 | CaUGT43 | CaUGT101 | CaUGT102 | CaUGT67 | CaUGT15 | CaUGT137 |
| 最大值成员 | CaUGT99 | CaUGT99 | CaUGT137 | CaUGT61 | CaUGT49 | CaUGT91 | CaUGT49 |
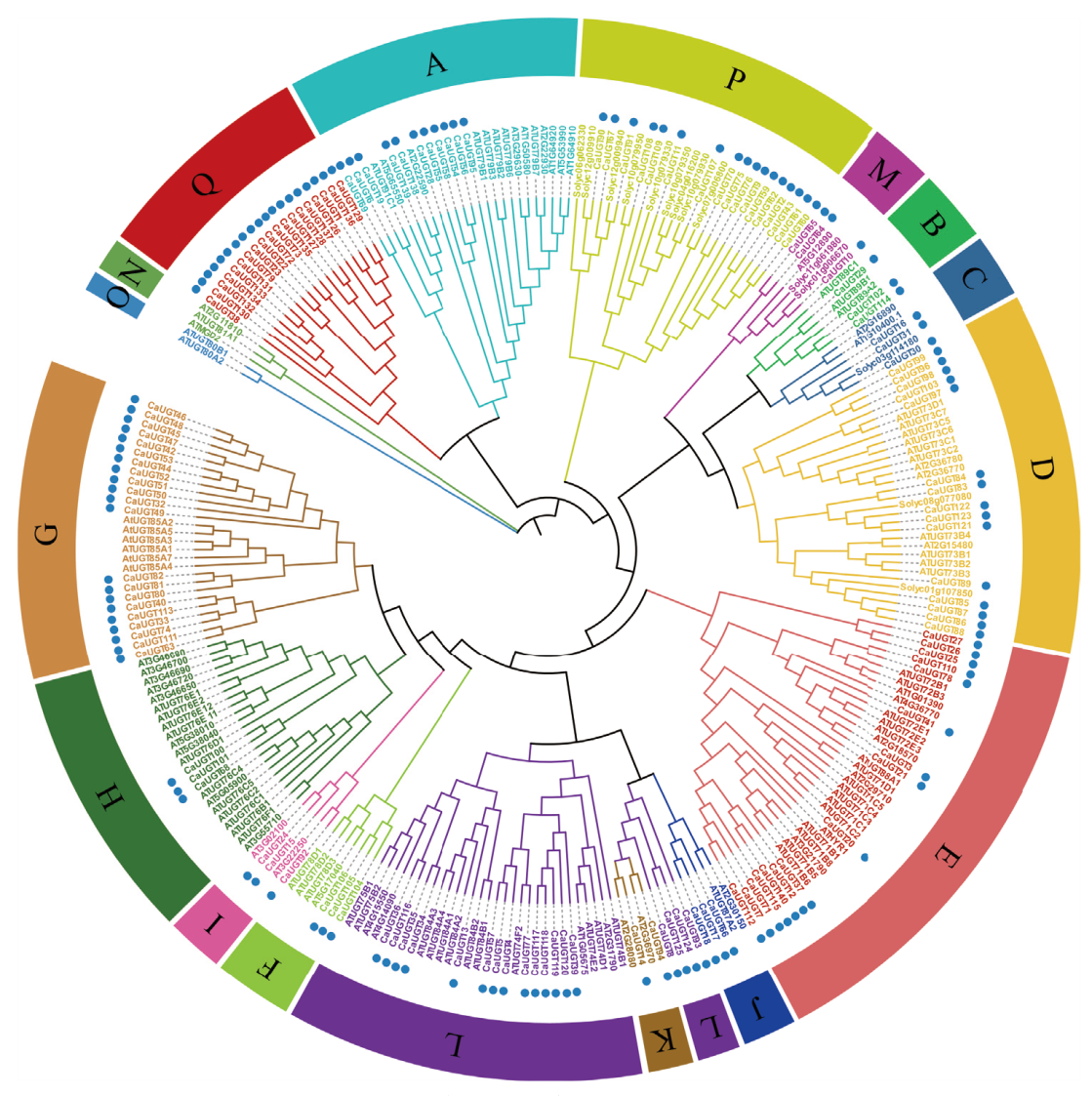
图1 拟南芥(112)、辣椒(140)、番茄(14)UGTs系统发育进化树 蓝色圆点表示辣椒UGT成员
Fig. 1 Phylogenetic tree of Arabidopsis thaliana(112), pepper(140), tomato(14)UGTs The blue dots indicate pepper UGTS
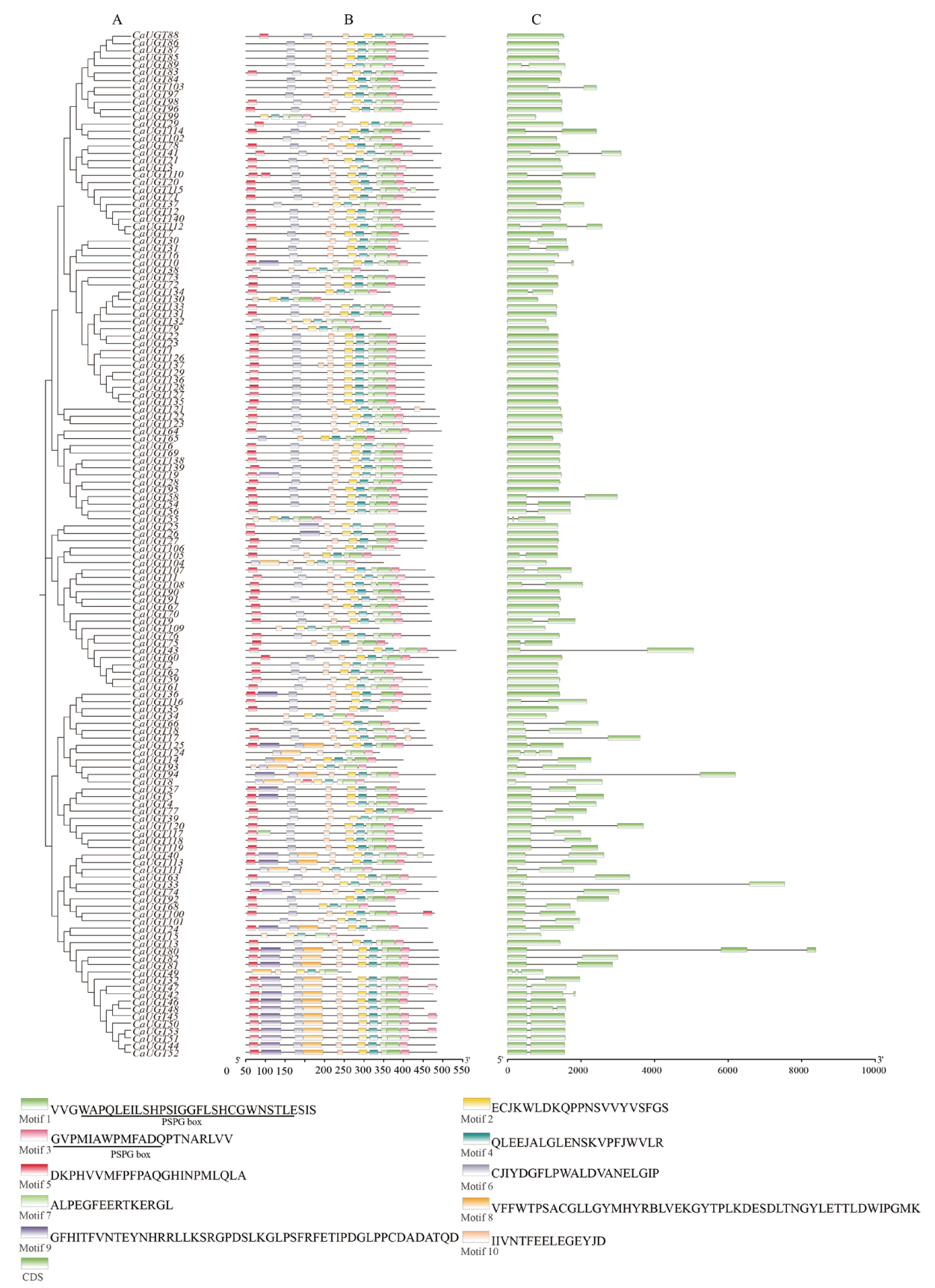
图2 辣椒CaUGTs家族成员系统进化树、基因结构及motif分布 A:系统进化树;B:保守结构域;C:基因结构(外显子/内含子)
Fig. 2 Phylogngntic tree, gene structure and motif distribution of the pepper CaUGTs family member A: Phylogenetic tree; B: conserved domain composition; C: gene structure(exon/intron)

图7 辣椒UGT家族在不同器官中的表达分析 L:叶;F:花;G:果实;ST:种子和胎座;S:种子;T:胎座
Fig. 7 Expression analysis of UGT genes in different organs L: Leaf; F: flower; G: fruit; ST: seed and placenta; S: seed; T: placenta
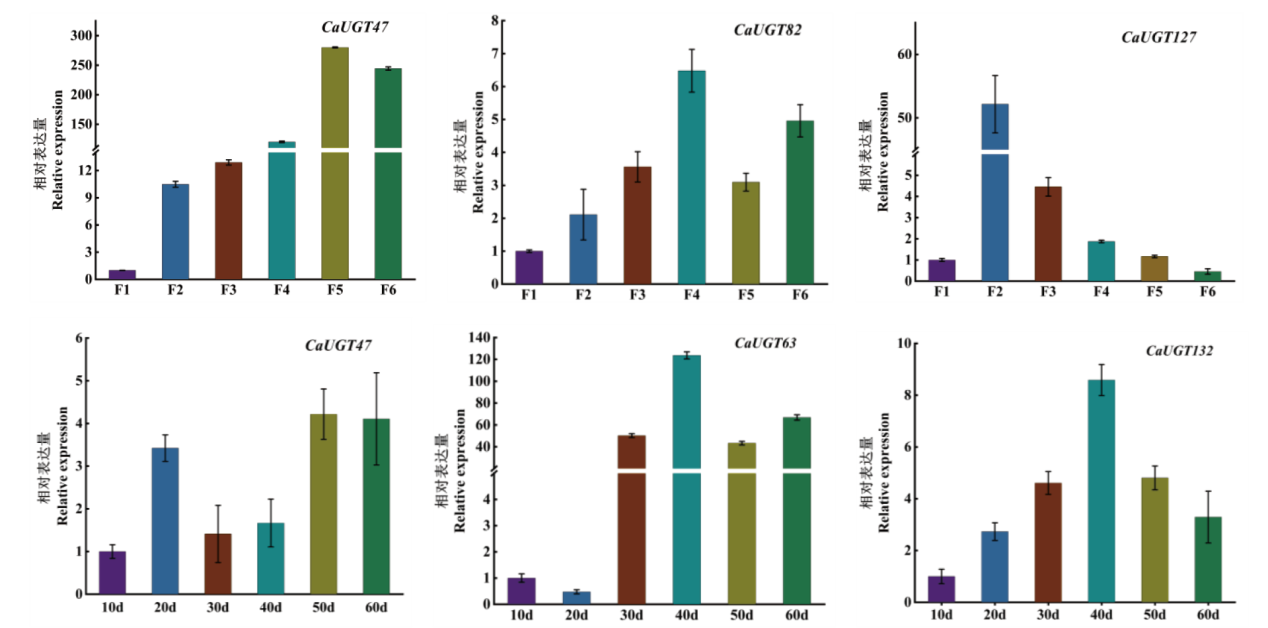
图8 CaUGTs在花、果实不同发育时期的表达分析 F1、F2、F3、F4、F5、F5、F6分别代表现蕾后1、3、5、7、8、9 d
Fig. 8 Expression analysis of CaUGTs at different developmental stages of flowers and fruits F1, F2, F3, F4, F5, F5, and F6 indicate 1, 3, 5, 7, 8 and 9 d respectively
| [1] | 吴永红, 周书栋, 李雪峰, 等. 2019年辣椒科学研究进展[J]. 辣椒杂志, 2020, 18(2): 1-7, 13. |
| Wu YH, Zhou SD, Li XF, et al. The research progresses on pepper in 2019[J]. J China Capsicum, 2020, 18(2): 1-7, 13. | |
| [2] | 陶兴林, 朱惠霞, 王晓巍, 等. 5个不同产区地理标志辣椒营养成分分析[J]. 食品安全质量检测学报, 2023, 14(3): 303-310. |
| Tao XL, Zhu HX, Wang XW, et al. Nutrient composition analysis of different geographical indications peppers in 5 different production areas[J]. J Food Saf Qual, 2023, 14(3): 303-310. | |
| [3] | 王楠艺, 付文婷, 吴迪, 等. 辣椒品质研究进展[J]. 江苏农业科学, 2022, 50(16): 21-27. |
| Wang NY, Fu WT, Wu D, et al. Research progress on pepper quality[J]. Jiangsu Agric Sci, 2022, 50(16): 21-27. | |
| [4] | McKay S, Oranje P, Helin J, et al. Development of an affordable, sustainable and efficacious plant-based immunomodulatory food ingredient based on bell pepper or carrot RG-I pectic polysaccharides[J]. Nutrients, 2021, 13(3): 963. |
| [5] | 武晓艳, 叶钰怡, 任文凯, 等. 植物多糖研究进展: 功能活性及潜在机制[J]. 中国科学: 生命科学, 2023, 53(6): 808-824. |
| Wu XY, Ye YY, Ren WK, et al. Research progress of plant polysaccharides: functional activities and potential mechanisms[J]. Sci Sin Vitae, 2023, 53(6): 808-824. | |
| [6] | 刘思扬, 陆雅琦, 海日汉, 等. 功能性植物多糖及其应用研究进展[J]. 食品工业科技, 2022, 43(21): 444-453. |
| Liu SY, Lu YQ, Hai RH, et al. Research progress on plant functional polysaccharides and its application[J]. Sci Technol Food Ind, 2022, 43(21): 444-453. | |
| [7] | 吴一凡, 张睿琪, 董泽安, 等. 植物糖基转移酶的分类、分布及在果实发育中作用研究进展[J]. 食品科技, 2021, 46(7): 1-6. |
| Wu YF, Zhang RQ, Dong ZA, et al. Classification and distribution of glycosyltransferases in plant and its roles in the fruit development[J]. Food Sci Technol, 2021, 46(7): 1-6. | |
| [8] | Zhang P, Zhang Z, Zhang LJ, et al. Glycosyltransferase GT1 family: Phylogenetic distribution, substrates coverage, and representative structural features[J]. Comput Struct Biotechnol J, 2020, 18: 1383-1390. |
| [9] | 陈锁英, 刘晓曼, 刘小刚, 等. 甜橙UDP-糖基转移酶基因的筛选及其功能分析[J]. 植物生理学报, 2021, 57(9): 1745-1754. |
| Chen SY, Liu XM, Liu XG, et al. Screening and functional analysis of UDP-glycosyltransferase genes in sweet orange(Citrus sinensis)[J]. Plant Physiol J, 2021, 57(9): 1745-1754. | |
| [10] | Yin QG, Shen GA, Di SK, et al. Genome-wide identification and functional characterization of UDP-glucosyltransferase genes involved in flavonoid biosynthesis in Glycine max[J]. Plant Cell Physiol, 2017, 58(9): 1558-1572. |
| [11] |
姚宇, 顾佳珺, 孙超, 等. 植物类黄酮UDP-糖基转移酶研究进展[J]. 生物技术通报, 2022, 38(12): 47-57.
doi: 10.13560/j.cnki.biotech.bull.1985.2022-0236 |
|
Yao Y, Gu JJ, Sun C, et al. Advances in plant flavonoids UDP-glycosyltransferase[J]. Biotechnol Bull, 2022, 38(12): 47-57.
doi: 10.13560/j.cnki.biotech.bull.1985.2022-0236 |
|
| [12] | Gharabli H, Della Gala V, Welner DH. The function of UDP-glycosyltransferases in plants and their possible use in crop protection[J]. Biotechnol Adv, 2023, 67: 108182. |
| [13] | Wu BP, Liu XH, Xu K, et al. Genome-wide characterization, evolution and expression profiling of UDP-glycosyltransferase family in pomelo(Citrus grandis)fruit[J]. BMC Plant Biol, 2020, 20(1): 459. |
| [14] | 赵倩倩, 宋艳红, 宋盼, 等. 森林草莓糖基转移酶基因家族生物信息学及其表达分析[J]. 南方农业学报, 2021, 52(6): 1615-1624. |
| Zhao QQ, Song YH, Song P, et al. Analysis of bioinformatics and gene expression of glycosyltransferase gene family in Fragaria vesca[J]. J South Agric, 2021, 52(6): 1615-1624. | |
| [15] | 祝小雅, 刘烨, 闫蕴韬, 等. 水稻UGT基因家族及成员SS4的生物信息学分析[J/OL]. 分子植物育种, 2023: 1-11. http://kns.cnki.net/kcms/detail/46.1068.S.20230505.1453.013.html. |
| Zhu XY, Liu Y, Yan YT, et al. Bioinformatics analysis of UGT gene family and its member SS4 in rice(Oryza sativa L.)[J/OL]. Molecular Plant Breeding, 2023: 1-11. http://kns.cnki.net/kcms/detail/46.1068.S.20230505.1453.013.html. | |
| [16] | 吕中睿, 刘宏, 张国昀, 等. 沙棘UGT基因家族的全基因组鉴定与表达分析[J]. 林业科学研究, 2021, 34(6): 9-19. |
| Lv ZR, Liu H, Zhang GY, et al. Genome-wide identification, characterization, and expression analysis of UGT gene family members in sea buckthorn(Hippophae rhamnoides L.)[J]. For Res, 2021, 34(6): 9-19. | |
| [17] |
Liu F, Yu HY, Deng YT, et al. PepperHub, an informatics hub for the chili pepper research community[J]. Mol Plant, 2017, 10(8): 1129-1132.
doi: S1674-2052(17)30076-X pmid: 28343897 |
| [18] |
Le Roy J, Huss B, Creach A, et al. Glycosylation is a major regulator of phenylpropanoid availability and biological activity in plants[J]. Front Plant Sci, 2016, 7: 735.
doi: 10.3389/fpls.2016.00735 pmid: 27303427 |
| [19] | 唐乙钧. 糖基异构酶OsUGE3调节水稻细胞壁多糖代谢和耐盐性的机制研究[D]. 沈阳: 沈阳农业大学, 2022. |
| Tang YJ. Mechanism of OsUGE3 regulating rice cell wall polysaccharides metabolism and salt tolerance[D]. Shenyang: Shenyang Agricultural University, 2022. | |
| [20] | 陈家栋, 姜武, 宋敏全, 等. 铁皮石斛尿苷二磷酸糖基转移酶(UGT)基因家族鉴定与表达分析[J]. 中国中药杂志, 2023, 48(7): 1840-1850. |
| Chen JD, Jiang W, Song MQ, et al. Identification and expression of uridine diphosphate glycosyltransferase(UGT)gene family from Dendrobium officinale[J]. China J Chin Mater Med, 2023, 48(7): 1840-1850. | |
| [21] | Montefiori M, Espley RV, Stevenson D, et al. Identification and characterisation of F3GT1 and F3GGT1, two glycosyltransferases responsible for anthocyanin biosynthesis in red-fleshed kiwifruit(Actinidia chinensis)[J]. Plant J, 2011, 65(1): 106-118. |
| [22] | 招雪晴, 杨静, 沈雨, 等. 石榴PgUGT基因表达特性与重组表达分析[J]. 西北植物学报, 2022, 42(3): 390-397. |
| Zhao XQ, Yang J, Shen Y, et al. Expression profiles and recombinant expression of PgUGT in pomegranate[J]. Acta Bot Boreali Occidentalia Sin, 2022, 42(3): 390-397. | |
| [23] | Filyushin MA, Shchennikova AV, Kochieva EZ. Antocyandin-3-O-glucosyltransferase genes in pepper(Capsicum spp.) and their role in anthocyanin biosynthesis[J]. Russ J Genet, 2023, 59(5): 441-452. |
| [24] | Zheng SW, Chen ZF, Liu TT, et al. Identification and characterization of the tomato UGT gene family and effects of GAME 17 overexpression on plants and growth and development under high-CO2 conditions[J]. Agronomy, 2022, 12(9): 1998. |
| [25] | 刘蒲东, 张舒婷, 申序, 等. 龙眼UGT家族全基因组鉴定、功能预测及表达模式[J]. 应用与环境生物学报, 2021, 27(6): 1626-1635. |
| Liu PD, Zhang ST, Shen X, et al. Genome-wide identification, function prediction, and expression pattern analysis of the UGT family in Dimocarpus longan Lour[J]. Chin J Appl Environ Biol, 2021, 27(6): 1626-1635. | |
| [26] | Caputi L, Malnoy M, Goremykin V, et al. A genome-wide phylogenetic reconstruction of family 1 UDP-glycosyltransferases revealed the expansion of the family during the adaptation of plants to life on land[J]. Plant J, 2012, 69(6): 1030-1042. |
| [27] | Zhao X, Feng Y, Ke D, et al. Molecular identification and characterization of UDP-glycosyltransferase(UGT)multigene family in Pomegranate[J]. Horticulturae, 2023, 9(5): 540-557. |
| [28] | Cui LL, Yao SB, Dai XL, et al. Identification of UDP-glycosyltransferases involved in the biosynthesis of astringent taste compounds in tea(Camellia sinensis)[J]. J Exp Bot, 2016, 67(8): 2285-2297. |
| [29] | Ren ZY, Ji XY, Jiao ZB, et al. Functional analysis of a novel C-glycosyltransferase in the orchid Dendrobium catenatum[J]. Hortic Res, 2020, 7: 111. |
| [30] | Rojas Rodas F, Rodriguez TO, Murai Y, et al. Linkage mapping, molecular cloning and functional analysis of soybean gene Fg2 encoding flavonol 3-O-glucoside(1→6)rhamnosyltransferase[J]. Plant Mol Biol, 2014, 84(3): 287-300. |
| [31] | Dong LL, Tang ZY, Yang TY, et al. Genome-wide analysis of UGT genes in Petunia and identification of PhUGT51 involved in the regulation of salt resistance[J]. Plants, 2022, 11(18): 2434. |
| [32] | Sun LQ, Zhao LJ, Huang H, et al. Genome-wide identification, evolution and function analysis of UGTs superfamily in cotton[J]. Front Mol Biosci, 2022, 9: 965403. |
| [33] | 许永华, 卫宝瑞, 佐月, 等. 低温影响药用植物次生代谢产物积累研究进展[J]. 分子植物育种, 2022, 20(5): 1708-1715. |
| Xu YH, Wei BR, Zuo Y, et al. Research progress on the effects of low temperature on the accumulation of secondary metabolites of medicinal plants[J]. Mol Plant Breed, 2022, 20(5): 1708-1715. | |
| [34] | Javot H, Penmetsa RV, Breuillin F, et al. Medicago truncatula mtpt4 mutants reveal a role for nitrogen in the regulation of arbuscule degeneration in arbuscular mycorrhizal symbiosis[J]. Plant J, 2011, 68(6): 954-965. |
| [35] | Zhang JX, He CM, Wu KL, et al. Transcriptome analysis of Dendrobium officinale and its application to the identification of genes associated with polysaccharide synthesis[J]. Front Plant Sci, 2016, 7: 5. |
| [36] | Zhang GQ, Xu Q, Bian C, et al. The Dendrobium catenatum Lindl. genome sequence provides insights into polysaccharide synthase, floral development and adaptive evolution[J]. Sci Rep, 2016, 6: 19029. |
| [1] | 申鹏, 高雅彬, 丁红. 马铃薯SAT基因家族的鉴定和表达分析[J]. 生物技术通报, 2024, 40(9): 64-73. |
| [2] | 宋兵芳, 柳宁, 程新艳, 徐晓斌, 田文茂, 高悦, 毕阳, 王毅. 马铃薯G6PDH基因家族鉴定及其在损伤块茎的表达分析[J]. 生物技术通报, 2024, 40(9): 104-112. |
| [3] | 满全财, 孟姿诺, 李伟, 蔡心汝, 苏润东, 付长青, 高顺娟, 崔江慧. 马铃薯AQP基因家族鉴定及表达分析[J]. 生物技术通报, 2024, 40(9): 51-63. |
| [4] | 李雨晴, 吴楠, 罗建让. 卵叶牡丹花色苷合成相关基因bHLH的克隆与功能分析[J]. 生物技术通报, 2024, 40(8): 174-185. |
| [5] | 周冉, 王兴平, 李彦霞, 罗仍卓么. 金黄色葡萄球菌型乳房炎奶牛乳腺组织的lncRNA差异表达分析[J]. 生物技术通报, 2024, 40(8): 320-328. |
| [6] | 崔原瑗, 王昭懿, 白双宇, 任毓昭, 豆飞飞, 刘彩霞, 刘凤楼, 王掌军, 李清峰. 大麦非特异性磷脂酶C基因家族全基因组鉴定及苗期胁迫表达分析[J]. 生物技术通报, 2024, 40(8): 74-82. |
| [7] | 杨巍, 赵丽芬, 唐兵, 周麟笔, 杨娟, 莫传园, 张宝会, 李飞, 阮松林, 邓英. 芥菜SRO基因家族全基因组鉴定与表达分析[J]. 生物技术通报, 2024, 40(8): 129-141. |
| [8] | 李亦君, 杨小贝, 夏琳, 罗朝鹏, 徐馨, 杨军, 宁黔冀, 武明珠. 烟草NtPRR37基因克隆及功能分析[J]. 生物技术通报, 2024, 40(8): 221-231. |
| [9] | 张明亚, 庞胜群, 刘玉东, 苏永峰, 牛博文, 韩琼琼. 番茄FAD基因家族的鉴定与表达分析[J]. 生物技术通报, 2024, 40(7): 150-162. |
| [10] | 臧文蕊, 马明, 砗根, 哈斯阿古拉. 甜瓜BZR转录因子家族基因的全基因组鉴定及表达模式分析[J]. 生物技术通报, 2024, 40(7): 163-171. |
| [11] | 王玉书, 赵琳琳, 赵爽, 胡琦, 白慧霞, 王欢, 曹业萍, 范震宇. 大白菜BrCYP83B1基因的克隆及表达分析[J]. 生物技术通报, 2024, 40(6): 152-160. |
| [12] | 常雪瑞, 王田田, 王静. 辣椒E2基因家族的鉴定及分析[J]. 生物技术通报, 2024, 40(6): 238-250. |
| [13] | 刘蓉, 田闵玉, 李光泽, 谭成方, 阮颖, 刘春林. 甘蓝型油菜REVEILLE家族鉴定及诱导表达分析[J]. 生物技术通报, 2024, 40(6): 161-171. |
| [14] | 李博静, 郑腊梅, 吴乌云, 高飞, 周宜君. 西蒙得木HSP20基因家族的进化、表达和功能分析[J]. 生物技术通报, 2024, 40(6): 190-202. |
| [15] | 郝思怡, 张君珂, 王斌, 曲朋燕, 李瑞得, 程春振. 香蕉ELF3的克隆与表达分析[J]. 生物技术通报, 2024, 40(5): 131-140. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||