







生物技术通报 ›› 2023, Vol. 39 ›› Issue (9): 236-245.doi: 10.13560/j.cnki.biotech.bull.1985.2023-0022
收稿日期:2023-01-11
出版日期:2023-09-26
发布日期:2023-10-24
作者简介:韩浩章,男,硕士,副教授,研究方向:樟属植物种质资源创新与应用;E-mail: 21011@squ.edu.cn;韩浩章同为本文通讯作者
基金资助:
HAN Hao-zhang( ), ZHANG Li-hua, LI Su-hua, ZHAO Rong, WANG Fang, WANG Xiao-li
), ZHANG Li-hua, LI Su-hua, ZHAO Rong, WANG Fang, WANG Xiao-li
Received:2023-01-11
Published:2023-09-26
Online:2023-10-24
摘要:
盐碱土壤环境是限制猴樟引种推广的主要因素,脯氨酸是植物适应盐碱胁迫过程中主要的渗透调节物质,筛选并鉴定出调控脯氨酸合成的转录因子对于猴樟耐盐碱分子机理研究具有重要意义。以猴樟实生苗为材料,在水培培养基础上,采用0 mmol/L和10 mmol/L的Na2CO3溶液分别进行处理,选取处理6 h、48 h的根系组织,提取总RNA,构建盐碱胁迫诱导的猴樟根系酵母cDNA文库;以猴樟根系总DNA为模板,克隆CbP5CS启动子序列,采用Y1H酵母单杂交技术筛选出与CbP5CS启动子存在互作的蛋白,并通过高通量测序技术鉴定出调控猴樟脯氨酸合成的转录因子。结果表明,所构建的cDNA文库库容为4.88×107 CFU/mL,总克隆数为9.76×107 CFU,文库插入片段平均长度在1 000 bp左右,cDNA片段重组率为100%,符合酵母杂交试验的要求。克隆出的CbP5CS启动子序列长2 012 bp,成功构建出诱饵质粒pHIS2-CbP5CS,利用共转化方法从文库中筛选到31个与CbP5CS互作的EST序列。经酵母回转验证,31个EST序列均与CbP5CS存在互作。经NGS测序和BLAST比对搜索,获得6个转录因子基因,分别为锌指CCCH结构域蛋白25异构体 x1、AtbHLH104、转录因子 TFIIIC、RING/FYVE/PHD锌指超家族蛋白、GATA型锌指转录因子家族蛋白、AtbHLH96。以上结果为进一步研究植物脯氨酸代谢响应盐碱胁迫的分子机制奠定基础。
韩浩章, 张丽华, 李素华, 赵荣, 王芳, 王晓立. 盐碱胁迫诱导的猴樟酵母cDNA文库构建及CbP5CS上游调控因子筛选[J]. 生物技术通报, 2023, 39(9): 236-245.
HAN Hao-zhang, ZHANG Li-hua, LI Su-hua, ZHAO Rong, WANG Fang, WANG Xiao-li. Construction of cDNA Library of Cinnamomun bodinieri Induced by Saline-alkali Stress and Screening of CbP5CS Upstream Regulators[J]. Biotechnology Bulletin, 2023, 39(9): 236-245.
| 引物名称Primer name | 序列Sequence(5'-3') | 应用Application |
|---|---|---|
| CDS III/3' PCR primer | AAGCAGTGGTATCAACGCAGAGTGGCCATTATGGCCGGG | 逆转录 |
| SMART IV oligonucleotide | ATTCTAGAGGCCGAGGCGGCCGACATGTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTVN | 逆转录 |
| P1-F | CATATGGCCATGGAGGCCAGTGAATTCAAGCAGTGGTATCAACGCAGAGTGG | ds cDNA扩增 |
| P2-F | CATATGGCCATGGAGGCCAGTGAATTCAAAGCAGTGGTATCAACGCAGAGTGG | ds cDNA扩增 |
| P3-F | CATATGGCCATGGAGGCCAGTGAATTCAAAAGCAGTGGTATCAACGCAGAGTGG | ds cDNA扩增 |
| P4-R | CATCTGCAGCTCGAGCTCGATGGATCCCTAGAGGCCGAGGCGGCCGACATG | ds cDNA扩增 |
| Prime M1 | AAGCAGTGGTATCAACGCAGAGT | 均一化 |
| T7-F | taatacgactcactatagggcgag | 菌落PCR |
| AD-R | GCACGATGCACAGTTGAAG | 菌落PCR |
表1 试验所用的引物
Table 1 Oligonucleotide primers used in this study
| 引物名称Primer name | 序列Sequence(5'-3') | 应用Application |
|---|---|---|
| CDS III/3' PCR primer | AAGCAGTGGTATCAACGCAGAGTGGCCATTATGGCCGGG | 逆转录 |
| SMART IV oligonucleotide | ATTCTAGAGGCCGAGGCGGCCGACATGTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTVN | 逆转录 |
| P1-F | CATATGGCCATGGAGGCCAGTGAATTCAAGCAGTGGTATCAACGCAGAGTGG | ds cDNA扩增 |
| P2-F | CATATGGCCATGGAGGCCAGTGAATTCAAAGCAGTGGTATCAACGCAGAGTGG | ds cDNA扩增 |
| P3-F | CATATGGCCATGGAGGCCAGTGAATTCAAAAGCAGTGGTATCAACGCAGAGTGG | ds cDNA扩增 |
| P4-R | CATCTGCAGCTCGAGCTCGATGGATCCCTAGAGGCCGAGGCGGCCGACATG | ds cDNA扩增 |
| Prime M1 | AAGCAGTGGTATCAACGCAGAGT | 均一化 |
| T7-F | taatacgactcactatagggcgag | 菌落PCR |
| AD-R | GCACGATGCACAGTTGAAG | 菌落PCR |

图2 ds cDNA琼脂糖电泳图 M:Maker;1:P1-F/P4-R扩增 ds cDNA;2:P2-F/P4-R扩增 ds cDNA;3:P3-F/P4-R扩增 ds cDNA
Fig. 2 Agarose gel electrophoresis of ds cDNA M: Marker. 1: ds cDNA amplified with P1-F/P4-R. 2: ds cDNA amplified with P2-F/P4-R. 3: ds cDNA amplified with P3-F/P4-R
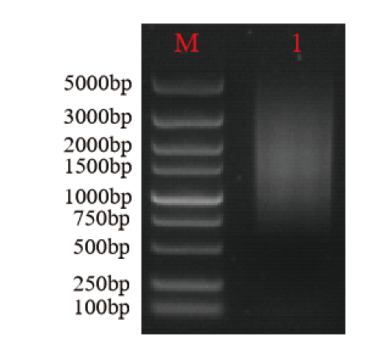
图3 琼脂糖凝胶电泳检测均一化与去小片段 M:Maker;1:3种ds cDNA混合后纯化结果
Fig. 3 Detection of normalization and removal of small fragments using agarose gel electrophoresis M: Marker. 1: Mixture of ds cDNA amplified with P1-F/P4-R, P2-F/P4-R, and P3-F/P4-R
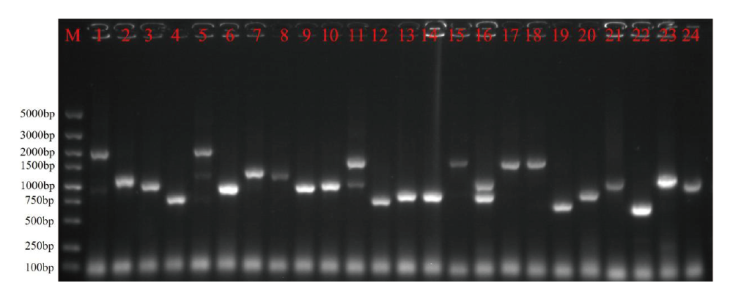
图4 文库质量鉴定电泳图 M:Maker;1-24:随机挑取的24个克隆进行PCR
Fig. 4 Library quality assessment using agarose gel electrophoresis M: Marker. 1-24: Colony PCR with 24 randomly selected E. coli colonies
| 位点 Site | 位置 Position | 序列 Sequence | 重复次数 Number of repetitions | 功能 Function |
|---|---|---|---|---|
| ABRE | -1 713 | ACGTG | 1 | 脱落酸响应元件 |
| AE-box | +191 | AGAAACAA | 1 | 部分光响应模块 |
| ARE | +680、-1 262、-693 | AAACCA | 3 | 厌氧诱导必需元件 |
| AT~TATA-box | +551 | TATATA | 1 | 光响应元件 |
| Box 4 | -1 846 | ATTAAT | 1 | 光响应中的保守DNA模块 |
| CAAT-box | +58、+75、-99、+139、+184、+196、+414、+513、+704、-743、-753、+770、+817、-819、+869、-902、+914、+941、+942、+1 005、-1 008、+1 115、 -1 118、1 140、-1 168、+1 195、+1 198、-1 275、 -1 293、+1 348、-1 351、-1 394、-1 429、+1 479、 +1 480、-1 498、+1 523、+1 540、+1 541、+1 570、+1 591、+1 617、-1 636、-1 813 | CAAAT/CAAT/CCAAT/TGCCAAC | 44 | 启动子和增强子区域普通顺式作用元件 |
| CAT-box | +1 976 | GCCACT | 1 | 分生组织表达响应元件 |
| CGTCA-motif | -143、-732、+146 | CGTCA | 3 | 茉莉酸甲酯响应的元件 |
| ERE | -1 743 | ATTTCATA | 1 | 乙烯响应元件 |
| G-Box | +1 713 | CACGTT | 1 | 光响应元件 |
| G-box | +80 | CACGAC | 1 | 光响应元件 |
| GT1-motif | -610、+1 225、+665 | GGTTAA/GTGTGTGAA | 3 | 光响应元件 |
| MYB | +2、-1 258、+1 201、-1 927、+1 001、-1 224 | CAACAG/CAACCA/TAACCA/ | 6 | MYB转录因子结合位点 |
| MYB-like sequence | -1 224、-1 927 | TAACCA | 2 | MYB转录因子结合位点 |
| MYC | -139、+817、-184 | CATTTG/CAATTG | 3 | bHLH转录因子结合位点 |
| Myb | -1 154、+1 485 | TAACTG | 2 | MYB转录因子结合位点 |
| Myb-binding site | +2、+1 201 | CAACAG | 2 | MYB转录因子结合位点 |
| P-box | -158 | CCTTTTG | 1 | 赤霉素响应元件 |
| SARE | -992 | TTCGACCATCTT | 1 | 水杨酸响应元件 |
| STRE | -265、+1 080、+1 801、+572、+1 962、-1 402、 -1 834、-281、-712 | AGGGG | 9 | 压力响应元件 |
| TATA-box | +202、-550、+267、+551、+552、+553、-1 161、 -1 162、+1 178、-1 179、-1 180、+1 471、-1 472、 -1 473、+1 554、-1 741、+1 850、-1 851、-1 917 | TATA/TATATAA/ccTATAAAaa/TATATA/ATATAA/ATATAT/ATTATA/TATAA/TACAAAA | 19 | -30位置附近核心启动子转录起始位点 |
| TATC-box | -258 | TATCCCA | 1 | 赤霉素响应元件 |
| TCA-element | -989、+1 408 | CCATCTTTTT | 2 | 水杨酸响应元件 |
| TCCC-motif | +1 870 | TCTCCCT | 1 | 光响应元件 |
| TGACG-motif | +143、+732、-146 | TGACG | 3 | 茉莉酸甲酯响应元件 |
| Unnamed | -234、+1 640、+256、-1 712 | CGTGG | 4 | 未知 |
| Unnamed | -628 | TCCACGTAGA | 1 | 未知 |
| Unnamed | -9、-1 083、+643、+1 871、-568、1 377、-833、 -1 191、-536、+1 863、+710、-635、-921 | CTCC | 13 | 未知 |
| WRE3 | -829、+1 865、-1 098 | CCACCT | 3 | 愈伤调节响应元件 |
| WUN-motif | -559 | AAATTACT | 1 | 愈伤调节响应元件 |
表2 猴樟CbP5CS基因启动子顺式调控元件
Table 2 cis-acting regulatory elements of the CbP5CS promoter in C. bodinieri
| 位点 Site | 位置 Position | 序列 Sequence | 重复次数 Number of repetitions | 功能 Function |
|---|---|---|---|---|
| ABRE | -1 713 | ACGTG | 1 | 脱落酸响应元件 |
| AE-box | +191 | AGAAACAA | 1 | 部分光响应模块 |
| ARE | +680、-1 262、-693 | AAACCA | 3 | 厌氧诱导必需元件 |
| AT~TATA-box | +551 | TATATA | 1 | 光响应元件 |
| Box 4 | -1 846 | ATTAAT | 1 | 光响应中的保守DNA模块 |
| CAAT-box | +58、+75、-99、+139、+184、+196、+414、+513、+704、-743、-753、+770、+817、-819、+869、-902、+914、+941、+942、+1 005、-1 008、+1 115、 -1 118、1 140、-1 168、+1 195、+1 198、-1 275、 -1 293、+1 348、-1 351、-1 394、-1 429、+1 479、 +1 480、-1 498、+1 523、+1 540、+1 541、+1 570、+1 591、+1 617、-1 636、-1 813 | CAAAT/CAAT/CCAAT/TGCCAAC | 44 | 启动子和增强子区域普通顺式作用元件 |
| CAT-box | +1 976 | GCCACT | 1 | 分生组织表达响应元件 |
| CGTCA-motif | -143、-732、+146 | CGTCA | 3 | 茉莉酸甲酯响应的元件 |
| ERE | -1 743 | ATTTCATA | 1 | 乙烯响应元件 |
| G-Box | +1 713 | CACGTT | 1 | 光响应元件 |
| G-box | +80 | CACGAC | 1 | 光响应元件 |
| GT1-motif | -610、+1 225、+665 | GGTTAA/GTGTGTGAA | 3 | 光响应元件 |
| MYB | +2、-1 258、+1 201、-1 927、+1 001、-1 224 | CAACAG/CAACCA/TAACCA/ | 6 | MYB转录因子结合位点 |
| MYB-like sequence | -1 224、-1 927 | TAACCA | 2 | MYB转录因子结合位点 |
| MYC | -139、+817、-184 | CATTTG/CAATTG | 3 | bHLH转录因子结合位点 |
| Myb | -1 154、+1 485 | TAACTG | 2 | MYB转录因子结合位点 |
| Myb-binding site | +2、+1 201 | CAACAG | 2 | MYB转录因子结合位点 |
| P-box | -158 | CCTTTTG | 1 | 赤霉素响应元件 |
| SARE | -992 | TTCGACCATCTT | 1 | 水杨酸响应元件 |
| STRE | -265、+1 080、+1 801、+572、+1 962、-1 402、 -1 834、-281、-712 | AGGGG | 9 | 压力响应元件 |
| TATA-box | +202、-550、+267、+551、+552、+553、-1 161、 -1 162、+1 178、-1 179、-1 180、+1 471、-1 472、 -1 473、+1 554、-1 741、+1 850、-1 851、-1 917 | TATA/TATATAA/ccTATAAAaa/TATATA/ATATAA/ATATAT/ATTATA/TATAA/TACAAAA | 19 | -30位置附近核心启动子转录起始位点 |
| TATC-box | -258 | TATCCCA | 1 | 赤霉素响应元件 |
| TCA-element | -989、+1 408 | CCATCTTTTT | 2 | 水杨酸响应元件 |
| TCCC-motif | +1 870 | TCTCCCT | 1 | 光响应元件 |
| TGACG-motif | +143、+732、-146 | TGACG | 3 | 茉莉酸甲酯响应元件 |
| Unnamed | -234、+1 640、+256、-1 712 | CGTGG | 4 | 未知 |
| Unnamed | -628 | TCCACGTAGA | 1 | 未知 |
| Unnamed | -9、-1 083、+643、+1 871、-568、1 377、-833、 -1 191、-536、+1 863、+710、-635、-921 | CTCC | 13 | 未知 |
| WRE3 | -829、+1 865、-1 098 | CCACCT | 3 | 愈伤调节响应元件 |
| WUN-motif | -559 | AAATTACT | 1 | 愈伤调节响应元件 |
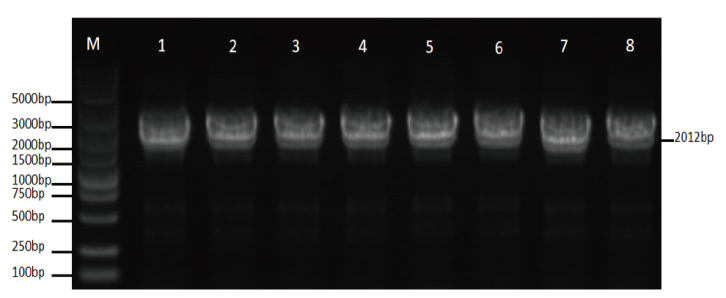
图5 诱饵质粒pHIS2-CbP5CS 菌落PCR鉴定 M:DNA marker; 1-8:随机挑取的8个pHIS2-CbP5CS菌落的扩增产物
Fig. 5 Colony PCR identification of decoy plasmid pHIS2-CbP5CS M: DNA marker. 1-8: Colony PCR with 8 randomly selected pHIS2-CbP5CS colonies

图6 自激活检测背景筛选和最小3-AT浓度 阳性对照为pHIS2-p53+pGAD53m
Fig. 6 Auto-activation detection background screening and minimal 3-AT concentration Positive control is pHIS2-p53+pGAD53m
| 基因ID Gene ID | 读数 Read count | 拟南芥同源序列 Arabidopsis thaliana homolog | E值E-value | 功能注释 Functional annotation |
|---|---|---|---|---|
| GW020491 | 1 | AT3G47120.1 | 8.00E-76 | 锌指 CCCH结构域蛋白25异构体 x1 |
| GW028183 | 15 | AT4G14410.2 | 6.00E-51 | AtbHLH104 |
| GW000650 | 3 | AT3G15420.1 | 1.00E-24 | 转录因子 TFIIIC |
| GW009089 | 7 | AT4G00755.2 | 6.00E-59 | F-box 家族蛋白 |
| GW009287 | 8 | AT1G64230.2 | 3.00E-83 | 泛素连接酶 |
| GW012884 | 24 | AT5G15730.2 | 2.00E-137 | 蛋白激酶超家族 |
| GW000705 | 24 | AT4G02340.1 | 2.00E-47 | α/β 水解酶超家族蛋白 |
| GW003120 | 4 | AT5G04480.1 | 0 | 葡萄糖基转移酶超家族蛋白 |
| GW004800 | 7 | AT5G63030.1 | 4.00E-42 | 硫氧还蛋白超家族蛋白 |
| GW006791 | 16 | ATMG01360.1 | 0 | 细胞色素氧化酶 |
| GW007525 | 13 | AT3G47550.6 | 6.00E-41 | RING/FYVE/PHD锌指超家族蛋白 |
| GW008185 | 15 | AT2G20360.1 | 1.00E-161 | NAD(P)结合罗斯曼卷曲超家族蛋白 |
| GW008711 | 19 | AT4G14305.1 | 8.00E-79 | 过氧化物酶体膜22 kD(Mpv17/PMP22)家族蛋白 |
| GW010514 | 3 | AT2G27030.3 | 2.00E-82 | 钙调素 |
| GW011043 | 24 | AT4G35450.1 | 3.00E-115 | 锚蛋白重复序列含有蛋白 |
| GW011868 | 16 | AT4G26220.1 | 3.00E-80 | S-腺苷甲硫氨酸依赖的甲基转移酶超家族蛋白 |
| GW011935 | 13 | AT1G55160.1 | 2.00E-35 | 功能未知的蛋白质 |
| GW012148 | 1 | AT1G54290.1 | 3.00E-48 | 翻译起始因子eIF1 |
| GW012186 | 19 | AT5G42850.2 | 4.00E-40 | 硫氧还蛋白超家族蛋白 |
| GW014171 | 56 | AT2G44860.2 | 2.00E-62 | 核糖体蛋白质 L24e 家族蛋白 |
| GW014289 | 34 | AT2G27020.1 | 1.00E-129 | 20s 蛋白酶体α亚基G1 |
| GW014358 | 24 | AT1G68590.1 | 1.00E-43 | 核糖体蛋白质 PSRP-3/Ycf65 |
| GW014549 | 10 | AT4G32140.1 | 4.00E-98 | 类EamA转运家族蛋白 |
| GW015686 | 1 | AT1G10200.1 | 2.00E-80 | GATA型锌指转录因子家族蛋白 |
| GW015796 | 64 | AT1G13950.1 | 4.00E-80 | 真核延伸因子5A-1 |
| GW017908 | 15 | AT3G55440.1 | 4.00E-111 | 磷酸丙糖异构酶 |
| GW020994 | 23 | AT5G53560.1 | 2.00E-48 | 细胞色素 B5亚型E |
| GW022429 | 11 | ATMG01360.1 | 0 | 细胞色素氧化酶 |
| GW024339 | 14 | AT4G19420.1 | 2.00E-153 | 果胶乙酰酯酶家族蛋白 |
| GW025798 | 12 | AT1G53680.1 | 1.00E-69 | 谷胱甘肽S-转移酶TAU28 |
| GW027120 | 11 | AT1G72210.1 | 4.00E-52 | AtbHLH96 |
表3 EST序列比对结果
Table 3 EST blast and annotation results
| 基因ID Gene ID | 读数 Read count | 拟南芥同源序列 Arabidopsis thaliana homolog | E值E-value | 功能注释 Functional annotation |
|---|---|---|---|---|
| GW020491 | 1 | AT3G47120.1 | 8.00E-76 | 锌指 CCCH结构域蛋白25异构体 x1 |
| GW028183 | 15 | AT4G14410.2 | 6.00E-51 | AtbHLH104 |
| GW000650 | 3 | AT3G15420.1 | 1.00E-24 | 转录因子 TFIIIC |
| GW009089 | 7 | AT4G00755.2 | 6.00E-59 | F-box 家族蛋白 |
| GW009287 | 8 | AT1G64230.2 | 3.00E-83 | 泛素连接酶 |
| GW012884 | 24 | AT5G15730.2 | 2.00E-137 | 蛋白激酶超家族 |
| GW000705 | 24 | AT4G02340.1 | 2.00E-47 | α/β 水解酶超家族蛋白 |
| GW003120 | 4 | AT5G04480.1 | 0 | 葡萄糖基转移酶超家族蛋白 |
| GW004800 | 7 | AT5G63030.1 | 4.00E-42 | 硫氧还蛋白超家族蛋白 |
| GW006791 | 16 | ATMG01360.1 | 0 | 细胞色素氧化酶 |
| GW007525 | 13 | AT3G47550.6 | 6.00E-41 | RING/FYVE/PHD锌指超家族蛋白 |
| GW008185 | 15 | AT2G20360.1 | 1.00E-161 | NAD(P)结合罗斯曼卷曲超家族蛋白 |
| GW008711 | 19 | AT4G14305.1 | 8.00E-79 | 过氧化物酶体膜22 kD(Mpv17/PMP22)家族蛋白 |
| GW010514 | 3 | AT2G27030.3 | 2.00E-82 | 钙调素 |
| GW011043 | 24 | AT4G35450.1 | 3.00E-115 | 锚蛋白重复序列含有蛋白 |
| GW011868 | 16 | AT4G26220.1 | 3.00E-80 | S-腺苷甲硫氨酸依赖的甲基转移酶超家族蛋白 |
| GW011935 | 13 | AT1G55160.1 | 2.00E-35 | 功能未知的蛋白质 |
| GW012148 | 1 | AT1G54290.1 | 3.00E-48 | 翻译起始因子eIF1 |
| GW012186 | 19 | AT5G42850.2 | 4.00E-40 | 硫氧还蛋白超家族蛋白 |
| GW014171 | 56 | AT2G44860.2 | 2.00E-62 | 核糖体蛋白质 L24e 家族蛋白 |
| GW014289 | 34 | AT2G27020.1 | 1.00E-129 | 20s 蛋白酶体α亚基G1 |
| GW014358 | 24 | AT1G68590.1 | 1.00E-43 | 核糖体蛋白质 PSRP-3/Ycf65 |
| GW014549 | 10 | AT4G32140.1 | 4.00E-98 | 类EamA转运家族蛋白 |
| GW015686 | 1 | AT1G10200.1 | 2.00E-80 | GATA型锌指转录因子家族蛋白 |
| GW015796 | 64 | AT1G13950.1 | 4.00E-80 | 真核延伸因子5A-1 |
| GW017908 | 15 | AT3G55440.1 | 4.00E-111 | 磷酸丙糖异构酶 |
| GW020994 | 23 | AT5G53560.1 | 2.00E-48 | 细胞色素 B5亚型E |
| GW022429 | 11 | ATMG01360.1 | 0 | 细胞色素氧化酶 |
| GW024339 | 14 | AT4G19420.1 | 2.00E-153 | 果胶乙酰酯酶家族蛋白 |
| GW025798 | 12 | AT1G53680.1 | 1.00E-69 | 谷胱甘肽S-转移酶TAU28 |
| GW027120 | 11 | AT1G72210.1 | 4.00E-52 | AtbHLH96 |
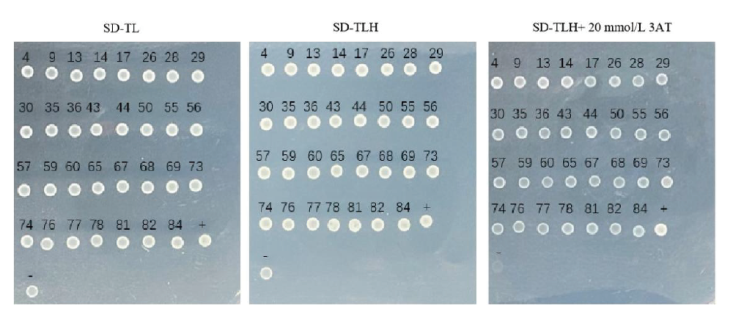
图9 酵母阳性克隆回转验证 “+”:阳性对照pGAD53m+ pHIS2-p53;“-”:阴性对pGAD53m+ pHIS2
Fig. 9 Rotation and confirmation of yeast positive colonies “+”: Positive control, pGAD53m+ pHIS2-p53; “-”: negative control, pGAD53m+ pHIS2
| [1] |
Xiao ZF, Jin ZN, Zhang BH, et al. Effects of IBA on rooting ability of Cinnamomum bodinieri citral type micro-shoots from transcriptomics analysis[J]. Plant Biotechnol Rep, 2020, 14(4): 467-477.
doi: 10.1007/s11816-020-00626-5 |
| [2] |
Guo HJ, Hu ZQ, Zhang HM, et al. Comparative effects of salt and alkali stress on antioxidant system in cotton(Gossypium hirsutum L.) leaves[J]. Open Chem, 2019, 17(1): 1352-1360.
doi: 10.1515/chem-2019-0147 URL |
| [3] |
Liu J, Wang YQ, Li QT. Analysis of differentially expressed genes and adaptive mechanisms of Prunus triloba Lindl. under alkaline stress[J]. Hereditas, 2017, 154: 10.
doi: 10.1186/s41065-017-0031-7 URL |
| [4] | Liu BS, Kang CL, Wang X, et al. Tolerance mechanisms of Leymus chinensis to salt-alkaline stress[J]. Acta Agric Scand Sect B Soil Plant Sci, 2015, 65(8): 723-734. |
| [5] |
Wang Y, Wang JC, Guo DD, et al. Physiological and comparative transcriptome analysis of leaf response and physiological adaption to saline alkali stress across pH values in alfalfa(Medicago sativa)[J]. Plant Physiol Biochem, 2021, 167: 140-152.
doi: 10.1016/j.plaphy.2021.07.040 URL |
| [6] |
Noreen S, Akhter MS, Yaamin T, et al. The ameliorative effects of exogenously applied proline on physiological and biochemical parameters of wheat(Triticum aestivum L.) crop under copper stress condition[J]. J Plant Interact, 2018, 13(1): 221-230.
doi: 10.1080/17429145.2018.1437480 URL |
| [7] |
Iqbal N. Photosynthetic differences in mustard genotypes under salinity stress: significance of proline metabolism[J]. Annu Res Rev Biol, 2014, 4: 3274-3296.
doi: 10.9734/ARRB URL |
| [8] |
Funck D, Baumgarten L, Stift M, et al. Differential contribution of P5CS isoforms to stress tolerance in Arabidopsis[J]. Front Plant Sci, 2020, 11: 565134.
doi: 10.3389/fpls.2020.565134 URL |
| [9] |
He L, Shi XX, Wang YM, et al. Arabidopsis ANAC069 binds to C[A/G]CG[T/G]sequences to negatively regulate salt and osmotic stress tolerance[J]. Plant Mol Biol, 2017, 93(4): 369-387.
doi: 10.1007/s11103-016-0567-3 URL |
| [10] |
Verma D, Jalmi SK, Bhagat PK, et al. A bHLH transcription factor, MYC2, imparts salt intolerance by regulating proline biosynthesis in Arabidopsis[J]. FEBS J, 2020, 287(12): 2560-2576.
doi: 10.1111/febs.v287.12 URL |
| [11] |
Jin C, Li KQ, Xu XY, et al. A novel NAC transcription factor, PbeNAC1, of Pyrus betulifolia confers cold and drought tolerance via interacting with PbeDREBs and activating the expression of stress-responsive genes[J]. Front Plant Sci, 2017, 8: 1049.
doi: 10.3389/fpls.2017.01049 URL |
| [12] |
Dai WS, Wang M, Gong XQ, et al. The transcription factor FcWRKY40 of Fortunella crassifolia functions positively in salt tolerance through modulation of ion homeostasis and proline biosynthesis by directly regulating SOS2 and P5CS1 homologs[J]. New Phytol, 2018, 219(3): 972-989.
doi: 10.1111/nph.2018.219.issue-3 URL |
| [13] | Liang JH, Zheng J, Wu Z, et al. Strawberry FaNAC2 enhances tolerance to abiotic stress by regulating proline metabolism[J]. Plants(Basel), 2020, 9(11): 1417. |
| [14] |
Zhang WH, Yang GY, Mu D, et al. An ethylene-responsive factor BpERF11 negatively modulates salt and osmotic tolerance in Betula platyphylla[J]. Sci Rep, 2016, 6: 23085.
doi: 10.1038/srep23085 |
| [15] |
Qin LP, Wang LQ, Guo Y, et al. An ERF transcription factor from Tamarix hispida, ThCRF1, can adjust osmotic potential and reactive oxygen species scavenging capability to improve salt tolerance[J]. Plant Sci, 2017, 265: 154-166.
doi: 10.1016/j.plantsci.2017.10.006 URL |
| [16] |
Zhang H, Liu XL, Zhang RX, et al. Root damage under alkaline stress is associated with reactive oxygen species accumulation in rice(Oryza sativa L.)[J]. Front Plant Sci, 2017, 8: 1580.
doi: 10.3389/fpls.2017.01580 pmid: 28943882 |
| [17] | Legocka J, Sobieszczuk-Nowicka E, Ludwicki D, et al. Putrescine catabolism via DAO contributes to proline and GABA accumulation in roots of lupine seedlings growing under salt stress[J]. Acta Soc Bot Pol, 2017, 86(3): 3549. |
| [18] |
Liu LJ, Huang L, Lin XY, et al. Hydrogen peroxide alleviates salinity-induced damage through enhancing proline accumulation in wheat seedlings[J]. Plant Cell Rep, 2020, 39(5): 567-575.
doi: 10.1007/s00299-020-02513-3 pmid: 32025801 |
| [19] |
Ghosh UK, Islam MN, Siddiqui MN, et al. Proline, a multifaceted signaling molecule in plant responses to abiotic stress: understanding the physiological mechanisms[J]. Plant Biol J, 2022, 24(2): 227-239.
doi: 10.1111/plb.v24.2 URL |
| [20] |
Han GL, Wang MJ, Yuan F, et al. The CCCH zinc finger protein gene AtZFP1 improves salt resistance in Arabidopsis thaliana[J]. Plant Mol Biol, 2014, 86(3): 237-253.
doi: 10.1007/s11103-014-0226-5 URL |
| [21] |
Huang J, Sun SJ, Xu DQ, et al. A TFIIIA-type zinc finger protein confers multiple abiotic stress tolerances in transgenic rice(Oryza sativa L.)[J]. Plant Mol Biol, 2012, 80(3): 337-350.
doi: 10.1007/s11103-012-9955-5 pmid: 22930448 |
| [22] |
Yang TR, Yao SF, Hao L, et al. Wheat bHLH-type transcription factor gene TabHLH1 is crucial in mediating osmotic stresses tolerance through modulating largely the ABA-associated pathway[J]. Plant Cell Rep, 2016, 35(11): 2309-2323.
doi: 10.1007/s00299-016-2036-5 URL |
| [23] |
Guo MX, Li SP, Tian S, et al. Transcriptome analysis of genes involved in defense against alkaline stress in roots of wild jujube(Ziziphus acidojujuba)[J]. PLoS One, 2017, 12(10): e0185732.
doi: 10.1371/journal.pone.0185732 URL |
| [24] |
Qian YC, Zhang TY, Yu Y, et al. Regulatory mechanisms of bHLH transcription factors in plant adaptive responses to various abiotic stresses[J]. Front Plant Sci, 2021, 12: 677611.
doi: 10.3389/fpls.2021.677611 URL |
| [25] |
Wang FB, Zhu H, Kong WL, et al. The Antirrhinum AmDEL gene enhances flavonoids accumulation and salt and drought tolerance in transgenic Arabidopsis[J]. Planta, 2016, 244(1): 59-73.
doi: 10.1007/s00425-016-2489-3 URL |
| [26] |
Zhang HF, Guo JB, Chen XQ, et al. Pepper bHLH transcription factor CabHLH035 contributes to salt tolerance by modulating ion homeostasis and proline biosynthesis[J]. Hortic Res, 2022, 9: uhac203.
doi: 10.1093/hr/uhac203 URL |
| [1] | 黄小龙, 孙贵连, 马丹丹, 闫慧清. 水稻幼苗酵母单杂文库构建及LAZY1上游调控因子筛选[J]. 生物技术通报, 2023, 39(9): 126-135. |
| [2] | 吕秋谕, 孙培媛, 冉彬, 王佳蕊, 陈庆富, 李洪有. 苦荞转录因子基因FtbHLH3的克隆、亚细胞定位及表达分析[J]. 生物技术通报, 2023, 39(8): 194-203. |
| [3] | 徐靖, 朱红林, 林延慧, 唐力琼, 唐清杰, 王效宁. 甘薯IbHQT1启动子的克隆及上游调控因子的鉴定[J]. 生物技术通报, 2023, 39(8): 213-219. |
| [4] | 李博, 刘合霞, 陈宇玲, 周兴文, 朱宇林. 金花茶CnbHLH79转录因子的克隆、亚细胞定位及表达分析[J]. 生物技术通报, 2023, 39(8): 241-250. |
| [5] | 陈晓, 于茗兰, 吴隆坤, 郑晓明, 逄洪波. 植物lncRNA及其对低温胁迫响应的研究进展[J]. 生物技术通报, 2023, 39(7): 1-12. |
| [6] | 孔德真, 段震宇, 王刚, 张鑫, 席琳乔. 盐、碱胁迫下高丹草苗期生理特征及转录组学分析[J]. 生物技术通报, 2023, 39(6): 199-207. |
| [7] | 郭怡婷, 赵文菊, 任延靖, 赵孟良. 菊芋NAC转录因子家族基因的鉴定及分析[J]. 生物技术通报, 2023, 39(6): 217-232. |
| [8] | 冯珊珊, 王璐, 周益, 王幼平, 方玉洁. WOX家族基因调控植物生长发育和非生物胁迫响应的研究进展[J]. 生物技术通报, 2023, 39(5): 1-13. |
| [9] | 王兵, 赵会纳, 余婧, 余世洲, 雷波. 植物侧枝发育的调控研究进展[J]. 生物技术通报, 2023, 39(5): 14-22. |
| [10] | 张新博, 崔浩亮, 史佩华, 高锦春, 赵顺然, 陶晨雨. 低起始量的免疫共沉淀技术研究进展[J]. 生物技术通报, 2023, 39(4): 227-235. |
| [11] | 葛颜锐, 赵冉, 徐静, 李若凡, 胡云涛, 李瑞丽. 植物维管形成层发育及其调控的研究进展[J]. 生物技术通报, 2023, 39(3): 13-25. |
| [12] | 刘铖霞, 孙宗艳, 罗云波, 朱鸿亮, 曲桂芹. bHLH转录因子的磷酸化调控植物生理功能的研究进展[J]. 生物技术通报, 2023, 39(3): 26-34. |
| [13] | 赵孟良, 郭怡婷, 任延靖. 菊芋WRKY转录因子家族基因的鉴定及分析[J]. 生物技术通报, 2023, 39(2): 116-125. |
| [14] | 韩芳英, 胡昕, 王楠楠, 谢裕红, 王晓艳, 朱强. DREBs响应植物非生物逆境胁迫研究进展[J]. 生物技术通报, 2023, 39(11): 86-98. |
| [15] | 陈楚怡, 杨小梅, 陈胜艳, 陈斌, 岳莉然. ABA和干旱胁迫下菊花脑ZF-HD基因家族的表达分析[J]. 生物技术通报, 2023, 39(11): 270-282. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||